En genética , un gen regulador , regulador o gen regulador es un gen implicado en el control de la expresión de uno o más genes distintos. Las secuencias reguladoras , que codifican genes reguladores, suelen estar en el extremo cinco primario (5') del sitio de inicio de la transcripción del gen que regulan. Además, estas secuencias también se pueden encontrar en el extremo tres primario (3') del sitio de inicio de la transcripción. En ambos casos, ya sea que la secuencia reguladora ocurra antes (5') o después (3') del gen que regula, la secuencia suele estar a muchas kilobases de distancia del sitio de inicio de la transcripción . Un gen regulador puede codificar una proteína o puede funcionar a nivel de ARN , como en el caso de los genes que codifican microARN . Un ejemplo de gen regulador es un gen que codifica una proteína represora que inhibe la actividad de un operador (un gen que se une a proteínas represoras inhibiendo así la traducción de ARN en proteína a través de la ARN polimerasa ). [1]
En los procariotas , los genes reguladores a menudo codifican proteínas represoras . Las proteínas represoras se unen a operadores o promotores , impidiendo que la ARN polimerasa transcriba el ARN. Por lo general, se expresan constantemente, por lo que la célula siempre tiene a mano un suministro de moléculas represoras. [2] Los inductores hacen que las proteínas represoras cambien de forma o se vuelvan incapaces de unirse al ADN, lo que permite que la ARN polimerasa continúe la transcripción. Los genes reguladores pueden ubicarse dentro de un operón , adyacentes a él o alejados de él. [3]
Otros genes reguladores codifican proteínas activadoras . Un activador se une a un sitio de la molécula de ADN y provoca un aumento en la transcripción de un gen cercano. En procariotas, una proteína activadora muy conocida es la proteína activadora de catabolitos (CAP), implicada en el control positivo del operón lac .
En la regulación de la expresión génica , estudiada en biología evolutiva del desarrollo (evo-devo), tanto activadores como represores juegan papeles importantes. [4]
Los genes reguladores también pueden describirse como reguladores positivos o negativos, según las condiciones ambientales que rodean a la célula. Los reguladores positivos son elementos reguladores que permiten que la ARN polimerasa se una a la región promotora, permitiendo así que se produzca la transcripción. En términos del operón lac, el regulador positivo sería el complejo CRP-AMPc que debe unirse cerca del sitio de inicio de la transcripción de los genes lac. La unión de este regulador positivo permite que la ARN polimerasa se una con éxito al promotor de la secuencia del gen lac, lo que hace avanzar la transcripción de los genes lac ; lac Z, lac Y y lac A. Los reguladores negativos son elementos reguladores que obstruyen la unión de la ARN polimerasa a la región promotora, reprimiendo así la transcripción. En términos del operón lac, el regulador negativo sería el represor lac que se une al promotor en el mismo sitio al que normalmente se une la ARN polimerasa. La unión del represor lac al sitio de unión de la ARN polimerasa inhibe la transcripción de los genes lac. Sólo cuando un inductor se une al represor lac el sitio de unión quedará libre para que la ARN polimerasa lleve a cabo la transcripción de los genes lac. [5] [6] [7]
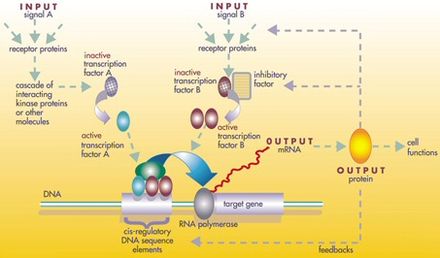
Los promotores residen al comienzo del gen y sirven como sitio donde se ensambla la maquinaria de transcripción y comienza la transcripción del gen. Los potenciadores activan a los promotores en lugares, momentos y niveles específicos y pueden definirse simplemente como los "promotores del promotor". Se cree que los silenciadores desactivan la expresión genética en momentos y lugares específicos. Los aislantes, también llamados elementos límite, son secuencias de ADN que crean límites reguladores en cis que evitan que los elementos reguladores de un gen afecten a los genes vecinos. El dogma general es que estos elementos reguladores se activan mediante la unión de factores de transcripción , proteínas que se unen a secuencias específicas de ADN y controlan la transcripción del ARNm . Podría haber varios factores de transcripción que deban unirse a un elemento regulador para activarlo. Además, varias otras proteínas, llamadas cofactores de transcripción, se unen a los propios factores de transcripción para controlar la transcripción. [8] [9]
Los reguladores negativos actúan para impedir la transcripción o traducción. Ejemplos como cFLIP suprimen los mecanismos de muerte celular que conducen a trastornos patológicos como el cáncer y, por tanto, desempeñan un papel crucial en la resistencia a los medicamentos . Evitar a estos actores es un desafío en la terapia contra el cáncer . [10] Los reguladores negativos de la muerte celular en el cáncer incluyen cFLIP , familia Bcl 2 , Survivin , HSP , IAP , NF-κB , Akt , mTOR y FADD . [10]
Existen varias técnicas diferentes para detectar genes reguladores, pero de muchas hay unas pocas que se utilizan con más frecuencia que otras. Uno de estos pocos elegidos se llama ChIP-chip. ChIP-chip es una técnica in vivo utilizada para determinar los sitios de unión genómica para factores de transcripción en reguladores de respuesta del sistema de dos componentes. El ensayo basado en microarrays in vitro (chip DAP) se puede utilizar para determinar objetivos genéticos y funciones de sistemas de transducción de señales de dos componentes . Este ensayo aprovecha el hecho de que los reguladores de respuesta pueden fosforilarse y así activarse in vitro utilizando donantes de moléculas pequeñas como el acetilfosfato . [11] [12]
La huella filogenética es una técnica que utiliza múltiples alineamientos de secuencias para determinar las ubicaciones de secuencias conservadas , como los elementos reguladores. Junto con múltiples alineamientos de secuencias, la huella filogenética también requiere tasas estadísticas de secuencias conservadas y no conservadas. Utilizando la información proporcionada por múltiples alineamientos de secuencias y tasas estadísticas, se pueden identificar los motivos mejor conservados en las regiones ortólogas de interés. [13] [14]
{{cite book}}: |journal=ignorado ( ayuda )