La familia de proteínas Argonaute , descubierta por primera vez por su función de célula madre conservada evolutivamente, [1] desempeña un papel central en los procesos de silenciamiento de ARN como componentes esenciales del complejo de silenciamiento inducido por ARN (RISC). RISC es responsable del fenómeno de silenciamiento génico conocido como interferencia de ARN (RNAi) . [2] Las proteínas Argonaute se unen a diferentes clases de ARN pequeños no codificantes , incluidos microARN (miARN), ARN pequeños interferentes (siRNA) y ARN que interactúan con Piwi (piRNA). Los ARN pequeños guían a las proteínas Argonaute a sus objetivos específicos a través de la complementariedad de secuencias (emparejamiento de bases), que luego conduce a la escisión del ARNm, la inhibición de la traducción y/o el inicio de la descomposición del ARNm. [3]
El nombre de esta familia de proteínas se deriva de un fenotipo mutante resultante de la mutación de AGO1 en Arabidopsis thaliana , que fue comparado por Bohmert et al. con la apariencia del pulpo pelágico Argonauta argo . [4]

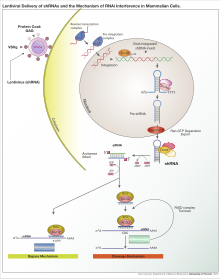
La interferencia de ARN (RNAi) es un proceso biológico en el que las moléculas de ARN inhiben la expresión génica , ya sea mediante la destrucción de moléculas de ARNm específicas o suprimiendo la traducción. [5] La interferencia de ARN tiene un papel importante en la defensa de las células contra secuencias de nucleótidos parásitas [ cita requerida ] . En eucariotas, incluidos los animales, la interferencia de ARN es iniciada por la enzima Dicer . Dicer escinde moléculas largas de ARN bicatenario (dsRNA, que a menudo se encuentran en virus y ARN interferente pequeño ) en fragmentos bicatenarios cortos de alrededor de 20 nucleótidos de ARNi. Luego, el dsRNA se separa en dos ARN monocatenarios (ssRNA): la cadena pasajera y la cadena guía. Posteriormente, la cadena pasajera se degrada, mientras que la cadena guía se incorpora al complejo de silenciamiento inducido por ARN (RISC). El resultado más estudiado del ARNi es el silenciamiento génico postranscripcional, que ocurre cuando la cadena guía se empareja con una secuencia complementaria en una molécula de ARN mensajero e induce la escisión por Argonaute, que se encuentra en el núcleo del complejo de silenciamiento inducido por ARN.
Las proteínas Argonauta son la parte activa del complejo de silenciamiento inducido por ARN, que escinde la cadena de ARNm diana complementaria a su ARNi unido. [6] En teoría, el dicer produce fragmentos cortos de doble cadena, por lo que también debería haber dos ARNi monocatenarios funcionales producidos. Pero solo uno de los dos ARN monocatenarios aquí se utilizará para aparearse con el ARNm diana . Se conoce como la cadena guía, que se incorpora a la proteína Argonauta y conduce al silenciamiento del gen. El otro ARN monocatenario, llamado cadena pasajera, se degrada durante el proceso del complejo de silenciamiento inducido por ARN. [7]
Una vez que el Argonauta se asocia con el ARN pequeño, la actividad enzimática conferida por el dominio PIWI escinde solo la cadena pasajera del ARN interferente pequeño. La separación de la cadena de ARN y la incorporación a la proteína Argonauta están guiadas por la fuerza de la interacción del enlace de hidrógeno en los extremos 5' del dúplex de ARN, conocida como regla de asimetría. Además, el grado de complementariedad entre las dos cadenas del dúplex de ARN intermedio define cómo se clasifican los miRNA en diferentes tipos de proteínas Argonauta.
En los animales, el Argonaute asociado con el miRNA se une a la región 3′ no traducida del ARNm y previene la producción de proteínas de varias maneras. El reclutamiento de proteínas Argonaute al ARNm objetivo puede inducir la degradación del ARNm. El complejo Argonaute-miRNA también puede afectar la formación de ribosomas funcionales en el extremo 5′ del ARNm. El complejo aquí compite con los factores de iniciación de la traducción y/o anula el ensamblaje de ribosomas . Además, el complejo Argonaute-miRNA puede ajustar la producción de proteínas al reclutar factores celulares como péptidos o enzimas modificadoras postraduccionales, que degradan el crecimiento de polipéptidos. [8]
En las plantas, una vez que se generan dúplex de ARN bicatenario (ds) de novo con el ARNm objetivo, una enzima desconocida similar a la ARNasa III produce nuevos ARNi, que luego se cargan en las proteínas Argonauta que contienen dominios PIWI, que carecen de los residuos de aminoácidos catalíticos , lo que podría inducir otro nivel de silenciamiento génico específico.
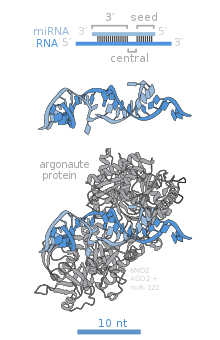
La familia de genes Argonaute (AGO) codifica seis dominios característicos: N-terminal (N), Linker-1 (L1), PAZ, Linker-2 (L2), Mid y un dominio PIWI C-terminal. [8]
El dominio PAZ recibe su nombre de Drosophila Piwi, Arabidopsis Argonaute-1 y Arabidopsis Zwille (también conocida como pinhead y posteriormente rebautizada como argonaute-10), donde se reconoció por primera vez que el dominio estaba conservado. El dominio PAZ es un módulo de unión de ARN que reconoce los extremos 3′ monocatenarios de siRNA , miRNA y piRNA , de manera independiente de la secuencia.
PIWI recibe su nombre de la proteína Piwi de Drosophila. El dominio PIWI, que se parece estructuralmente a la ARNasa H, es esencial para la escisión del objetivo. El sitio activo con la tríada aspartato-aspartato-glutamato alberga un ion metálico divalente, necesario para la catálisis. Los miembros de la familia de AGO que perdieron esta característica conservada durante la evolución carecen de la actividad de escisión. En la AGO humana, el motivo PIWI también media la interacción proteína-proteína en la caja PIWI, donde se une a Dicer en un dominio de la ARNasa III. [9]
En la interfaz de los dominios PIWI y Mid se encuentra el fosfato 5' de un ARNi, miARN o piARN, que se considera esencial para la funcionalidad. Dentro de Mid se encuentra un motivo MC, una estructura homóloga que se propuso que imita el motivo de la estructura de unión a la tapa que se encuentra en eIF4E. Más tarde se descubrió que el motivo MC no está involucrado en la unión a la tapa del ARNm [8].
En los seres humanos, hay ocho miembros de la familia AGO, algunos de los cuales se investigan intensamente. Sin embargo, aunque AGO1–4 son capaces de cargar miRNA, la actividad endonucleasa y, por lo tanto, el silenciamiento génico dependiente de RNAi pertenece exclusivamente a AGO2. Considerando la conservación de la secuencia de los dominios PAZ y PIWI en toda la familia, se presume que la singularidad de AGO2 surge del extremo N o de la región espaciadora que une los motivos PAZ y PIWI. [9]
Varios miembros de la familia AGO en plantas también atraen el estudio. AGO1 está involucrado en la degradación de ARN relacionada con miRNA y desempeña un papel central en la morfogénesis. En algunos organismos, es estrictamente necesario para el silenciamiento epigenético. Está regulado por el propio miRNA. AGO4 no participa en la degradación de ARN dirigida por RNAi, sino en la metilación de ADN y otra regulación epigenética, a través de la vía de ARN pequeño (smRNA). AGO10 está involucrado en el desarrollo de las plantas. AGO7 tiene una función distinta de AGO 1 y 10, y no se encuentra en el silenciamiento génico inducido por transgenes. En cambio, está relacionado con el momento del desarrollo en las plantas. [10]
Se ha informado que las proteínas argonautas están asociadas con cánceres. [11] [12] Para las enfermedades que están involucradas con la expresión selectiva o elevada de genes identificados particulares, como el cáncer de páncreas, la alta especificidad de secuencia de la interferencia de ARN podría hacer que sea adecuada para ser un tratamiento adecuado, particularmente apropiado para combatir cánceres asociados con secuencias de genes endógenos mutados . Se ha informado que varios ARN no codificantes diminutos (microARN) están relacionados con cánceres humanos, como miR-15a y miR-16a que se eliminan y/o regulan a la baja con frecuencia en pacientes. Aunque las funciones biológicas de los miARN no se comprenden completamente, se han descubierto los roles de los miARN en la coordinación de la proliferación celular y la muerte celular durante el desarrollo y el metabolismo. Se confía en que los miARN pueden dirigir la regulación negativa o positiva a diferentes niveles, lo que depende de los miARN específicos y la interacción del par de bases objetivo y los cofactores que los reconocen. [13]
Debido a que se sabe ampliamente que muchos virus tienen ARN en lugar de ADN como material genético y pasan por al menos una etapa de su ciclo de vida en la que producen ARN de doble cadena, se ha considerado que la interferencia de ARN es un mecanismo potencialmente evolutivamente antiguo para proteger a los organismos de los virus. Los pequeños ARN interferentes producidos por Dicer causan silenciamiento génico postranscripcional específico de secuencia al guiar una endonucleasa, el complejo de silenciamiento inducido por ARN (RISC), al ARNm. Este proceso se ha observado en una amplia gama de organismos, como el hongo Neurospora (en el que se conoce como quelling), las plantas (silenciamiento génico postranscripcional) y las células de mamíferos (RNAi). Si hay una complementariedad de secuencia completa o casi completa entre el ARN pequeño y el objetivo, el componente proteico Argonaute de RISC media la escisión de la transcripción objetivo, el mecanismo implica la represión de la traducción predominantemente [ cita requerida ] .
En 2016, un grupo de la Universidad de Ciencia y Tecnología de Hebei informó sobre la edición del genoma utilizando una proteína Argonauta procariota de Natronobacterium gregoryi . Sin embargo, la evidencia de la aplicación de las proteínas Argonauta como nucleasas guiadas por ADN para la edición del genoma ha sido cuestionada, con la retractación de la afirmación de la revista líder. [14] En 2017, un grupo de la Universidad de Illinois informó sobre el uso de una proteína Argonauta procariota tomada de Pyrococcus furiosus (PfAgo) junto con ADN guía para editar ADN in vitro como enzimas de restricción artificiales . [15] Las enzimas de restricción artificiales basadas en PfAgo también se utilizaron para almacenar datos sobre secuencias de ADN nativas a través de mellas enzimáticas. [16]