La poli (ADP-ribosa) polimerasa ( PARP ) es una familia de proteínas implicadas en una serie de procesos celulares como la reparación del ADN , la estabilidad genómica y la muerte celular programada . [1]
La familia PARP está compuesta por 17 miembros (10 putativos). [2] Varían mucho en estructura y función dentro de la célula.
PARP se compone de cuatro dominios de interés: un dominio de unión al ADN , un dominio escindido por caspasa (ver más abajo), un dominio de automodificación y un dominio catalítico . El dominio de unión al ADN está compuesto por dos motivos de dedos de zinc . En presencia de ADN dañado (pares de bases extirpados), el dominio de unión al ADN se unirá al ADN e inducirá un cambio conformacional . Se ha demostrado que esta unión se produce independientemente de los otros dominios. Esto es parte integral de un modelo de muerte celular programada basado en la inhibición de la escisión de caspasa de PARP. El dominio de automodificación es responsable de liberar la proteína del ADN después de la catálisis. Además, juega un papel integral en la inactivación inducida por escisión.
La función principal de PARP (que se encuentra en el núcleo celular ) es detectar e iniciar una respuesta celular inmediata a roturas de ADN monocatenario (SSB) metabólicas, químicas o inducidas por radiación mediante la señalización de la maquinaria enzimática involucrada en la reparación de SSB .
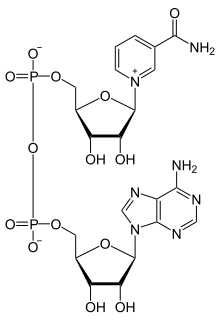
Una vez que PARP detecta una SSB, se une al ADN , sufre un cambio estructural y comienza la síntesis de una cadena polimérica de adenosina difosfato ribosa (poli (ADP-ribosa) o PAR), que actúa como señal para las otras cadenas de reparación del ADN. enzimas. Las enzimas objetivo incluyen la ADN ligasa III (LigIII), la ADN polimerasa beta (polβ) y proteínas de andamiaje como el gen 1 de complementación cruzada de rayos X (XRCC1). Después de la reparación, las cadenas PAR se degradan mediante la poli(ADP-ribosa) glicohidrolasa (PARG). [3]
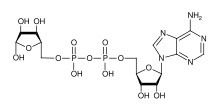
Se requiere NAD+ como sustrato para generar monómeros de ADP-ribosa. Se ha pensado que la sobreactivación de PARP puede agotar las reservas de NAD+ celular e inducir un agotamiento progresivo de ATP y muerte celular necrótica, ya que se inhibe la oxidación de la glucosa . [4] Pero más recientemente se sugirió que la inhibición de la actividad de la hexoquinasa conduce a defectos en la glucólisis (Andrabi, PNAS 2014). La actividad basal de PARP también regula la bioenergética basal. [5] Tenga en cuenta a continuación que PARP se inactiva mediante la escisión de caspasa-3 durante la muerte celular programada .
Las enzimas PARP son esenciales en una serie de funciones celulares, [6] incluida la expresión de genes inflamatorios: [7] PARP1 es necesaria para la inducción de la expresión del gen ICAM-1 por los miocitos cardíacos [8] y las células del músculo liso, en respuesta al TNF . [9]
El dominio catalítico es responsable de la polimerización de la poli (ADP-ribosa) . Este dominio tiene un motivo altamente conservado que es común a todos los miembros de la familia PARP. El polímero PAR puede alcanzar longitudes de hasta 200 nucleótidos antes de inducir procesos apoptóticos. La formación de polímero PAR es similar a la formación de polímero de ADN a partir de nucleósidos trifosfato. La síntesis normal de ADN requiere que un pirofosfato actúe como grupo saliente, dejando un solo grupo fosfato que une los azúcares desoxirribosa . PAR se sintetiza utilizando nicotinamida (NAM) como grupo saliente. Esto deja un pirofosfato como grupo de enlace entre los azúcares ribosa en lugar de grupos fosfato individuales. Esto crea un volumen especial para un puente PAR, que puede tener una función adicional en la señalización celular.
Una función importante de PARP es ayudar en la reparación de mellas de ADN monocatenario . Se une a sitios con roturas monocatenarias a través de sus dedos de zinc N-terminales y reclutará XRCC1 , ADN ligasa III, ADN polimerasa beta y una quinasa en la mella. Esto se llama reparación por escisión de base (BER). Se ha demostrado que PARP-2 se oligomeriza con PARP-1 y, por lo tanto, también está implicado en BER. También se ha demostrado que la oligomerización estimula la actividad catalítica de PARP. PARP-1 también es conocido por su papel en la transcripción mediante la remodelación de la cromatina mediante la PARilación de histonas y la relajación de la estructura de la cromatina, lo que permite que el complejo de transcripción acceda a los genes.
PARP-1 y PARP-2 se activan mediante roturas de una sola cadena de ADN, y tanto los ratones knockout para PARP-1 como para PARP-2 tienen graves deficiencias en la reparación del ADN y una mayor sensibilidad a los agentes alquilantes o la radiación ionizante. [10]
Actividad de PARP (que se debe principalmente a PARP1) medida en células sanguíneas de leucocitos mononucleares permeabilizados de trece especies de mamíferos (rata, cobaya, conejo, tití, oveja, cerdo, ganado vacuno, chimpancé pigmeo, caballo, burro, gorila, elefante y hombre). ) se correlaciona con la esperanza de vida máxima de la especie. [11] La diferencia en actividad entre las especies más longevas (humanos) y más cortas (ratas) analizadas fue cinco veces mayor. Aunque la cinética enzimática (constante de velocidad unimolecular (kcat), Km y kcat/km) de las dos enzimas no fue significativamente diferente, se encontró que la PARP-1 humana tenía una capacidad de automodificación específica dos veces mayor que la enzima de rata, que Los autores postularon que podrían explicar, en parte, la mayor actividad de PARP en humanos que en ratas. [12] Las líneas celulares linfoblastoides establecidas a partir de muestras de sangre de humanos centenarios (100 años o más) tienen una actividad PARP significativamente mayor que las líneas celulares de individuos más jóvenes (20 a 70 años), [13] lo que nuevamente indica un vínculo entre la longevidad y capacidad de reparación.
Estos hallazgos sugieren que la capacidad de reparación del ADN mediada por PARP contribuye a la longevidad de los mamíferos. Por lo tanto, estos hallazgos respaldan la teoría del envejecimiento del daño del ADN , que supone que el daño no reparado del ADN es la causa subyacente del envejecimiento y que la capacidad de reparación del ADN contribuye a la longevidad. [14] [15]
Las tankirasas (TNK) son PARP que comprenden repeticiones de anquirina , un dominio de oligomerización (SAM) y un dominio catalítico de PARP (PCD). Las tankirasas también se conocen como PARP-5a y PARP-5b. Fueron nombrados por su interacción con las proteínas TERF1 asociadas a los telómeros y las repeticiones de anquirina. Pueden permitir la eliminación de complejos inhibidores de la telomerasa de los extremos de los cromosomas para permitir el mantenimiento de los telómeros. A través de su dominio SAM y ANK, pueden oligomerizar e interactuar con muchas otras proteínas, como TRF1, TAB182 ( TNKS1BP1 ), GRB14 , IRAP, NuMa, EBNA-1 y Mcl-1 . Tienen múltiples funciones en la célula, como el tráfico vesicular a través de su interacción en las vesículas GLUT4 con la aminopeptidasa sensible a la insulina (IRAP). También desempeña un papel en el ensamblaje del huso mitótico a través de su interacción con la proteína 1 del aparato mitótico nuclear (NuMa), permitiendo así la orientación bipolar necesaria . En ausencia de TNK, la detención de la mitosis se observa en la preanafase a través del punto de control del huso Mad2 . Las TNK también pueden PARsilar Mcl-1L y Mcl-1S e inhibir su función pro y antiapoptótica; Aún no se conoce la relevancia de esto.
PARP se puede activar en células que experimentan estrés y/o daño en el ADN. La PARP activada puede agotar el ATP de la célula en un intento de reparar el ADN dañado. El agotamiento del ATP en una célula provoca lisis y muerte celular (necrosis). [16] [17] PARP también tiene la capacidad de inducir la muerte celular programada, mediante la producción de PAR, que estimula las mitocondrias para liberar AIF . [18] Este mecanismo parece ser independiente de caspasa. La escisión de PARP, mediante enzimas como caspasas o catepsinas, normalmente inactiva PARP. El tamaño de los fragmentos de escisión puede dar una idea de qué enzima fue responsable de la escisión y puede ser útil para determinar qué vía de muerte celular se ha activado.
El factor de unión a CCCTC ( CTCF ) induce la acumulación de PAR. [19] Los polímeros de ADP-ribosa, ya sea libres o unidos a PARP1, son capaces de inhibir la actividad de la ADN metiltransferasa en los sitios CpG . [20] Por lo tanto, CTCF participa en la interacción entre la poli(ADP-ribosil)ación y la metilación del ADN, un importante factor regulador epigenético. [19]
Se ha acumulado un conjunto sustancial de datos preclínicos y clínicos sobre los inhibidores de PARP en diversas formas de cáncer. En este contexto, el papel de PARP en la reparación de roturas de ADN monocatenario es relevante, lo que conduce a lesiones asociadas a la replicación que no pueden repararse si la reparación por recombinación homóloga (HRR) es defectuosa y conduce a la letalidad sintética de los inhibidores de PARP en HRR- cáncer defectuoso. Los defectos de HRR se asocian clásicamente con mutaciones BRCA1 y 2 asociadas con cáncer de mama y de ovario familiar, pero puede haber muchas otras causas de defectos de HRR. Por lo tanto, los inhibidores de PARP de varios tipos (por ejemplo, olaparib) para los cánceres de mama y de ovario con mutaciones BRCA pueden extenderse más allá de estos tumores si se pueden desarrollar biomarcadores apropiados para identificar defectos de HRR. Existen varias clases adicionales de nuevos inhibidores de PARP que se encuentran en diversas etapas de desarrollo clínico. [21]
Otro conjunto sustancial de datos se relaciona con el papel de PARP en indicaciones no oncológicas seleccionadas. En una serie de enfermedades agudas y graves (como accidentes cerebrovasculares, neurotraumatismos, shock circulatorio e infarto agudo de miocardio), los inhibidores de PARP ejercen un beneficio terapéutico (p. ej., reducción del tamaño del infarto o mejora de la función de los órganos). También hay datos de observación que demuestran la activación de PARP en muestras de tejido humano. En estas indicaciones de enfermedad, la sobreactivación de PARP debido al estrés oxidativo y nitrativo impulsa la necrosis celular y la expresión de genes proinflamatorios, lo que contribuye a la patología de la enfermedad. A medida que avanzan los ensayos clínicos con inhibidores de PARP en diversas formas de cáncer, se espera que se inicie una segunda línea de investigaciones clínicas, destinada a probar inhibidores de PARP para diversas indicaciones no oncológicas, en un proceso llamado "reutilización terapéutica". . [22]
PARP se inactiva por escisión de caspasa . Se cree que la inactivación normal ocurre en sistemas donde el daño al ADN es extenso. En estos casos, se invertiría más energía de la que es factible en reparar el daño, de modo que la energía se recupera para otras células del tejido mediante la muerte celular programada. Además de la degradación, existe evidencia reciente sobre mecanismos reversibles de regulación negativa de PARP, entre ellos un "bucle autorregulador", que es impulsado por el propio PARP1 y modulado por el factor de transcripción YY1 . [23]
Si bien la escisión in vitro por caspasa ocurre en toda la familia de las caspasas, los datos preliminares sugieren que las caspasas-3 y caspasa-7 son responsables de la escisión in vivo . La escisión se produce en el ácido aspártico 214 y la glicina 215, separando PARP en un segmento de 24 kDa y 89 kDa. El resto más pequeño incluye el motivo del dedo de zinc necesario para la unión al ADN. El fragmento de 89 kDa incluye el dominio de automodificación y el dominio catalítico. El supuesto mecanismo de activación de PCD mediante la inactivación de PARP se basa en la separación de la región de unión al ADN y el dominio de automodificación. La región de unión al ADN es capaz de hacerlo independientemente del resto de la proteína, escindida o no. Sin embargo, no puede disociarse sin el dominio de modificación automática. De esta manera, el dominio de unión al ADN se unirá a un sitio dañado y no podrá efectuar la reparación, ya que ya no tiene el dominio catalítico. El dominio de unión al ADN evita que otras PARP no escindidas accedan al sitio dañado e inicien reparaciones. Este modelo sugiere que este "tapón de azúcar" también puede iniciar la señal de apoptosis.
Se han estudiado las funciones de la poli(ADP-ribosil)ación en las respuestas de las plantas al daño del ADN, la infección y otros tipos de estrés. [24] [25] La PARP1 vegetal es muy similar a la PARP1 animal, pero curiosamente, en Arabidopsis thaliana y presumiblemente en otras plantas, PARP2 desempeña funciones más importantes que PARP1 en las respuestas protectoras al daño del ADN y la patogénesis bacteriana. [26] La planta PARP2 porta dominios reguladores y catalíticos de PARP con solo una similitud intermedia con PARP1, y porta motivos de unión al ADN de SAP N-terminal en lugar de los motivos de unión al ADN con dedos de zinc de las proteínas PARP1 de plantas y animales. [26]