Las bases de datos biológicas son bibliotecas de ciencias biológicas, recopiladas a partir de experimentos científicos, literatura publicada, tecnología experimental de alto rendimiento y análisis computacional. [ cita requerida ] Contienen información de áreas de investigación que incluyen genómica , proteómica , metabolómica , expresión génica de microarrays y filogenética . [2] La información contenida en las bases de datos biológicas incluye la función genética, la estructura, la localización (tanto celular como cromosómica), los efectos clínicos de las mutaciones, así como las similitudes de las secuencias y estructuras biológicas.
Las bases de datos biológicas se pueden clasificar según el tipo de datos que recopilan (véase más adelante). En líneas generales, existen bases de datos moleculares (para secuencias, moléculas, etc.), bases de datos funcionales (para fisiología, actividades enzimáticas, fenotipos, ecología, etc.), bases de datos taxonómicas (para especies y otros rangos taxonómicos), imágenes y otros medios, o especímenes (para colecciones de museos, etc.).
Las bases de datos son herramientas importantes para ayudar a los científicos a analizar y explicar una gran cantidad de fenómenos biológicos, desde la estructura de las biomoléculas y su interacción hasta el metabolismo completo de los organismos y la comprensión de la evolución de las especies . Este conocimiento ayuda a facilitar la lucha contra las enfermedades, ayuda al desarrollo de medicamentos , a predecir ciertas enfermedades genéticas y a descubrir relaciones básicas entre las especies en la historia de la vida .
Los conceptos de bases de datos relacionales de la informática y los conceptos de recuperación de información de las bibliotecas digitales son importantes para comprender las bases de datos biológicas. El diseño, desarrollo y gestión a largo plazo de bases de datos biológicas es un área central de la disciplina de la bioinformática . [3] Los contenidos de los datos incluyen secuencias de genes, descripciones textuales, atributos y clasificaciones ontológicas , citas y datos tabulares. Estos suelen describirse como datos semiestructurados y pueden representarse como tablas, registros delimitados por claves y estructuras XML . [ cita requerida ]
La mayoría de las bases de datos biológicas están disponibles a través de sitios web que organizan los datos de forma que los usuarios puedan navegar por ellos en línea. Además, los datos subyacentes suelen estar disponibles para su descarga en una variedad de formatos. Los datos biológicos vienen en muchos formatos. Estos formatos incluyen texto, datos de secuencias, estructura de proteínas y enlaces. Cada uno de ellos se puede encontrar en determinadas fuentes, por ejemplo: [ cita requerida ]
El conocimiento biológico se distribuye entre innumerables bases de datos, lo que a veces dificulta garantizar la coherencia de la información, por ejemplo, cuando se utilizan nombres diferentes para la misma especie o formatos de datos diferentes. En consecuencia, la interoperabilidad es un desafío constante para el intercambio de información. Por ejemplo, si una base de datos de secuencias de ADN almacena la secuencia de ADN junto con el nombre de una especie, un cambio de nombre de esa especie puede romper los vínculos con otras bases de datos que pueden utilizar un nombre diferente. La bioinformática integradora es un campo que intenta abordar este problema proporcionando un acceso unificado. Una solución es la forma en que las bases de datos biológicas se referencian de forma cruzada con otras bases de datos con números de acceso para vincular sus conocimientos relacionados (por ejemplo, de modo que el número de acceso permanezca igual incluso si cambia el nombre de una especie). La redundancia es otro problema, ya que muchas bases de datos deben almacenar la misma información, por ejemplo, las bases de datos de estructura de proteínas también contienen la secuencia de las proteínas que cubren, su secuencia y su información bibliográfica.
Existen bases de datos específicas para algunas especies, principalmente aquellas que se utilizan a menudo en la investigación ( organismos modelo ). Por ejemplo, EcoCyc es una base de datos de E. coli . Otras bases de datos de organismos modelo populares incluyen Mouse Genome Informatics para el ratón de laboratorio , Mus musculus , Rat Genome Database para Rattus , ZFIN para Danio Rerio (pez cebra), PomBase [4] para la levadura de fisión Schizosaccharomyces pombe , FlyBase para Drosophila , WormBase para los nematodos Caenorhabditis elegans y Caenorhabditis briggsae , y Xenbase para las ranas Xenopus tropicalis y Xenopus laevis .
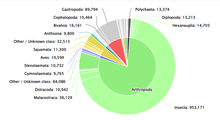
Numerosas bases de datos intentan documentar la diversidad de la vida en la Tierra. Un ejemplo destacado es el Catálogo de la Vida , creado por primera vez en 2001 por Species 2000 y el Sistema Integrado de Información Taxonómica. [6] El Catálogo de la Vida es un proyecto colaborativo que tiene como objetivo documentar la categorización taxonómica de todas las especies actualmente aceptadas en el mundo. [7] El Catálogo de la Vida proporciona una base de datos consolidada y consistente para que los investigadores y los responsables de las políticas puedan consultarla. El Catálogo de la Vida conserva conjuntos de datos actualizados de otras fuentes, como la base de datos Conifer, ICTV MSL (para virus) y LepIndex (para mariposas y polillas). En total, el Catálogo de la Vida se nutre de 165 bases de datos a mayo de 2022. [8] Los costos operativos del Catálogo de la Vida son pagados por el Global Biodiversity Information Facility , el Illinois Natural History Survey , el Naturalis Biodiversity Center y el Smithsonian Institution . [9]
Algunas bases de datos biológicas también documentan la distribución geográfica de diferentes especies. Shuang Dai et al. crearon una nueva base de datos de múltiples fuentes para documentar la distribución espacial/geográfica de 1.371 especies de aves en China, ya que las bases de datos existentes habían carecido gravemente de datos de distribución espacial para muchas especies. [10] Las fuentes para esta nueva base de datos incluían libros, literatura, seguimiento por GPS y datos de páginas web en línea. La nueva base de datos mostraba taxonomía, distribución, información sobre las especies y fuentes de datos para cada especie. Después de completar la base de datos de distribución espacial de las aves, se descubrió que el 61% de las especies conocidas en China se distribuían en regiones más allá de donde se las conocía anteriormente. [11]

Las bases de datos médicas son un caso especial de recursos de datos biomédicos y pueden abarcar desde bibliografías, como PubMed , hasta bases de datos de imágenes para el desarrollo de software de diagnóstico basado en IA. Por ejemplo, una de esas bases de datos de imágenes se desarrolló con el objetivo de ayudar en el desarrollo de algoritmos de monitoreo de heridas. [13] Se seleccionaron más de 188 conjuntos de imágenes multimodales de 79 visitas de pacientes, que consistían en fotografías, imágenes térmicas y mapas de profundidad de malla 3D. Los contornos de las heridas se dibujaron manualmente y se agregaron a los conjuntos de datos de fotografías. [14] La base de datos se puso a disposición del público en forma de un programa llamado WoundsDB, que se puede descargar desde el sitio web de Chronic Wound Database.
Un recurso importante para encontrar bases de datos biológicas es un número especial anual de la revista Nucleic Acids Research (NAR). El número de bases de datos de la NAR está disponible de forma gratuita y clasifica muchas de las bases de datos biológicas públicas. Una base de datos complementaria al número, llamada Online Molecular Biology Database Collection, enumera 1.380 bases de datos en línea. [15] Existen otras colecciones de bases de datos, como MetaBase y Bioinformatics Links Collection. [16] [17]