En biología molecular , la mutagénesis es una importante técnica de laboratorio mediante la cual las mutaciones del ADN se modifican deliberadamente para producir bibliotecas de genes, proteínas, cepas de bacterias u otros organismos genéticamente modificados . Los diversos constituyentes de un gen, así como sus elementos reguladores y sus productos genéticos, pueden mutarse de modo que pueda examinarse en detalle el funcionamiento de un locus, proceso o producto genético. La mutación puede producir proteínas mutantes con propiedades interesantes o funciones mejoradas o novedosas que pueden ser de uso comercial. También se pueden producir cepas mutantes que tengan una aplicación práctica o permitan investigar las bases moleculares de una función celular particular.
Hoy en día existen muchos métodos de mutagénesis. Inicialmente, el tipo de mutaciones inducidas artificialmente en el laboratorio eran completamente aleatorias mediante mecanismos como la irradiación ultravioleta. La mutagénesis aleatoria no puede apuntar a regiones o secuencias específicas del genoma; sin embargo, con el desarrollo de la mutagénesis dirigida al sitio , se pueden realizar cambios más específicos. Desde 2013, el desarrollo de la tecnología CRISPR /Cas9, basada en un sistema de defensa viral procariota, ha permitido la edición o mutagénesis de un genoma in vivo . [1] La mutagénesis dirigida al sitio ha demostrado ser útil en situaciones en las que la mutagénesis aleatoria no lo es. Otras técnicas de mutagénesis incluyen mutagénesis combinatoria y de inserción. La mutagénesis que no es aleatoria se puede utilizar para clonar ADN, [2] investigar los efectos de mutágenos, [3] y diseñar proteínas. [4] También tiene aplicaciones médicas como la ayuda a pacientes inmunodeprimidos, la investigación y el tratamiento de enfermedades como el VIH y el cáncer, y la curación de enfermedades como la beta talasemia . [5]

Los primeros enfoques de la mutagénesis se basaban en métodos que producían mutaciones totalmente aleatorias. En tales métodos, las células u organismos se exponen a mutágenos como la radiación UV o sustancias químicas mutagénicas, y luego se seleccionan mutantes con las características deseadas. Hermann Muller descubrió en 1927 que los rayos X pueden provocar mutaciones genéticas en las moscas de la fruta , [6] y pasó a utilizar los mutantes que creó para sus estudios de genética . [7] Para Escherichia coli , los mutantes pueden seleccionarse primero mediante exposición a radiación UV y luego sembrarse en placas en un medio de agar. Luego, las colonias formadas se replican en placas , una en un medio rico , otra en un medio mínimo, y los mutantes que tienen requisitos nutricionales específicos pueden identificarse por su incapacidad para crecer en el medio mínimo. Se pueden repetir procedimientos similares con otros tipos de células y con diferentes medios de selección.
Posteriormente se desarrollaron varios métodos para generar mutaciones aleatorias en proteínas específicas para detectar mutantes con propiedades interesantes o mejoradas. Estos métodos pueden implicar el uso de nucleótidos dopados en la síntesis de oligonucleótidos o la realización de una reacción de PCR en condiciones que mejoren la incorporación errónea de nucleótidos (PCR propensa a errores), por ejemplo, reduciendo la fidelidad de replicación o usando análogos de nucleótidos. [8] Una variación de este método para integrar mutaciones no sesgadas en un gen es la mutagénesis por saturación de secuencia . [9] Los productos de la PCR que contienen mutaciones se clonan luego en un vector de expresión y luego se pueden caracterizar las proteínas mutantes producidas.
En estudios con animales, se han utilizado agentes alquilantes como la N -etil- N -nitrosourea (ENU) para generar ratones mutantes. [10] [11] El metanosulfonato de etilo (EMS) también se utiliza a menudo para generar mutantes de animales, plantas y virus. [12] [13] [14]
En una ley de la Unión Europea (como la directiva 2001/18), este tipo de mutagénesis puede usarse para producir OGM, pero los productos están exentos de regulación: sin etiquetado, sin evaluación. [15]
Antes del desarrollo de técnicas de mutagénesis dirigida al sitio, todas las mutaciones realizadas eran aleatorias y los científicos tenían que utilizar la selección del fenotipo deseado para encontrar la mutación deseada. Las técnicas de mutagénesis aleatoria tienen una ventaja en cuanto a cuántas mutaciones se pueden producir; sin embargo, si bien la mutagénesis aleatoria puede producir un cambio en nucleótidos individuales, no ofrece mucho control sobre qué nucleótido se está cambiando. [5] Por lo tanto, muchos investigadores buscan introducir cambios seleccionados en el ADN de una manera precisa y específica del sitio. Los primeros intentos de utilizar análogos de nucleótidos y otras sustancias químicas se utilizaron por primera vez para generar mutaciones puntuales localizadas . [16] Tales sustancias químicas incluyen aminopurina , que induce una transición de AT a GC , [17] mientras que nitrosoguanidina , [18] bisulfito , [19] y N 4 -hidroxicitidina pueden inducir una transición de GC a AT. [20] [21] Estas técnicas permiten diseñar mutaciones específicas en una proteína; sin embargo, no son flexibles con respecto a los tipos de mutantes generados, ni son tan específicos como métodos posteriores de mutagénesis dirigida y, por lo tanto, tienen cierto grado de aleatoriedad. Gracias a otras tecnologías, como la escisión del ADN en sitios específicos del cromosoma, la adición de nuevos nucleótidos y el intercambio de pares de bases, ahora es posible decidir hacia dónde pueden ir las mutaciones. [11] [8]
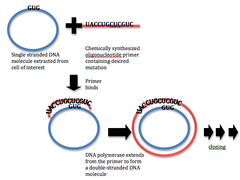
Las técnicas actuales para la mutación específica de sitio se originan a partir de la técnica de extensión del cebador desarrollada en 1978. Dichas técnicas comúnmente implican el uso de oligonucleótidos mutagénicos prefabricados en una reacción de extensión del cebador con ADN polimerasa . Este método permite la mutación puntual, la eliminación o la inserción de pequeños tramos de ADN en sitios específicos. Los avances en metodología han hecho que dicha mutagénesis sea ahora un proceso relativamente simple y eficiente. [3]
Constantemente se desarrollan métodos más nuevos y más eficientes de mutagénesis dirigida al sitio. Por ejemplo, una técnica llamada "extracto de clonación por ligación perfecta" (o SLiCE para abreviar) permite la clonación de ciertas secuencias de ADN dentro del genoma, y se puede insertar más de un fragmento de ADN en el genoma a la vez. [2]
La mutagénesis dirigida al sitio permite investigar el efecto de una mutación específica. Existen numerosos usos; por ejemplo, se ha utilizado para determinar qué tan susceptibles eran ciertas especies a los químicos que se usan a menudo en los laboratorios. El experimento utilizó mutagénesis dirigida al sitio para imitar las mutaciones esperadas de la sustancia química específica. La mutación resultó en un cambio en aminoácidos específicos y se analizaron los efectos de esta mutación. [3]

El enfoque dirigido al sitio se puede realizar sistemáticamente en técnicas tales como la mutagénesis de barrido de alanina , mediante la cual los residuos se mutan sistemáticamente a alanina para identificar residuos importantes para la estructura o función de una proteína. [22] Otro enfoque integral es la mutagénesis de saturación de sitio , donde un codón o un conjunto de codones pueden sustituirse con todos los aminoácidos posibles en posiciones específicas. [23] [24]
La mutagénesis combinatoria es una técnica de ingeniería de proteínas dirigida al sitio mediante la cual se pueden diseñar simultáneamente múltiples mutantes de una proteína basándose en el análisis de los efectos de mutaciones individuales aditivas. [25] Proporciona un método útil para evaluar el efecto combinatorio de un gran número de mutaciones en la función de las proteínas. [26] Se pueden seleccionar grandes cantidades de mutantes para detectar una característica particular mediante análisis combinatorio. [25] En esta técnica, se pueden modificar exhaustivamente múltiples posiciones o secuencias cortas a lo largo de una cadena de ADN para obtener una biblioteca completa de proteínas mutantes. [25] La tasa de incidencia de variantes beneficiosas se puede mejorar mediante diferentes métodos para construir bibliotecas de mutagénesis. Un enfoque de esta técnica es extraer y reemplazar una porción de la secuencia de ADN con una biblioteca de secuencias que contenga todas las combinaciones posibles en el sitio de mutación deseado. El contenido del segmento insertado puede incluir secuencias de importancia estructural, propiedad inmunogénica o función enzimática. También se puede insertar un segmento aleatoriamente en el gen para evaluar la importancia estructural o funcional de una parte particular de una proteína. [25]
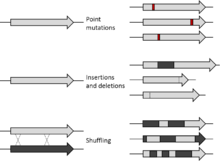
La inserción de uno o más pares de bases, que dan como resultado mutaciones en el ADN, también se conoce como mutagénesis por inserción . [27] Mutaciones diseñadas como estas pueden proporcionar información importante en la investigación del cáncer, como conocimientos mecanicistas sobre el desarrollo de la enfermedad. Los retrovirus y los transposones son las principales herramientas instrumentales en la mutagénesis por inserción. Los retrovirus, como el virus del tumor mamario de ratón y el virus de la leucemia murina, se pueden utilizar para identificar genes implicados en la carcinogénesis y comprender las vías biológicas de cánceres específicos. [28] Los transposones, segmentos cromosómicos que pueden sufrir transposición, pueden diseñarse y aplicarse a la mutagénesis por inserción como instrumento para el descubrimiento de genes cancerosos. [28] Estos segmentos cromosómicos permiten que la mutagénesis por inserción se aplique a prácticamente cualquier tejido de elección y al mismo tiempo permiten una profundidad más completa e imparcial en la secuenciación del ADN. [28]
Los investigadores han encontrado cuatro mecanismos de mutagénesis por inserción que pueden utilizarse en humanos. El primer mecanismo se llama inserción de potenciador. Los potenciadores estimulan la transcripción de un gen particular al interactuar con un promotor de ese gen. Este mecanismo particular se utilizó por primera vez para ayudar a los pacientes gravemente inmunocomprometidos que necesitaban médula ósea. Luego se insertaron gammaretrovirus portadores de potenciadores en los pacientes. El segundo mecanismo se conoce como inserción de promotor. Los promotores proporcionan a nuestras células las secuencias específicas necesarias para comenzar la traducción. La inserción de promotores ha ayudado a los investigadores a aprender más sobre el virus del VIH. El tercer mecanismo es la inactivación de genes. Un ejemplo de inactivación genética es el uso de mutagénesis por inserción para insertar un retrovirus que altera el genoma de las células T en pacientes con leucemia y les proporciona un antígeno específico llamado CAR que permite que las células T se dirijan a las células cancerosas. El mecanismo final se denomina sustitución del extremo 3' del ARNm. Nuestros genes ocasionalmente sufren mutaciones puntuales que causan beta-talasemia que interrumpe la función de los glóbulos rojos. Para solucionar este problema, se introduce la secuencia genética correcta de los glóbulos rojos y se realiza una sustitución. [5]
La recombinación homóloga se puede utilizar para producir una mutación específica en un organismo. Se introduce en la célula un vector que contiene una secuencia de ADN similar al gen que se va a modificar y, mediante un proceso de recombinación, reemplaza el gen diana en el cromosoma. Este método se puede utilizar para introducir una mutación o desactivar un gen, por ejemplo como se utiliza en la producción de ratones knockout . [29]
Desde 2013, el desarrollo de la tecnología CRISPR -Cas9 ha permitido la introducción eficiente de diferentes tipos de mutaciones en el genoma de una amplia variedad de organismos. El método no requiere un sitio de inserción de transposón, no deja marcador y su eficiencia y simplicidad lo han convertido en el método preferido para la edición del genoma . [30] [31]
A medida que disminuye el costo de la síntesis de oligonucleótidos de ADN, la síntesis artificial de un gen completo es ahora un método viable para introducir mutaciones en un gen. Este método permite una mutación extensa en múltiples sitios, incluido el rediseño completo del uso de codones de un gen para optimizarlo para un organismo en particular. [32]