La mutagénesis dirigida es un método de biología molecular que se utiliza para realizar cambios mutacionales específicos e intencionales en la secuencia de ADN de un gen y en cualquier producto génico . También llamada mutagénesis específica del sitio o mutagénesis dirigida por oligonucleótidos , se utiliza para investigar la estructura y la actividad biológica de las moléculas de ADN , ARN y proteínas , y para la ingeniería de proteínas .
La mutagénesis dirigida es una de las técnicas de laboratorio más importantes para crear bibliotecas de ADN mediante la introducción de mutaciones en secuencias de ADN. Existen numerosos métodos para lograr la mutagénesis dirigida, pero con la disminución de los costos de la síntesis de oligonucleótidos , la síntesis artificial de genes se utiliza ahora ocasionalmente como una alternativa a la mutagénesis dirigida. Desde 2013, el desarrollo de la tecnología CRISPR /Cas9, basada en un sistema de defensa viral procariota, también ha permitido la edición del genoma , y la mutagénesis puede realizarse in vivo con relativa facilidad. [1]
Los primeros intentos de mutagénesis utilizando radiación o mutágenos químicos no eran específicos del sitio, generando mutaciones aleatorias. [2] Posteriormente se utilizaron análogos de nucleótidos y otros productos químicos para generar mutaciones puntuales localizadas , [3] ejemplos de dichos productos químicos son aminopurina , [4] nitrosoguanidina , [5] y bisulfito . [6] La mutagénesis dirigida al sitio se logró en 1974 en el laboratorio de Charles Weissmann utilizando un análogo de nucleótido N 4 -hidroxicitidina, que induce la transición de GC a AT. [7] [8] Sin embargo, estos métodos de mutagénesis están limitados por el tipo de mutación que pueden lograr, y no son tan específicos como los métodos de mutagénesis dirigida al sitio posteriores.
En 1971, Clyde Hutchison y Marshall Edgell demostraron que es posible producir mutantes con pequeños fragmentos del fago ϕX174 y nucleasas de restricción . [9] [10] Hutchison produjo más tarde con su colaborador Michael Smith en 1978 un enfoque más flexible para la mutagénesis dirigida al sitio mediante el uso de oligonucleótidos en un método de extensión de cebadores con ADN polimerasa. [11] Por su parte en el desarrollo de este proceso, Michael Smith compartió más tarde el Premio Nobel de Química en octubre de 1993 con Kary B. Mullis , quien inventó la reacción en cadena de la polimerasa .
El procedimiento básico requiere la síntesis de un cebador de ADN corto. Este cebador sintético contiene la mutación deseada y es complementario al ADN molde alrededor del sitio de mutación para que pueda hibridar con el ADN en el gen de interés. La mutación puede ser un cambio de una sola base (una mutación puntual ), cambios de múltiples bases, deleción o inserción . El cebador de cadena sencilla se extiende luego utilizando una ADN polimerasa , que copia el resto del gen. El gen así copiado contiene el sitio mutado y luego se introduce en una célula huésped en un vector y se clona . Finalmente, los mutantes se seleccionan mediante secuenciación de ADN para verificar que contienen la mutación deseada.
El método original que utilizaba la extensión con un único cebador era ineficiente debido a la baja producción de mutantes. La mezcla resultante contiene tanto la plantilla original no mutada como la cadena mutante, lo que produce una población mixta de progenies mutantes y no mutantes. Además, la plantilla utilizada está metilada mientras que la cadena mutante no está metilada, y los mutantes pueden ser contraseleccionados debido a la presencia de un sistema de reparación de desajustes que favorece al ADN de plantilla metilado, lo que da como resultado menos mutantes. Desde entonces se han desarrollado muchos enfoques para mejorar la eficiencia de la mutagénesis.
Hay una gran cantidad de métodos disponibles para efectuar mutagénesis dirigida al sitio, [12] aunque la mayoría de ellos rara vez se han utilizado en laboratorios desde principios de la década de 2000, ya que las técnicas más nuevas permiten formas más simples y fáciles de introducir mutaciones específicas del sitio en los genes.
En 1985, Thomas Kunkel introdujo una técnica que reduce la necesidad de seleccionar mutantes. [13] El fragmento de ADN que se va a mutar se inserta en un fagémido como M13mp18/19 y luego se transforma en una cepa de E. coli deficiente en dos enzimas, dUTPasa ( dut ) y uracilo desglicosidasa ( udg ). Ambas enzimas son parte de una vía de reparación del ADN que protege al cromosoma bacteriano de mutaciones por la desaminación espontánea de dCTP a dUTP. La deficiencia de dUTPasa previene la descomposición de dUTP, lo que resulta en un alto nivel de dUTP en la célula. La deficiencia de uracilo desglicosidasa previene la eliminación de uracilo del ADN recién sintetizado. A medida que la E. coli doblemente mutante replica el ADN del fago, su maquinaria enzimática puede, por lo tanto, incorporar incorrectamente dUTP en lugar de dTTP, lo que resulta en ADN de cadena sencilla que contiene algunos uracilos (ssUDNA). El ssUDNA se extrae del bacteriófago que se libera en el medio y luego se utiliza como plantilla para la mutagénesis. Se utiliza un oligonucleótido que contiene la mutación deseada para la extensión del cebador. El ADN heterodúplex que se forma consta de una cadena parental no mutada que contiene dUTP y una cadena mutada que contiene dTTP. Luego, el ADN se transforma en una cepa de E. coli que porta los genes dut y udg de tipo salvaje . Aquí, la cadena de ADN parental que contiene uracilo se degrada, de modo que casi todo el ADN resultante consiste en la cadena mutada.
A diferencia de otros métodos, la mutagénesis en casete no necesita implicar la extensión de cebadores utilizando la ADN polimerasa. En este método, se sintetiza un fragmento de ADN y luego se inserta en un plásmido. [14] Implica la escisión por una enzima de restricción en un sitio del plásmido y la posterior ligadura de un par de oligonucleótidos complementarios que contienen la mutación en el gen de interés al plásmido. Por lo general, las enzimas de restricción que cortan en el plásmido y el oligonucleótido son las mismas, lo que permite que los extremos pegajosos del plásmido y el inserto se liguen entre sí. Este método puede generar mutantes con una eficiencia cercana al 100%, pero está limitado por la disponibilidad de sitios de restricción adecuados que flanqueen el sitio que se va a mutar.
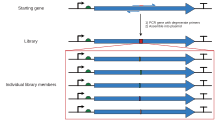
La limitación de los sitios de restricción en la mutagénesis en casete puede superarse utilizando la reacción en cadena de la polimerasa con " cebadores " de oligonucleótidos , de modo que se pueda generar un fragmento más grande, que cubra dos sitios de restricción convenientes. La amplificación exponencial en PCR produce un fragmento que contiene la mutación deseada en cantidad suficiente para separarse del plásmido original no mutado mediante electroforesis en gel , que luego se puede insertar en el contexto original utilizando técnicas estándar de biología molecular recombinante. Existen muchas variaciones de la misma técnica. El método más simple coloca el sitio de mutación hacia uno de los extremos del fragmento, por lo que uno de los dos oligonucleótidos utilizados para generar el fragmento contiene la mutación. Esto implica un solo paso de PCR, pero aún tiene el problema inherente de requerir un sitio de restricción adecuado cerca del sitio de mutación a menos que se utilice un cebador muy largo. Otras variaciones, por lo tanto, emplean tres o cuatro oligonucleótidos, dos de los cuales pueden ser oligonucleótidos no mutagénicos que cubran dos sitios de restricción convenientes y generen un fragmento que se puede digerir y ligar en un plásmido, mientras que el oligonucleótido mutagénico puede ser complementario a una ubicación dentro de ese fragmento bien alejada de cualquier sitio de restricción conveniente. Estos métodos requieren múltiples pasos de PCR para que el fragmento final que se ligará pueda contener la mutación deseada. El proceso de diseño para generar un fragmento con la mutación deseada y los sitios de restricción relevantes puede ser engorroso. Herramientas de software como SDM-Assist [15] pueden simplificar el proceso.
Para las manipulaciones de plásmidos, otras técnicas de mutagénesis dirigida al sitio han sido suplantadas en gran medida por técnicas que son altamente eficientes pero relativamente simples, fáciles de usar y disponibles comercialmente como un kit. Un ejemplo de estas técnicas es el método "Quikchange", [16] en el que se utiliza un par de cebadores mutagénicos complementarios para amplificar el plásmido completo en una reacción de termociclado utilizando una ADN polimerasa de alta fidelidad que no desplaza la cadena, como la polimerasa Pfu . La reacción genera un ADN circular mellado . El ADN molde debe eliminarse mediante digestión enzimática con una enzima de restricción como Dpn I , que es específica para el ADN metilado. Todo el ADN producido a partir de la mayoría de las cepas de Escherichia coli estaría metilado; el plásmido molde que se biosintetiza en E. coli , por lo tanto, se digerirá, mientras que el plásmido mutado, que se genera in vitro y, por lo tanto, no está metilado, se dejaría sin digerir. Obsérvese que, en estos métodos de mutagénesis de plásmidos de doble cadena, si bien se puede utilizar la reacción de termociclado, el ADN no se amplifica exponencialmente si los dos cebadores están diseñados de manera que se unan simétricamente a la misma región alrededor del sitio de mutagénesis, como se describe en el protocolo original. En este caso, la amplificación es lineal y, por lo tanto, es inexacto describir el procedimiento como una PCR, ya que no hay reacción en cadena. Sin embargo, si los cebadores están diseñados para unirse de manera desfasada de manera que el sitio de mutagénesis esté cerca del extremo 5' de ambos cebadores, la región 3' de los cebadores también puede unirse a los productos amplificados y, por lo tanto, se observa la formación exponencial del producto. El nombre "Quikchange" se origina de la marca registrada "QuikChange mutagenesis" de Stratagene , ahora Agilent Technologies , para kits de mutagénesis dirigida al sitio. El método fue desarrollado por científicos que trabajan en Stratagene. [16]
Tenga en cuenta que la polimerasa Pfu puede desplazar la cadena a una temperatura de extensión más alta (≥70 °C), lo que puede provocar el fracaso del experimento; por lo tanto, la reacción de extensión debe realizarse a la temperatura recomendada de 68 °C. En algunas aplicaciones, se ha observado que este método conduce a la inserción de múltiples copias de cebadores. [17] Una variación de este método, llamada SPRINP, evita este artefacto y se ha utilizado en diferentes tipos de mutagénesis dirigida al sitio. [17]
Otras técnicas, como la mutagénesis por escaneo de objetivos dirigidos a oligonucleótidos (SMOOT), pueden combinar de forma semialeatoria oligonucleótidos mutagénicos en la mutagénesis plasmídica. [18] Esta técnica puede crear bibliotecas de mutagénesis plasmídica que van desde mutaciones individuales hasta mutagénesis de codones integral en un gen entero.
Desde 2013, el desarrollo de la tecnología CRISPR -Cas9 ha permitido introducir de forma eficiente diversas mutaciones en el genoma de una amplia variedad de organismos. El método no requiere un sitio de inserción de transposón, no deja ningún marcador y su eficiencia y simplicidad lo han convertido en el método preferido para la edición genómica . [21] [22]

La mutagénesis dirigida se utiliza para generar mutaciones que puedan producir una proteína diseñada racionalmente que tenga propiedades mejoradas o especiales (es decir, ingeniería de proteínas).
Herramientas de investigación : las mutaciones específicas en el ADN permiten investigar la función y las propiedades de una secuencia de ADN o una proteína de una manera racional. Además, los cambios de aminoácidos individuales mediante mutagénesis dirigida en proteínas pueden ayudar a comprender la importancia de las modificaciones postraduccionales. Por ejemplo, cambiar una serina particular (fosfoaceptor) por una alanina (fosfonoaceptor) en una proteína sustrato bloquea la unión de un grupo fosfato, lo que permite investigar la fosforilación. Este enfoque se ha utilizado para descubrir la fosforilación de la proteína CBP por la quinasa HIPK2 [23]. Otro enfoque integral es la mutagénesis de saturación del sitio , en la que un codón o un conjunto de codones pueden sustituirse con todos los aminoácidos posibles en las posiciones específicas. [24]
Aplicaciones comerciales : las proteínas pueden modificarse para producir formas mutantes que estén diseñadas para una aplicación específica. Por ejemplo, los detergentes para ropa de uso común pueden contener subtilisina , cuya forma silvestre tiene una metionina que puede oxidarse con lejía, lo que reduce significativamente la actividad de la proteína en el proceso. [25] Esta metionina puede reemplazarse por alanina u otros residuos, lo que la hace resistente a la oxidación y mantiene así la proteína activa en presencia de lejía. [26]
A medida que el coste de la síntesis de oligonucleótidos de ADN disminuye, la síntesis artificial de un gen completo es ahora un método viable para introducir mutaciones en un gen. Este método permite una mutagénesis extensa en múltiples sitios, incluido el rediseño completo del uso de codones del gen para optimizarlo para un organismo en particular. [27]
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ){{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace )