En bioquímica, un ribonucleótido es un nucleótido que contiene ribosa como componente pentosa . Se considera un precursor molecular de los ácidos nucleicos . Los nucleótidos son los componentes básicos del ADN y el ARN . Los propios ribonucleótidos son componentes monoméricos básicos del ARN. Los desoxirribonucleótidos , formados mediante la reducción de ribonucleótidos con la enzima ribonucleótido reductasa (RNR), son componentes esenciales del ADN. [1] Existen varias diferencias entre los desoxirribonucleótidos de ADN y los ribonucleótidos de ARN. Los nucleótidos sucesivos están unidos entre sí mediante enlaces fosfodiéster.
Los ribonucleótidos también se utilizan en otras funciones celulares. Estos monómeros especiales se utilizan tanto en la regulación celular como en la señalización celular, como se ve en el monofosfato de adenosina ( AMP ). Además, los ribonucleótidos se pueden convertir en trifosfato de adenosina ( ATP ), la moneda energética de los organismos. Los ribonucleótidos también se pueden convertir en monofosfato de adenosina cíclico ( AMP cíclico ) para regular las hormonas en los organismos. [1] En los organismos vivos, las bases más comunes de los ribonucleótidos son la adenina (A), la guanina (G), la citosina (C) o el uracilo (U). Las bases nitrogenadas se clasifican en dos compuestos originales, purina y pirimidina .

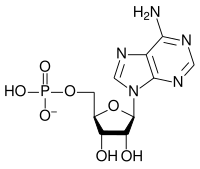
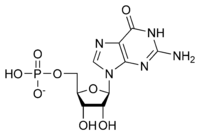


La estructura general de un ribonucleótido consta de un grupo fosfato, un grupo de azúcar ribosa y una nucleobase, en la que la nucleobase puede ser adenina, guanina, citosina o uracilo. Sin el grupo fosfato, la composición de la nucleobase y el azúcar se conoce como nucleósido. Las nucleobases nitrogenadas intercambiables se derivan de dos compuestos originales, purina y pirimidina. Los nucleótidos son compuestos heterocíclicos , es decir, contienen al menos dos elementos químicos diferentes como miembros de sus anillos. [ cita necesaria ]
Tanto el ARN como el ADN contienen dos bases purínicas principales, adenina (A) y guanina (G), y dos pirimidinas principales. Tanto en el ADN como en el ARN, una de las pirimidinas es la citosina (C). Sin embargo, el ADN y el ARN se diferencian en la segunda pirimidina principal. El ADN contiene timina (T) mientras que el ARN contiene uracilo (U). Hay algunos casos raros en los que la timina se encuentra en el ARN y el uracilo en el ADN. [1]
Aquí están los cuatro ribonucleótidos principales (ribonucleósido 5'-monofosfato), que son las unidades estructurales de los ARN.
En los ribonucleótidos, el componente de azúcar es la ribosa, mientras que en los desoxirribonucleótidos, el componente de azúcar es la desoxirribosa. En lugar de un grupo hidroxilo en el segundo carbono del anillo de ribosa, se reemplaza por un átomo de hidrógeno. [2]
Ambos tipos de pentosas en el ADN y el ARN se encuentran en su forma de β-furanosa (anillo cerrado de cinco miembros) y definen la identidad de un ácido nucleico. El ADN se define por contener ácido nucleico 2'-desoxirribosa, mientras que el ARN se define por contener ácido nucleico ribosa. [1]
En algunas ocasiones, el ADN y el ARN pueden contener algunas bases menores. Las formas metiladas de las bases principales son las más comunes en el ADN. En el ADN viral, algunas bases pueden estar hidroximetiladas o glucosiladas. En el ARN, las bases menores o modificadas aparecen con mayor frecuencia. Algunos ejemplos incluyen hipoxantina, dihidrouracilo, formas metiladas de uracilo, citosina y guanina, así como pseudouridina de nucleósido modificado. [3] También se han observado nucleótidos con grupos fosfato en posiciones distintas al carbono 5'. Los ejemplos incluyen ribonucleósidos 2',3'-monofosfatos cíclicos que son intermedios aislables, y ribonucleósidos 3'-monofosfatos que son productos finales de la hidrólisis del ARN por ciertas ribonucleasas. Otras variaciones incluyen adenosina 3',5'-monofosfato cíclico (cAMP) y guanosina 3',5'-monofosfato cíclico (cGMP). [4]
Los ribonucleótidos se unen entre sí para formar cadenas de ARN mediante enlaces fosfodiéster . El grupo 5'-fosfato de un nucleótido está unido al grupo 3'-hidroxilo del siguiente nucleótido, creando una columna vertebral de residuos alternados de fosfato y pentosa. No hay enlace fosfodiéster en cada extremo del polinucleótido. [5] Los enlaces fosfodiéster se forman entre ribonucleótidos mediante la enzima ARN polimerasa . La cadena de ARN se sintetiza desde el extremo 5' al extremo 3' cuando el grupo 3'-hidroxilo del último ribonucleótido de la cadena actúa como nucleófilo y lanza un ataque hidrófilo contra el 5'-trifosfato del ribonucleótido entrante, liberando pirofosfato como subproducto [6] . Debido a las propiedades físicas de los nucleótidos, la columna vertebral del ARN es muy hidrofílica y polar. A pH neutro, los ácidos nucleicos están muy cargados ya que cada grupo fosfato tiene una carga negativa. [7]
Tanto el ADN como el ARN se construyen a partir de nucleósidos fosfatos, también conocidos como monómeros mononucleótidos, que termodinámicamente tienen menos probabilidades de combinarse que los aminoácidos. Los enlaces fosfodiéster, cuando se hidrolizan, liberan una cantidad considerable de energía libre. Por tanto, los ácidos nucleicos tienden a hidrolizarse espontáneamente en mononucleótidos. Los precursores del ARN son GTP, CTP, UTP y ATP, que es una fuente importante de energía en las reacciones de transferencia de grupos. [8]
Los científicos creen que el ARN se desarrolló antes que el ADN. [9]
La reducción de ribonucleótidos a desoxirribonucleótidos está catalizada por la ribonucleótido reductasa . La ribonucleótido reductasa (RNR) es una enzima esencial para todos los organismos vivos ya que es responsable del último paso en la síntesis de los cuatro desoxirribonucleótidos (dNTP) necesarios para la replicación y reparación del ADN. [10] La reacción también requiere otras dos proteínas: tioredoxina y tioredoxina reductasa . La tioredoxina reduce el ribonucleósido difosfato (NDP) a un desoxirribonucleósido difosfato (dNTP).
La reacción general es: Ribonucleósido difosfato + NADPH + H + -> Desoxirribonucleósido difosfato + NADP + + H 2 O [11]
Para ilustrar esta ecuación, dATP y dGTP se sintetizan a partir de ADP y PIB, respectivamente. Primero son reducidos por RNR y luego fosforilados por nucleósido difosfato quinasas a dATP y dGTP. La ribonucleótido reductasa está controlada por interacciones alostéricas. Una vez que dATP se une a la ribonucleótido reductasa, la actividad catalítica general de la enzima disminuye, ya que significa una abundancia de desoxirribonucleótidos. Esta inhibición por retroalimentación se revierte una vez que se une el ATP. [12]
Durante la síntesis de ADN, las ADN polimerasas deben seleccionar contra los ribonucleótidos, presentes en niveles mucho más altos en comparación con los desoxirribonucleótidos. Es crucial que haya selectividad ya que la replicación del ADN debe ser precisa para mantener el genoma del organismo. Se ha demostrado que los sitios activos de las ADN polimerasas de la familia Y son responsables de mantener una alta selectividad contra los ribonucleótidos. [13] La mayoría de las ADN polimerasas también están equipadas para excluir ribonucleótidos de su sitio activo a través de un residuo de cadena lateral voluminoso que puede bloquear estéricamente el grupo 2'-hidroxilo del anillo de ribosa. Sin embargo, muchas ADN polimerasas nucleares de replicación y reparación incorporan ribonucleótidos en el ADN, [14] [15], lo que sugiere que el mecanismo de exclusión no es perfecto. [dieciséis]
Los ribonucleótidos pueden sintetizarse en organismos a partir de moléculas más pequeñas mediante la vía de novo o reciclarse mediante la vía de rescate. En el caso de la vía de novo, tanto las purinas como las pirimidinas se sintetizan a partir de componentes derivados de precursores de aminoácidos, ribosa-5-fosfatos, CO 2 y NH 3 . [1] [17]

La biosíntesis de novo de nucleótidos de purina es bastante compleja y consta de varias reacciones enzimáticas. Utilizando la estructura del azúcar de cinco anillos como base, el anillo de purina se construye con unos pocos átomos a la vez en un proceso de once pasos que conduce a la formación de inosinato (IMP). Básicamente, el IMP se convierte en los nucleótidos de purina necesarios para la síntesis de ácidos nucleicos. [1]
La vía comienza con la conversión de ribosa-5-fosfato (R5P) en fosforribosilpirofosfato (PRPP) por la enzima ribosa-fosfato difosfoquinasa (PRPS1). Luego, el PRPP se convierte en 5-fosforribosilamina (5-PRA) cuando la glutamina dona un grupo amino al C-1 del PRPP. En una reacción de condensación, la enzima GAR sintetasa, junto con la glicina y el ATP, activa el grupo glicina carboxilasa del 5-PRA para formar ribonucleótido de glicinamida (GAR). La coenzima N10-formil-THF, junto con la enzima GAR transformilasa, luego dona una unidad de un carbono al grupo amino de la glicina de GAR, seguido de la adición de glutamina por la enzima FGAR amidotransferasa, lo que lleva a la formación de ribonucleótido de formilglicinamidina (FGAM). ). La deshidratación de FGAM por la enzima FGAM ciclasa da como resultado el cierre del anillo de imidazol, como ribonucleótido de 5-aminoimidazol (AIR). Un grupo carboxilo se une al AIR mediante la N5-CAIR sintetasa para formar el ribonucleótido N5-carboxiaminoimidazol (N5-CAIR), que luego se convierte en ribonucleótido carboxiamino-imidazol (CAIR) con la enzima N5-CAIR mutasa. La enzima SAICAR sintetasa, junto con el grupo amino del aspartato, forma un enlace amida para crear el ribonucleótido N-succinil-5-aminoimidazol-4-carboxamida (SAICAR). Continuando por el camino, la eliminación del esqueleto carbonado del aspartato por la SAICAR liasa da como resultado el ribonucleótido 5-aminoimidazol-4-carboxamida (AICAR). La enzima AICAR transformilasa ayuda en la transferencia final de carbono del N10-formiltetrahidrofolato, formando el ribonucleótido de N-formilaminoimidazol-4-carboxamida (FAICAR). Por último, el cierre de la estructura del segundo anillo lo lleva a cabo la IMP sintasa para formar IMP, donde el destino de IMP conduciría a la formación de un nucleótido de purina. [1]

La síntesis de nucleótidos de pirimidina es un proceso mucho más sencillo. La formación del anillo de pirimidina comienza con la conversión de aspartato en N-carbamoilaspartato mediante una reacción de condensación con carbamoil fosfato. La dihidroorotasa y la dihidroorotasa deshidrogenasa luego convierten el N-carbamoilaspartato en orotato. El orotato está unido covalentemente con el pirofosfato de fosforribosil (PRPP) mediante la orotato fosforibisol-transferasa produciendo monofosfato de orotidina (OMP). OMP sigue con la descarboxilación por la orotidilato descarboxilasa para formar la estructura de ribonucleótido uridilato (UMP). Luego, la UMP se puede convertir en uridina-5'-trifosfato (UTP) mediante la reacción de dos quinasas. La formación de citidina-5'-trifosfato (CTP) a partir de UTP se puede lograr mediante la citidilato sintetasa mediante un intermedio de acilfosfato. [1]
Para comprender cómo surgió la vida , se requiere conocimiento de las vías químicas que permiten la formación de los componentes clave de la vida en condiciones prebióticas plausibles . Según la hipótesis del mundo del ARN, en la sopa primitiva había ribonucleótidos que flotaban libremente. Estas fueron las moléculas fundamentales que se combinaron en serie para formar ARN . Moléculas tan complejas como el ARN debieron surgir a partir de pequeñas moléculas cuya reactividad estaba regida por procesos fisicoquímicos. El ARN está compuesto de nucleótidos de purina y pirimidina , los cuales son necesarios para la transferencia confiable de información y, por lo tanto, para la selección natural y la evolución darwiniana . La síntesis de ribonucleótidos de pirimidina activados se demostró en condiciones prebióticas plausibles. [18] Los materiales de partida para la síntesis ( cianamida , cianoacetileno , glicolaldehído , gliceraldehído y fosfato inorgánico ) se consideraron moléculas de materia prima prebióticas plausibles. [18] Nam et al. [19] demostraron la condensación directa de nucleobases con ribosa para dar ribonucleósidos en microgotas acuosas, un paso clave que conduce a la formación de ARN. Además, Becker et al. presentaron un proceso prebiótico plausible para sintetizar ribonucleótidos de pirimidina y purina mediante ciclos húmedo-seco. [20]

Antes del artículo histórico de James Watson y Francis Crick que detallaba la estructura del ADN a partir de la imagen de cristalografía de rayos X de Rosalind Franklin , hubo varios científicos históricos que también contribuyeron a su descubrimiento. [21] Friedrich Miescher , un médico suizo que, en 1869, fue el primero en aislar e identificar una sustancia nucleica de los núcleos de los glóbulos blancos a la que más tarde llamó "nucleína", allanando el camino para el descubrimiento del ADN. [22] Siguiendo el trabajo de Miescher, fue el bioquímico alemán Albrecht Kossel quien, en 1878, aisló los componentes no proteicos de la "nucleína" y descubrió las cinco nucleobases presentes en los ácidos nucleicos: adenina, citosina, guanina, timina y uracilo. . [23] Aunque se conocían algunos hechos fundamentales sobre los ácidos nucleicos debido a estos primeros descubrimientos, su estructura y función seguían siendo un misterio.
No fue hasta el descubrimiento de los nucleótidos en 1919 por Phoebus Levene , un bioquímico ruso-lituano, que volvió a abrir las puertas al descubrimiento del ADN. Levene identificó por primera vez que el componente carbohidrato presente en el ARN de la levadura era en realidad ribosa . Sin embargo, no fue hasta su descubrimiento de que el componente carbohidrato del ácido nucleico del timo también era un azúcar pero carecía de un átomo de oxígeno, denominado desoxirribosa , que su descubrimiento fue ampliamente apreciado por la comunidad científica. Finalmente, Levene pudo identificar el orden correcto en el que se unen los componentes del ARN y el ADN, una unidad fosfato-azúcar-base, a la que más tarde llamó nucleótido . Aunque Levene entendía bien el orden de los componentes de los nucleótidos, la estructura de la disposición de los nucleótidos en el espacio y su código genético seguían siendo un misterio durante los primeros años de su carrera. [24]
{{cite journal}}: Mantenimiento CS1: varios nombres: lista de autores ( enlace )