El Proyecto Genoma del Chimpancé fue un esfuerzo por determinar la secuencia de ADN del genoma del chimpancé . La secuenciación comenzó en 2005 y en 2013 se habían secuenciado veinticuatro chimpancés individuales. Este proyecto se incorporó al Proyecto Genoma del Gran Simio . [1]

En 2013 se publicaron secuencias de alta resolución de cada una de las cuatro subespecies de chimpancé [2] [3] reconocidas : chimpancé central , Pan troglodytes troglodytes , 10 secuencias; Chimpancé occidental , Pan troglodytes verus , 6 secuencias; Chimpancé Nigeria-Camerún , Pan troglodytes ellioti , 4 secuencias; y chimpancé oriental , Pan troglodytes schweinfurthii , 4 secuencias. Todos fueron secuenciados con una cobertura media de 25 veces por individuo. [1]
La investigación mostró una considerable diversidad genómica en chimpancés con muchos rasgos específicos de la población. Los chimpancés centrales conservan la mayor diversidad en el linaje de chimpancés, mientras que las otras subespecies muestran signos de cuellos de botella en la población . [4]
Los cromosomas humanos y de chimpancé son muy parecidos. La principal diferencia es que los humanos tienen un par de cromosomas menos que otros grandes simios . Los humanos tienen 23 pares de cromosomas y otros grandes simios tienen 24 pares de cromosomas. En el linaje evolutivo humano, dos cromosomas de simios ancestrales se fusionaron en sus telómeros , produciendo el cromosoma 2 humano . [5] Hay otras nueve diferencias cromosómicas importantes entre chimpancés y humanos: inversiones de segmentos cromosómicos en los cromosomas humanos 1 , 4 , 5 , 9 , 12 , 15 , 16 , 17 y 18 . Tras la finalización del proyecto del genoma humano , se inició un proyecto del genoma común del chimpancé . En diciembre de 2003, un análisis preliminar de 7.600 genes compartidos entre los dos genomas confirmó que ciertos genes, como el factor de transcripción P2 forkhead-box , que participa en el desarrollo del habla, son diferentes en el linaje humano. También se descubrió que varios genes implicados en la audición habían cambiado durante la evolución humana, lo que sugiere que la selección involucra el comportamiento relacionado con el lenguaje humano . Se estima que las diferencias entre humanos individuales y chimpancés comunes son aproximadamente 10 veces la diferencia típica entre parejas de humanos. [6]
Otro estudio demostró que los patrones de metilación del ADN, que son un conocido mecanismo de regulación de la expresión genética, difieren en la corteza prefrontal de los humanos frente a los chimpancés, e implicaron esta diferencia en la divergencia evolutiva de las dos especies. [7]

Un análisis de la secuencia del genoma del chimpancé se publicó en Nature el 1 de septiembre de 2005, en un artículo elaborado por el Consorcio de Análisis y Secuenciación de Chimpancés , un grupo de científicos que cuenta con el apoyo parcial del Instituto Nacional de Investigación del Genoma Humano , uno de los Institutos Nacionales de Investigación del Genoma Humano. Institutos de Salud . El artículo marcó la finalización del borrador de la secuencia del genoma. [6]
Ahora existe una base de datos que contiene las diferencias genéticas entre los genes humanos y los de chimpancé, con alrededor de treinta y cinco millones de cambios de un solo nucleótido , cinco millones de eventos de inserción/deleción y varios reordenamientos cromosómicos . [10] Las duplicaciones de genes representan la mayoría de las diferencias de secuencia entre humanos y chimpancés. Las sustituciones de un solo par de bases representan aproximadamente la mitad del cambio genético que la duplicación de genes.
Los homólogos típicos de proteínas de humanos y chimpancés difieren sólo en un promedio de dos aminoácidos . Alrededor del 30 por ciento de todas las proteínas humanas son idénticas en secuencia a la proteína correspondiente del chimpancé. Como se mencionó anteriormente, las duplicaciones de genes son una fuente importante de diferencias entre el material genético de humanos y chimpancés, y alrededor del 2,7 por ciento del genoma ahora representa diferencias producidas por duplicaciones o eliminaciones de genes durante aproximadamente 6 millones de años [11] desde que los humanos y los chimpancés divergieron. de su ancestro evolutivo común. La variación comparable dentro de las poblaciones humanas es del 0,5 por ciento. [12]
Se identificaron alrededor de 600 genes que pueden haber estado experimentando una fuerte selección positiva en los linajes de humanos y chimpancés; muchos de estos genes están involucrados en la defensa del sistema inmunológico contra enfermedades microbianas (ejemplo: la granulisina protege contra Mycobacterium tuberculosis [13] ) o son receptores específicos de microorganismos patógenos (ejemplo: glicoforina C y Plasmodium falciparum ). Al comparar genes humanos y de chimpancé con genes de otros mamíferos, se ha descubierto que los genes que codifican factores de transcripción , como el forkhead-box P2 ( FOXP2 ), a menudo han evolucionado más rápido en el ser humano que en el chimpancé; Cambios relativamente pequeños en estos genes pueden explicar las diferencias morfológicas entre humanos y chimpancés. Un conjunto de 348 genes de factores de transcripción codifican proteínas con un promedio de aproximadamente un 50 por ciento más de cambios de aminoácidos en el linaje humano que en el linaje de chimpancé.
Se encontraron seis regiones cromosómicas humanas que pueden haber estado bajo una selección particularmente fuerte y coordinada durante los últimos 250.000 años. Estas regiones contienen al menos un alelo marcador que parece exclusivo del linaje humano, mientras que toda la región cromosómica muestra una variación genética inferior a la normal. Este patrón sugiere que uno o unos pocos genes fuertemente seleccionados en la región cromosómica pueden haber estado impidiendo la acumulación aleatoria de cambios neutrales en otros genes cercanos. Una de esas regiones en el cromosoma 7 contiene el gen FOXP2 (mencionado anteriormente) y esta región también incluye el gen regulador de la conductancia transmembrana de la fibrosis quística (CFTR), que es importante para el transporte de iones en tejidos como el epitelio secretor de sal de las glándulas sudoríparas. Se podrían seleccionar mutaciones humanas en el gen CFTR como forma de sobrevivir al cólera . [14]
Otra región de este tipo en el cromosoma 4 puede contener elementos que regulan la expresión de un gen de protocadherina cercano que puede ser importante para el desarrollo y la función del cerebro . Aunque los cambios en la expresión de genes que se expresan en el cerebro tienden a ser menores que en otros órganos (como el hígado) en promedio, los cambios en la expresión genética en el cerebro han sido más dramáticos en el linaje humano que en el linaje de los chimpancés. [15] Esto es consistente con la dramática divergencia del patrón único de desarrollo del cerebro humano observado en el linaje humano en comparación con el patrón ancestral de los grandes simios. El grupo de genes de protocadherina beta en el cromosoma 5 también muestra evidencia de una posible selección positiva. [16]
Los resultados de los análisis del genoma humano y de chimpancé deberían ayudar a comprender algunas enfermedades humanas. Los humanos parecen haber perdido un gen funcional de Caspasa 12 , que en otros primates codifica una enzima que puede proteger contra la enfermedad de Alzheimer .
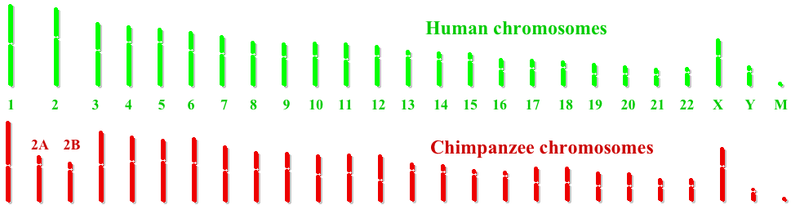

Los resultados del proyecto del genoma del chimpancé sugieren que cuando los cromosomas ancestrales 2A y 2B se fusionaron para producir el cromosoma 2 humano, no se perdió ningún gen de los extremos fusionados de 2A y 2B. En el sitio de fusión, hay aproximadamente 150.000 pares de bases de secuencia que no se encuentran en los cromosomas 2A y 2B del chimpancé. Existen copias vinculadas adicionales de los genes PGML/FOXD/CBWD en otras partes del genoma humano, particularmente cerca del extremo p del cromosoma 9 . Esto sugiere que es posible que se haya agregado una copia de estos genes al final del 2A o 2B ancestral antes del evento de fusión. Queda por determinar si estos genes insertados confieren una ventaja selectiva.