Una repetición invertida (o IR ) es una secuencia monocatenaria de nucleótidos seguida en sentido descendente por su complemento inverso . [1] La secuencia intermedia de nucleótidos entre la secuencia inicial y el complemento inverso puede tener cualquier longitud, incluido cero. Por ejemplo, 5'---TTACGnnnnnn CGTAA---3' es una secuencia repetida invertida. Cuando la longitud intermedia es cero, la secuencia compuesta es una secuencia palindrómica . [2]
Tanto las repeticiones invertidas como las repeticiones directas constituyen tipos de secuencias de nucleótidos que ocurren de forma repetitiva. Estas secuencias repetidas de ADN a menudo varían desde un par de nucleótidos hasta un gen completo , mientras que la proximidad de las secuencias repetidas varía entre conjuntos en tándem simples y ampliamente dispersos . [3] Las secuencias cortas repetidas en tándem pueden existir desde unas pocas copias en una pequeña región hasta miles de copias dispersas por todo el genoma de la mayoría de los eucariotas . [4] Las secuencias repetidas con alrededor de 10 a 100 pares de bases se conocen como minisatélites , mientras que las secuencias repetidas más cortas que tienen principalmente de 2 a 4 pares de bases se conocen como microsatélites . [5] Las repeticiones más comunes incluyen las repeticiones de dinucleótidos, que tienen las bases AC en una cadena de ADN y GT en la cadena complementaria. [3] Algunos elementos del genoma con secuencias únicas funcionan como exones , intrones y ADN regulador. [6] Aunque los loci más familiares de las secuencias repetitivas son el centrómero y el telómero , [6] una gran parte de las secuencias repetidas en el genoma se encuentran entre el ADN no codificante . [5]
Las repeticiones invertidas tienen una serie de funciones biológicas importantes. Definen los límites en los transposones e indican regiones capaces de emparejamiento de bases autocomplementarias (regiones dentro de una única secuencia que pueden emparejarse entre sí). Estas propiedades desempeñan un papel importante en la inestabilidad del genoma [7] y contribuyen no sólo a la evolución celular y la diversidad genética [8] sino también a las mutaciones y las enfermedades . [9] Para estudiar estos efectos en detalle, se han desarrollado varios programas y bases de datos para ayudar en el descubrimiento y anotación de repeticiones invertidas en varios genomas.
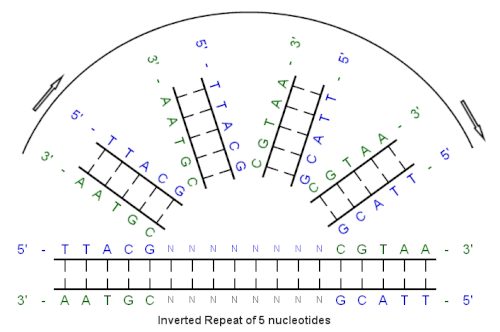
Comenzando con esta secuencia inicial: 5'-TTACG-3'
El complemento creado por el emparejamiento de bases es: 3'-AATGC-5'
El complemento inverso es: 5'-CGTAA-3'
Y la secuencia repetida invertida es: 5'---TTACGnnnnnn CGTAA---3'
"nnnnnn" representa cualquier número de nucleótidos intermedios.
Una repetición directa ocurre cuando una secuencia se repite con el mismo patrón en sentido descendente. [1] No hay inversión ni complemento inverso asociados con una repetición directa. La secuencia de nucleótidos escrita en negrita significa la secuencia repetida. Puede tener o no nucleótidos intermedios.
Lingüísticamente, una repetición directa típica es comparable a la rima, como en "time on ad ime ".
Una repetición directa sin nucleótidos intermedios entre la secuencia inicial y su copia posterior es una repetición en tándem . La secuencia de nucleótidos escrita en negrita significa la secuencia repetida.
Lingüísticamente, una repetición típica en tándem es comparable a la tartamudez o a palabras repetidas deliberadamente, como en "adiós".
Una secuencia repetida invertida sin nucleótidos intermedios entre la secuencia inicial y su complemento inverso aguas abajo es un palíndromo . [1]
EJEMPLO:
Paso 1: comenzar con una repetición invertida: 5' TTACGnnnnnnCGTAA 3'
Paso 2: eliminar los nucleótidos intermedios: 5' TTACGCGTAA 3'
Esta secuencia resultante es palindrómica porque es el complemento inverso de sí misma. [1]
Las diversas repeticiones de todo el genoma se derivan de elementos transponibles , que ahora se entiende que "salta" entre diferentes ubicaciones genómicas, sin transferir sus copias originales. [10] El traslado posterior de las mismas secuencias a lo largo de numerosas generaciones garantiza su multiplicidad en todo el genoma. [10] La recombinación limitada de las secuencias entre dos elementos de secuencia distintos conocida como recombinación conservadora específica de sitio (CSSR) da como resultado inversiones del segmento de ADN, basadas en la disposición de las secuencias de reconocimiento de recombinación en el ADN del donante y el ADN del receptor. [10] Nuevamente, la orientación de dos de los sitios de recombinación dentro de la molécula de ADN donante en relación con la asimetría de las secuencias de escisión del ADN interpuestas, conocida como región de cruce, es fundamental para la formación de repeticiones invertidas o repeticiones directas. [10] Por lo tanto, la recombinación que ocurre en un par de sitios invertidos invertirá la secuencia de ADN entre los dos sitios. [10] Se han observado cromosomas muy estables con un número comparativamente menor de repeticiones invertidas que de repeticiones directas, lo que sugiere una relación entre la estabilidad cromosómica y el número de repeticiones. [11]
Se han observado repeticiones terminales invertidas en el ADN de varios transposones eucariotas, aunque su origen sigue siendo desconocido. [12] Las repeticiones invertidas se encuentran principalmente en los orígenes de replicación de organismos celulares y orgánulos que van desde plásmidos de fagos, mitocondrias y virus eucariotas hasta células de mamíferos. [13] Los orígenes de replicación del fago G4 y otros fagos relacionados comprenden un segmento de casi 139 bases de nucleótidos que incluyen tres repeticiones invertidas que son esenciales para el cebado de la replicación. [13]
En gran medida, las porciones de repeticiones de nucleótidos se observan con bastante frecuencia como parte de combinaciones raras de ADN. [14] Las tres repeticiones principales que se encuentran en gran medida en construcciones de ADN particulares incluyen las repeticiones invertidas de homopurina-homopirimidina, muy precisas, que también se conocen como palíndromos H, una ocurrencia común en conformaciones H de triple hélice que pueden comprender TAT o CGC. tríadas de nucleótidos. Los otros podrían describirse como repeticiones invertidas largas que tienen tendencia a producir horquillas y cruciformes, y finalmente repeticiones directas en tándem, que existen comúnmente en estructuras descritas como ADN-Z de bucle deslizado, cruciforme y zurdo. [14]
Estudios anteriores sugieren que las repeticiones son una característica común de los eucariotas, a diferencia de los procariotas y las arqueas . [14] Otros informes sugieren que, independientemente de la escasez comparativa de elementos repetidos en los genomas procarióticos, contienen cientos o incluso miles de repeticiones grandes. [15] Los análisis genómicos actuales parecen sugerir la existencia de un gran exceso de repeticiones invertidas perfectas en muchos genomas procarióticos en comparación con los genomas eucariotas. [dieciséis]

Para la cuantificación y comparación de repeticiones invertidas entre varias especies, concretamente en arqueas, consulte [17]
Los pseudonudos son motivos estructurales comunes que se encuentran en el ARN. Están formados por dos bucles de vástago anidados de modo que el vástago de una estructura se forma a partir del bucle de la otra. Existen múltiples topologías de plegado entre los pseudonudos y una gran variación en las longitudes de los bucles, lo que los convierte en un grupo estructuralmente diverso. [18]
Las repeticiones invertidas son un componente clave de los pseudonudos, como se puede ver en la ilustración de un pseudonudo natural que se encuentra en el componente de ARN de la telomerasa humana . [19] En esta estructura intervienen cuatro conjuntos diferentes de repeticiones invertidas. Los conjuntos 1 y 2 son el vástago del bucle de vástago A y son parte del bucle del bucle de vástago B. De manera similar, los conjuntos 3 y 4 son el vástago del bucle de vástago B y son parte del bucle del bucle de vástago A.
Los pseudonudos desempeñan diferentes funciones en biología. El pseudonudo de telomerasa en la ilustración es fundamental para la actividad de esa enzima. [19] La ribozima del virus de la hepatitis delta (VHD) se pliega en una estructura de pseudonudo doble y autoescinde su genoma circular para producir un ARN de longitud de genoma único. Los pseudonudos también desempeñan un papel en el cambio de marco ribosómico programado que se encuentra en algunos virus y es necesario en la replicación de los retrovirus . [18]
Las repeticiones invertidas juegan un papel importante en los riboswitches , que son elementos reguladores del ARN que controlan la expresión de los genes que producen el ARNm, del que forman parte. [10] En la ilustración se muestra un ejemplo simplificado del riboswitch del mononucleótido de flavina (FMN). Este riboswitch existe en la transcripción del ARNm y tiene varias estructuras de tallo-bucle aguas arriba de la región codificante . Sin embargo, en la ilustración sólo se muestran los bucles de vástago clave, que se han simplificado enormemente para ayudar a mostrar el papel de las repeticiones invertidas. Hay múltiples repeticiones invertidas en este riboswitch como se indica en verde (fondo amarillo) y azul (fondo naranja).
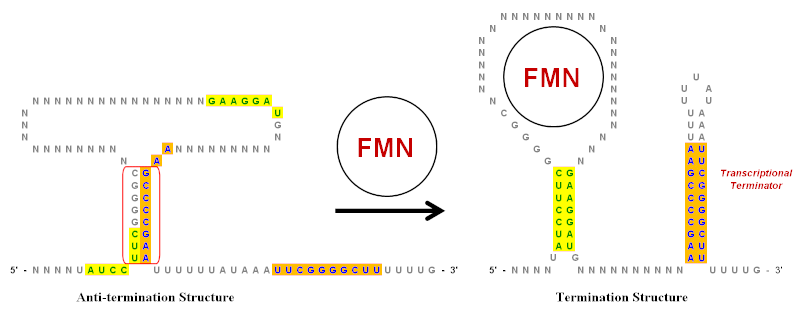
En ausencia de FMN, la estructura anti-terminación es la conformación preferida para la transcripción de ARNm. Se crea mediante el emparejamiento de bases de la región de repetición invertida rodeada de un círculo rojo. Cuando FMN está presente, puede unirse al bucle e impedir la formación de la estructura anti-terminación. Esto permite que dos conjuntos diferentes de repeticiones invertidas formen pares de bases y formen la estructura de terminación. [20] El bucle del tallo en el extremo 3' es un terminador transcripcional porque la secuencia que lo sigue inmediatamente es una cadena de uracilos (U). Si se forma este bucle (debido a la presencia de FMN) a medida que la cadena de ARN en crecimiento emerge del complejo de ARN polimerasa , creará suficiente tensión estructural para hacer que la cadena de ARN se disocia y, por lo tanto, termine la transcripción. La disociación ocurre fácilmente porque el emparejamiento de bases entre las U en el ARN y las A en la cadena plantilla son los más débiles de todos los pares de bases. [10] Por lo tanto, a niveles de concentración más altos, FMN regula negativamente su propia transcripción al aumentar la formación de la estructura de terminación.
Las repeticiones invertidas a menudo se describen como "puntos críticos" de inestabilidad genómica eucariota y procariótica. [7] Se considera que las repeticiones invertidas largas influyen en gran medida en la estabilidad del genoma de varios organismos. [21] Esto se ejemplifica en E. coli , donde las secuencias genómicas con repeticiones invertidas largas rara vez se replican, sino que se eliminan con rapidez. [21] Nuevamente, las largas repeticiones invertidas observadas en la levadura favorecen en gran medida la recombinación dentro del mismo cromosoma y los adyacentes, lo que resulta en una tasa igualmente muy alta de deleción. [21] Finalmente, también se observó una tasa muy alta de deleción y recombinación en regiones de cromosomas de mamíferos con repeticiones invertidas. [21] Las diferencias reportadas en la estabilidad de los genomas de organismos interrelacionados son siempre una indicación de una disparidad en las repeticiones invertidas. [11] La inestabilidad resulta de la tendencia de las repeticiones invertidas a plegarse en estructuras de ADN en forma de horquilla o cruciformes. Estas estructuras especiales pueden obstaculizar o confundir la replicación del ADN y otras actividades genómicas. [7] Por lo tanto, las repeticiones invertidas conducen a configuraciones especiales tanto en el ARN como en el ADN que, en última instancia, pueden causar mutaciones y enfermedades . [9]

La ilustración muestra una repetición invertida sometida a extrusión cruciforme. El ADN en la región de la repetición invertida se desenrolla y luego se recombina, formando una unión de cuatro vías con dos estructuras de tallo-bucle . La estructura cruciforme se produce porque las secuencias repetidas invertidas se autoemparejan entre sí en su propia hebra. [22]
Los cruciformes extruidos pueden provocar mutaciones por cambio de marco cuando una secuencia de ADN tiene repeticiones invertidas en forma de palíndromo combinadas con regiones de repeticiones directas en ambos lados. Durante la transcripción , el deslizamiento y la disociación parcial de la polimerasa de la cadena plantilla pueden provocar mutaciones tanto de deleción como de inserción . [9] La eliminación ocurre cuando una porción de la hebra plantilla desenrollada forma un bucle que la maquinaria de transcripción "salta". La inserción ocurre cuando se forma un bucle de tallo en una porción disociada de la hebra naciente (recién sintetizada), lo que provoca que una porción de la hebra plantilla se transcriba dos veces. [9]
Las repeticiones invertidas imperfectas pueden provocar mutaciones mediante cambios intra e intercatenarios. [9] La región codificante del gen de antitrombina III es un ejemplo de una repetición invertida imperfecta, como se muestra en la figura de la derecha. La estructura de vástago-bucle se forma con un bulto en la parte inferior porque la G y la T no se emparejan. Un evento de cambio de hebra podría dar como resultado que la G (en la protuberancia) sea reemplazada por una A que elimina la "imperfección" en la repetición invertida y proporciona una estructura de vástago-bucle más fuerte. Sin embargo, el reemplazo también crea una mutación puntual que convierte el codón GCA en ACA. Si al evento de cambio de cadena le sigue una segunda ronda de replicación del ADN , la mutación puede quedar fijada en el genoma y provocar una enfermedad. En concreto, la mutación sin sentido conduciría a un gen defectuoso y a una deficiencia de antitrombina que podría dar lugar al desarrollo de tromboembolismo venoso (coágulos de sangre dentro de una vena). [9]
Las mutaciones en el gen del colágeno pueden provocar la enfermedad Osteogénesis Imperfecta , que se caracteriza por huesos frágiles. [9] En la ilustración, un tallo-bucle formado a partir de una repetición invertida imperfecta se muta con una inserción de nucleótido de timina (T) como resultado de un cambio entre o intracadenas. La adición de la T crea una "coincidencia" de emparejamiento de bases con la adenina (A) que anteriormente era una "protuberancia" en el lado izquierdo del tallo. Si bien esta adición fortalece el tallo y perfecciona la repetición invertida, también crea una mutación por cambio de marco en la secuencia de nucleótidos que altera el marco de lectura y dará como resultado una expresión incorrecta del gen. [9]
La siguiente lista proporciona información y enlaces externos a varios programas y bases de datos para repeticiones invertidas:
{{cite book}}: Mantenimiento CS1: varios nombres: lista de autores ( enlace )