En bioquímica , las isoenzimas (también conocidas como isoenzimas o, de manera más general, como formas múltiples de enzimas ) son enzimas que difieren en la secuencia de aminoácidos pero catalizan la misma reacción química. Las isoenzimas suelen tener diferentes parámetros cinéticos (por ejemplo, diferentes valores de K M ) o se regulan de manera diferente. Permiten el ajuste fino del metabolismo para satisfacer las necesidades particulares de un tejido o una etapa de desarrollo determinados.
En muchos casos, las isoenzimas están codificadas por genes homólogos que han divergido con el tiempo. En sentido estricto, las enzimas con diferentes secuencias de aminoácidos que catalizan la misma reacción son isoenzimas si están codificadas por genes diferentes, o alozimas si están codificadas por alelos diferentes del mismo gen ; los dos términos se usan a menudo indistintamente.
Las isoenzimas fueron descritas por primera vez por RL Hunter y Clement Markert (1957), quienes las definieron como diferentes variantes de la misma enzima que tienen funciones idénticas y están presentes en el mismo individuo . [1] Esta definición abarca (1) variantes enzimáticas que son el producto de diferentes genes y, por lo tanto, representan diferentes loci (descritas como isoenzimas ) y (2) enzimas que son el producto de diferentes alelos del mismo gen (descritas como alozimas ). [2]
Las isoenzimas suelen ser el resultado de la duplicación de genes , pero también pueden surgir de la poliploidización o la hibridación de ácidos nucleicos . A lo largo del tiempo evolutivo, si la función de la nueva variante permanece idéntica a la original, es probable que una u otra se pierda a medida que se acumulan las mutaciones , lo que da lugar a un pseudogén . Sin embargo, si las mutaciones no impiden inmediatamente el funcionamiento de la enzima, sino que modifican su función o su patrón de expresión , entonces las dos variantes pueden verse favorecidas por la selección natural y especializarse en diferentes funciones. [3] Por ejemplo, pueden expresarse en diferentes etapas del desarrollo o en diferentes tejidos. [4]
Las alozimas pueden ser resultado de mutaciones puntuales o de eventos de inserción-deleción ( indel ) que afectan la secuencia codificante del gen. Al igual que con cualquier otra mutación nueva, hay tres cosas que pueden sucederle a una nueva alozima:
Un ejemplo de isoenzima es la glucoquinasa , una variante de la hexoquinasa que no es inhibida por la glucosa 6-fosfato . Sus diferentes características reguladoras y su menor afinidad por la glucosa (en comparación con otras hexoquinasas) le permiten cumplir diferentes funciones en células de órganos específicos, como el control de la liberación de insulina por las células beta del páncreas o el inicio de la síntesis de glucógeno por las células hepáticas . Ambos procesos deben ocurrir solo cuando la glucosa es abundante.
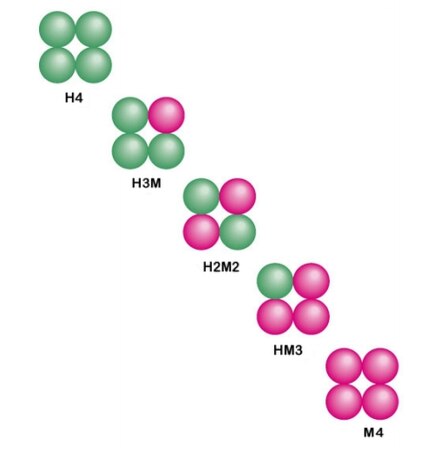

1.) La enzima lactato deshidrogenasa es un tetrámero formado por dos subunidades diferentes, la forma H y la forma M. Estas se combinan en diferentes combinaciones según el tejido: [7]
2.) Isoenzimas de la creatina fosfoquinasa: [7] La creatina quinasa (CK) o creatina fosfoquinasa (CPK) cataliza la interconversión de fosfocreatina a creatina.
La CPK existe en tres isoenzimas. Cada isoenzima es un dímero de dos subunidades: M (músculo), B (cerebro) o ambas [7]
3.) Isoenzimas de la fosfatasa alcalina: [7] Se han identificado seis isoenzimas. La enzima es un monómero, las isoenzimas se deben a las diferencias en el contenido de carbohidratos (residuos de ácido siálico). Las isoenzimas de ALP más importantes son α 1 -ALP, α 2 -ALP termolábil, α 2 -ALP termoestable, pre-β ALP y γ-ALP. El aumento de α 2 -ALP termolábil sugiere hepatitis mientras que pre-β ALP indica enfermedades óseas.
Las isozimas (y las alozimas) son variantes de la misma enzima. A menos que sean idénticas en sus propiedades bioquímicas, por ejemplo sus sustratos y cinética enzimática , se pueden distinguir mediante un ensayo bioquímico . Sin embargo, dichas diferencias suelen ser sutiles, en particular entre las alozimas , que a menudo son variantes neutras . Esta sutileza es de esperar, porque es poco probable que dos enzimas que difieren significativamente en su función hayan sido identificadas como isozimas .
Si bien las isoenzimas pueden ser casi idénticas en su función, pueden diferir en otros aspectos. En particular, las sustituciones de aminoácidos que cambian la carga eléctrica de la enzima son fáciles de identificar mediante electroforesis en gel , y esto constituye la base para el uso de isoenzimas como marcadores moleculares . Para identificar las isoenzimas, se elabora un extracto de proteína cruda moliendo tejido animal o vegetal con un tampón de extracción, y los componentes del extracto se separan según su carga mediante electroforesis en gel. Históricamente, esto se ha hecho generalmente utilizando geles hechos de almidón de patata , pero los geles de acrilamida proporcionan una mejor resolución.
Todas las proteínas del tejido están presentes en el gel, por lo que las enzimas individuales deben identificarse mediante un ensayo que vincule su función con una reacción de tinción. Por ejemplo, la detección puede basarse en la precipitación localizada de colorantes indicadores solubles , como las sales de tetrazolio , que se vuelven insolubles cuando se reducen por cofactores como NAD o NADP , que se generan en zonas de actividad enzimática. Este método de ensayo requiere que las enzimas sigan siendo funcionales después de la separación ( electroforesis en gel nativa ) y supone el mayor desafío para el uso de isoenzimas como técnica de laboratorio.
Las isoenzimas difieren en cinética (tienen diferentes valores de K M y V max ).
La genética de poblaciones es esencialmente un estudio de las causas y efectos de la variación genética dentro y entre poblaciones, y en el pasado, las isoenzimas han estado entre los marcadores moleculares más utilizados para este propósito. Aunque ahora han sido ampliamente reemplazadas por enfoques basados en ADN más informativos (como la secuenciación directa de ADN , polimorfismos de un solo nucleótido y microsatélites ), todavía están entre los sistemas de marcadores más rápidos y económicos de desarrollar, y siguen siendo (a partir de 2005 [actualizar]) una excelente opción para proyectos que solo necesitan identificar niveles bajos de variación genética, por ejemplo, cuantificar los sistemas de apareamiento .