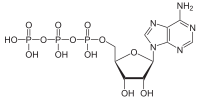
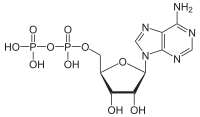
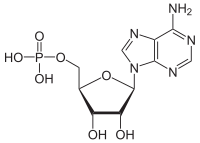
Las ATPasas ( EC 3.6.1.3, A denosina 5'- Trifosfatasa , adenilpirofosfatasa, ATP monofosfatasa, trifosfatasa, antígeno T SV40, ATP hidrolasa, complejo V (transporte de electrones mitocondrial), (Ca 2+ + Mg 2+ )-ATPasa, HCO 3 − -ATPasa , adenosina trifosfatasa) son una clase de enzimas que catalizan la descomposición de ATP en ADP y un ion fosfato libre [1] [2] [ 3] [4] [5] [6] o la reacción inversa. Esta reacción de desfosforilación libera energía , que la enzima (en la mayoría de los casos) aprovecha para impulsar otras reacciones químicas que de otra manera no ocurrirían. Este proceso se usa ampliamente en todas las formas de vida conocidas .
Algunas de estas enzimas son proteínas integrales de membrana (ancladas dentro de las membranas biológicas ) y mueven solutos a través de la membrana, generalmente en contra de su gradiente de concentración. Estas enzimas se denominan ATPasas transmembrana.

Las ATPasas transmembrana importan metabolitos necesarios para el metabolismo celular y exportan toxinas, desechos y solutos que pueden obstaculizar los procesos celulares. Un ejemplo importante es la bomba de sodio-potasio (Na + /K + ATPasa) que mantiene el potencial de membrana celular . Otro ejemplo es la ATPasa de hidrógeno y potasio (H + /K + ATPasa o bomba de protones gástrica) que acidifica el contenido del estómago. La ATPasa se conserva genéticamente en los animales; por lo tanto, los cardenólidos , que son esteroides tóxicos producidos por plantas que actúan sobre las ATPasas, forman toxinas animales generales y efectivas que actúan de manera dependiente de la dosis. [7]
Además de los intercambiadores, otras categorías de ATPasa transmembrana incluyen cotransportadores y bombas (sin embargo, algunos intercambiadores también son bombas). Algunos de estos, como la ATPasa Na + /K + , causan un flujo neto de carga, pero otros no. Estos se denominan transportadores electrogénicos y transportadores electroneutrales, respectivamente. [8]
Los motivos Walker son un motivo de secuencia de proteínas revelador de la unión de nucleótidos y la hidrólisis. Más allá de esta amplia función, los motivos Walker se pueden encontrar en casi todas las ATPasas naturales, con la notable excepción de las tirosina quinasas . [9] Los motivos Walker forman comúnmente una hélice alfa de hoja Beta -giro- que se autoorganiza como un Nido (motivo estructural de proteína) . Se cree que esto se debe a que las ATPasas modernas evolucionaron a partir de pequeños péptidos de unión a NTP que tuvieron que autoorganizarse. [10]
El diseño de proteínas ha sido capaz de replicar la función de la ATPasa (débilmente) sin utilizar secuencias o estructuras de ATPasa naturales. Es importante destacar que, si bien todas las ATPasas naturales tienen cierta estructura de lámina beta, la "ATPasa alternativa" diseñada carece de estructura de lámina beta, lo que demuestra que esta función esencial para la vida es posible con secuencias y estructuras que no se encuentran en la naturaleza. [11]
La ATPasa (también llamada F 0 F 1 -ATP sintasa) es un complejo de transferencia de carga que cataliza el ATP para realizar la síntesis de ATP moviendo iones a través de la membrana. [12]
El acoplamiento de la hidrólisis y el transporte de ATP es una reacción química en la que se transporta un número fijo de moléculas de soluto por cada molécula de ATP hidrolizada; para el intercambiador Na + /K + , esto es tres iones Na + fuera de la célula y dos iones K+ dentro por cada molécula de ATP hidrolizada.
Las ATPasas transmembrana utilizan la energía potencial química del ATP realizando trabajo mecánico: transportan solutos en la dirección opuesta a su dirección de movimiento preferida termodinámicamente, es decir, desde el lado de la membrana con baja concentración al lado con alta concentración. Este proceso se conoce como transporte activo .
Por ejemplo, la inhibición de las H + -ATPasas vesiculares daría lugar a un aumento del pH dentro de las vesículas y una caída del pH del citoplasma.
Todas las ATPasas comparten una estructura básica común. Cada ATPasa rotatoria está compuesta por dos componentes principales: F 0 /A 0 /V 0 y F 1 /A 1 /V 1 . Están conectados por 1-3 tallos para mantener la estabilidad, controlar la rotación y evitar que giren en la otra dirección. Un tallo se utiliza para transmitir el par. [13] La cantidad de tallos periféricos depende del tipo de ATPasa: las F-ATPasas tienen uno, las A-ATPasas tienen dos y las V-ATPasas tienen tres. El dominio catalítico F 1 se encuentra en el lado N de la membrana y está involucrado en la síntesis y degradación de ATP y está involucrado en la fosforilación oxidativa . El dominio transmembrana F 0 está involucrado en el movimiento de iones a través de la membrana. [12]
La F 0 F 1 -ATPasa bacteriana consta del dominio soluble F 1 y del dominio transmembrana F 0 , que está compuesto de varias subunidades con estequiometría variable. Hay dos subunidades, γ y ε, que forman el tallo central y están unidas a F 0 . F 0 contiene un oligómero de subunidad c en forma de anillo (anillo c). La subunidad α está cerca de la subunidad b 2 y forma el tallo que conecta las subunidades transmembrana a las subunidades α3β3 y δ. Las F-ATP sintasas son idénticas en apariencia y función, excepto por la F 0 F 1 -ATP sintasa mitocondrial, que contiene 7-9 subunidades adicionales. [12]
El potencial electroquímico es lo que hace que el anillo c rote en el sentido de las agujas del reloj para la síntesis de ATP. Esto hace que el tallo central y el dominio catalítico cambien de forma. La rotación del anillo c hace que se formen tres moléculas de ATP, lo que a su vez hace que el H + se mueva del lado P de la membrana al lado N de la misma. La rotación en sentido contrario a las agujas del reloj del anillo c es impulsada por la hidrólisis del ATP y los iones se mueven del lado N al lado P, lo que ayuda a generar potencial electroquímico. [12]
La ATP sintasa de las mitocondrias y los cloroplastos es una enzima anabólica que aprovecha la energía de un gradiente transmembrana de protones como fuente de energía para agregar un grupo fosfato inorgánico a una molécula de difosfato de adenosina (ADP) para formar una molécula de trifosfato de adenosina (ATP).
Esta enzima funciona cuando un protón se mueve a favor del gradiente de concentración, lo que le da a la enzima un movimiento giratorio. Este movimiento giratorio único une el ADP y el P para crear ATP.
La ATP sintasa también puede funcionar a la inversa, es decir, utilizar la energía liberada por la hidrólisis del ATP para bombear protones contra su gradiente electroquímico.
Existen diferentes tipos de ATPasas, que pueden diferir en función (síntesis y/o hidrólisis de ATP), estructura (las ATPasas F, V y A contienen motores rotatorios) y en el tipo de iones que transportan.
Las P-ATPasas (conocidas también como E1-E2) se encuentran en bacterias y también en las membranas plasmáticas y organelos eucariotas. Su nombre se debe a la breve unión del fosfato inorgánico a los residuos de aspartato en el momento de la activación. La función de la P-ATPasa es transportar una variedad de compuestos diferentes, como iones y fosfolípidos, a través de una membrana utilizando la hidrólisis de ATP para obtener energía. Existen muchas clases diferentes de P-ATPasas, que transportan un tipo específico de ion. Las P-ATPasas pueden estar compuestas por uno o dos polipéptidos y, por lo general, pueden adoptar dos conformaciones principales, E1 y E2.