La edición primaria es una tecnología de edición genómica de "búsqueda y reemplazo" en biología molecular mediante la cual se puede modificar el genoma de organismos vivos. La tecnología escribe directamente nueva información genética en un sitio de ADN objetivo. Utiliza una proteína de fusión , que consiste en una endonucleasa Cas9 catalíticamente alterada fusionada a una enzima transcriptasa inversa diseñada , y un ARN guía de edición primaria (pegRNA), capaz de identificar el sitio objetivo y proporcionar la nueva información genética para reemplazar los nucleótidos de ADN objetivo. Media inserciones , deleciones y conversiones de base a base dirigidas sin la necesidad de roturas de doble cadena (DSB) o plantillas de ADN donante. [1]
La tecnología ha recibido atención de la prensa generalista debido a sus posibles usos en genética médica. Utiliza metodologías similares a las tecnologías de edición de genomas precursores, incluidos CRISPR/Cas9 y editores de bases . La edición primaria se ha utilizado en algunos modelos animales de enfermedades genéticas [2] [3] [4] y plantas. [5]
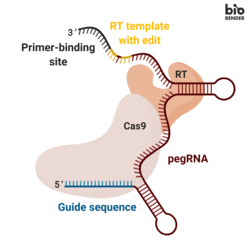
La edición principal implica tres componentes principales: [1]
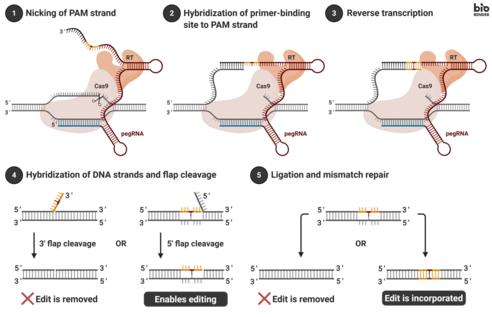
La edición genómica se lleva a cabo mediante la transfección de células con el pegRNA y la proteína de fusión. La transfección se suele llevar a cabo introduciendo vectores en una célula. Una vez internalizada, la proteína de fusión corta la secuencia de ADN diana, exponiendo un grupo hidroxilo 3' que se puede utilizar para iniciar (preparar) la transcripción inversa de la porción de plantilla RT del pegRNA. Esto da como resultado un intermedio ramificado que contiene dos solapas de ADN: una solapa 3' que contiene la secuencia recién sintetizada (editada) y una solapa 5' que contiene la secuencia de ADN prescindible, sin editar. La solapa 5' se escinde luego mediante endonucleasas específicas de la estructura o exonucleasas 5' . Este proceso permite la ligadura de la solapa 3' y crea un ADN heterodúplex compuesto por una hebra editada y una hebra sin editar. El ADN bicatenario rehibridado contiene desajustes de nucleótidos en el lugar donde se realizó la edición. Para corregir los desajustes, las células explotan el mecanismo intrínseco de reparación de desajustes (MMR), con dos resultados posibles: (i) la información de la cadena editada se copia en la cadena complementaria, instalando permanentemente la edición; (ii) los nucleótidos originales se reincorporan a la cadena editada, excluyendo la edición. [1]
Durante el desarrollo de esta tecnología se realizaron varias modificaciones a los componentes, con el fin de aumentar su efectividad. [1]
En el primer sistema, se fusionó una transcriptasa inversa del virus de la leucemia murina de Moloney (M-MLV) de tipo salvaje con el extremo C de la nickasa Cas9 H840A. Se observaron eficiencias de edición detectables. [1]
Para mejorar la afinidad ADN-ARN, la procesividad enzimática y la termoestabilidad, se incorporaron cinco sustituciones de aminoácidos en la transcriptasa inversa M-MLV. Luego, la RT M-MLV mutante se incorporó a PE1 para dar lugar a (Cas9 (H840A)-M-MLV RT(D200N/L603W/T330P/T306K/W313F)). Se observó una mejora en la eficiencia con respecto a PE1. [1]

A pesar de su mayor eficacia, la edición insertada por PE2 aún podría eliminarse debido a la reparación de desajustes de ADN de la cadena editada. Para evitar este problema durante la resolución del heterodúplex de ADN, se introduce un ARN guía único adicional (sgRNA). Este sgRNA está diseñado para coincidir con la secuencia editada introducida por el pegRNA, pero no con el alelo original. Dirige a la porción de nickasa Cas9 de la proteína de fusión para que corte la cadena no editada en un sitio cercano, opuesto al corte original. Cortar la cadena no editada hace que el sistema de reparación natural de la célula copie la información de la cadena editada en la cadena complementaria, instalando permanentemente la edición. [1] Sin embargo, este sistema tiene desventajas, ya que cortar la cadena inalterada puede conducir a indeles no deseados adicionales . [9]
El editor principal 4 utiliza la misma maquinaria que PE2, pero también incluye un plásmido que codifica la proteína MMR dominante negativa MLH1 . La MLH1 dominante negativa es capaz de eliminar esencialmente la MLH1 endógena por inhibición, reduciendo así la respuesta MMR celular y aumentando la eficiencia de la edición principal. [9]
El editor principal 5 utiliza la misma maquinaria que el PE3, pero también incluye un plásmido que codifica el gen MLH1 negativo dominante. Al igual que el PE4, esto permite una reducción de la respuesta MMR endógena, lo que aumenta la eficiencia de la edición principal. [9]
Nuclease Prime Editor utiliza la nucleasa Cas9 en lugar de la nickasa Cas9(H840A). A diferencia de Prime Editor 3 (PE3), que requiere una doble muesca en ambas cadenas de ADN para inducir una edición de primera eficiente, Nuclease Prime Editor requiere solo un único pegRNA, ya que el único gRNA ya crea una rotura de doble cadena en lugar de una muesca de cadena simple. [10]
El mecanismo de "edición de doble prima" (twinPE) presentado en 2021 permite editar grandes secuencias de ADN (secuencias tan grandes como genes), lo que soluciona el inconveniente clave del método. Utiliza una proteína editora de prima y dos ARN guía de edición de prima. [11] [12] [ se necesitan más detalles ]
La edición de primera calidad se desarrolló en el laboratorio de David R. Liu en el Broad Institute y se dio a conocer en Anzalone et al. (2019). [13] Desde entonces, la edición de primera calidad y la investigación que la produjo han recibido un amplio reconocimiento científico, [14] [6] [15] siendo calificada de "revolucionaria" [7] y una parte importante del futuro de la edición. [13]
La eficiencia de la edición primaria se puede aumentar con el uso de pegRNA modificados genéticamente (epegRNA). Un problema común con los pegRNA tradicionales es la degradación del extremo 3', lo que conduce a una disminución de la eficiencia de la edición primaria. Los epegRNA tienen un motivo de ARN estructurado agregado a su extremo 3' para evitar la degradación. [16]
Aunque se necesita más investigación para mejorar la eficiencia de la edición primaria, la tecnología ofrece mejoras científicas prometedoras con respecto a otras herramientas de edición genética. La tecnología de edición primaria tiene el potencial de corregir la gran mayoría de alelos patógenos que causan enfermedades genéticas, ya que puede reparar inserciones, deleciones y sustituciones de nucleótidos. [1]
La herramienta de edición primaria ofrece ventajas sobre las tecnologías tradicionales de edición genética. Las ediciones CRISPR/Cas9 se basan en la unión de extremos no homólogos (NHEJ) o la reparación dirigida por homología (HDR) para reparar las roturas del ADN, mientras que el sistema de edición primaria emplea la reparación de desajustes del ADN . Esta es una característica importante de esta tecnología dado que los mecanismos de reparación del ADN como NHEJ y HDR generan inserciones o deleciones aleatorias no deseadas (INDEL). Estos son subproductos que complican la recuperación de células que llevan la edición correcta. [1] [17]
El sistema de edición de bases introduce cortes de ADN monocatenario en lugar de los cortes de ADN bicatenario observados en otras herramientas de edición, como los editores de bases. En conjunto, la edición de bases y la edición de corte ofrecen fortalezas y debilidades complementarias para realizar mutaciones de transición dirigidas. Los editores de bases ofrecen una mayor eficiencia de edición y menos subproductos de INDEL si la edición deseada es una mutación de punto de transición y existe una secuencia PAM aproximadamente a 15 bases del sitio objetivo. Sin embargo, debido a que la tecnología de edición de corte no requiere una secuencia PAM posicionada con precisión para apuntar a una secuencia de nucleótidos, ofrece más flexibilidad y precisión de edición. Sorprendentemente, los editores de corte permiten insertar todo tipo de sustituciones, transiciones y transversiones en la secuencia objetivo. [1] [17] La edición de bases de citosina y la BE de adenina ya pueden realizar transiciones de bases precisas , pero para las transversiones de bases no ha habido buenas opciones. La edición de corte realiza transversiones con buena usabilidad. La PE puede insertar hasta 44 pb, eliminar hasta 80 o combinaciones de las mismas. [7]
Debido a que el sistema principal involucra tres eventos de unión de ADN separados (entre (i) la secuencia guía y el ADN objetivo, (ii) el sitio de unión del cebador y el ADN objetivo, y (iii) el extremo 3' de la cadena de ADN mellada y el pegRNA), se ha sugerido que tiene menos efectos no deseados fuera del objetivo que CRISPR/Cas9 . [1] [17]
Existe un interés considerable en aplicar métodos de edición genética al tratamiento de enfermedades con un componente genético. Sin embargo, existen múltiples desafíos asociados con este enfoque. Un tratamiento efectivo requeriría la edición de un gran número de células diana, lo que a su vez requeriría un método de administración eficaz y un gran nivel de especificidad tisular. [1] [18]
A partir de 2019, la edición primaria parece prometedora para alteraciones genéticas relativamente pequeñas, pero se necesita realizar más investigaciones para evaluar si la tecnología es eficiente para realizar alteraciones más grandes, como inserciones y deleciones dirigidas. Las alteraciones genéticas más grandes requerirían una plantilla RT más larga, lo que podría dificultar la entrega eficiente de pegRNA a las células objetivo. Además, un pegRNA que contenga una plantilla RT larga podría volverse vulnerable al daño causado por las enzimas celulares. [1] [18] La edición primaria en plantas sufre de baja eficiencia que va desde cero a un pequeño porcentaje y necesita una mejora significativa. [19]
Algunas de estas limitaciones se han mitigado con mejoras recientes en los editores principales, [2] [20] incluidos motivos que protegen a los pegRNA de la degradación. [21] Se necesita más investigación antes de que la edición principal se pueda utilizar para corregir alelos patógenos en humanos. [1] [18] La investigación también ha demostrado que la inhibición de ciertas proteínas MMR , incluida MLH1, puede mejorar la eficiencia de la edición principal. [9]
Los editores de bases utilizados para la edición primaria requieren la administración de una proteína y una molécula de ARN en células vivas. Introducir tecnologías de edición genética exógena en organismos vivos es un desafío significativo. Una forma potencial de introducir un editor de bases en animales y plantas es empaquetar el editor de bases en una cápside viral. El organismo objetivo puede luego ser transducido por el virus para sintetizar el editor de bases in vivo . Los vectores de transducción de laboratorio comunes, como el lentivirus, causan respuestas inmunes en humanos, por lo que las terapias humanas propuestas a menudo se centran en el virus adenoasociado (AAV), porque las infecciones por AAV son en gran medida asintomáticas. Desafortunadamente, la capacidad de empaquetamiento efectiva de los vectores AAV es pequeña, aproximadamente 4,4 kb sin incluir las repeticiones terminales invertidas. [22] A modo de comparación, una proteína de fusión SpCas9-transcriptasa inversa tiene 6,3 kb, [1] [23] que ni siquiera tiene en cuenta el ARN guía alargado necesario para apuntar y preparar el sitio de interés. Sin embargo, se ha logrado una administración exitosa en ratones dividiendo el editor en dos vectores AAV [2] [3] [4] [24] o utilizando un adenovirus, [3] que tiene una mayor capacidad de empaquetamiento.
Los editores principales pueden utilizarse en las unidades genéticas . Un editor principal puede incorporarse en la mitad Cleaver de un sistema Cleave and Rescue / ClvR . En este caso, no está destinado a realizar una alteración precisa, sino simplemente a interrumpir. [25]
La PE es una de las tecnologías introducidas recientemente que permiten la transferencia de polimorfismos de un solo nucleótido (SNP) de una planta de cultivo individual a otra. La PE es lo suficientemente precisa como para ser utilizada para recrear un SNP arbitrario en un objetivo arbitrario, [14] incluyendo deleciones, inserciones y las 12 mutaciones puntuales sin necesidad de realizar también una ruptura de doble cadena o llevar una plantilla donante. [6]