Una proteína de membrana integral o intrínseca ( IMP ) [ 1] es un tipo de proteína de membrana que está permanentemente unida a la membrana biológica . Todas las proteínas transmembrana pueden clasificarse como IMP, pero no todas las IMP son proteínas transmembrana. [2] Las IMP comprenden una fracción significativa de las proteínas codificadas en el genoma de un organismo . [3] Las proteínas que cruzan la membrana están rodeadas de lípidos anulares , que se definen como lípidos que están en contacto directo con una proteína de membrana. Dichas proteínas solo pueden separarse de las membranas mediante el uso de detergentes , solventes no polares o, a veces, agentes desnaturalizantes .
Las proteínas que se adhieren sólo temporalmente a las membranas celulares se conocen como proteínas periféricas de membrana . Estas proteínas pueden asociarse con proteínas integrales de membrana o insertarse de forma independiente en la bicapa lipídica de varias maneras.
Se han determinado las estructuras tridimensionales de aproximadamente 160 proteínas integrales de membrana diferentes con resolución atómica mediante cristalografía de rayos X o espectroscopia de resonancia magnética nuclear . Son temas desafiantes para el estudio debido a las dificultades asociadas con la extracción y la cristalización . Además, las estructuras de muchos dominios proteicos solubles en agua de las IMP están disponibles en el Protein Data Bank . Se han eliminado sus hélices α de anclaje a la membrana para facilitar la extracción y la cristalización . Busque proteínas integrales de membrana en el PDB (basado en la clasificación de ontología génica )
Los IMP se pueden dividir en dos grupos:
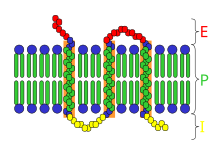
El tipo más común de IMP es la proteína transmembrana , que se extiende por toda la membrana biológica . Las proteínas de membrana de un solo paso cruzan la membrana solo una vez, mientras que las proteínas de membrana de múltiples pasos entran y salen, cruzando la membrana varias veces. Las proteínas de membrana de un solo paso se pueden clasificar como Tipo I, que están posicionadas de tal manera que su extremo carboxilo terminal está hacia el citosol , o Tipo II, que tienen su extremo amino terminal hacia el citosol. Las proteínas de Tipo III tienen múltiples dominios transmembrana en un solo polipéptido, mientras que el tipo IV consta de varios polipéptidos diferentes ensamblados juntos en un canal a través de la membrana. Las proteínas de Tipo V están ancladas a la bicapa lipídica a través de lípidos unidos covalentemente. Finalmente, las proteínas de Tipo VI tienen dominios transmembrana y anclajes lipídicos. [4]

Las proteínas monotópicas integrales están asociadas a la membrana desde un lado, pero no abarcan completamente la bicapa lipídica.
Muchos de los desafíos que enfrenta el estudio de las proteínas integrales de membrana se atribuyen a la extracción de dichas proteínas de la bicapa de fosfolípidos . Dado que las proteínas integrales abarcan todo el ancho de la bicapa de fosfolípidos, su extracción implica alterar los fosfolípidos que las rodean, sin causar ningún daño que pueda interrumpir la función o la estructura de las proteínas. Existen varios métodos exitosos para realizar la extracción, incluidos los usos de "detergentes, sal iónica baja (salting out), fuerza de corte y cambio rápido de presión". [5]
La Iniciativa de Estructura de Proteínas (PSI), financiada por el Instituto Nacional de Ciencias Médicas Generales (NIGMS) de los Estados Unidos, parte de los Institutos Nacionales de Salud (NIH), tiene entre sus objetivos determinar las estructuras tridimensionales de las proteínas y desarrollar técnicas para su uso en biología estructural , incluidas las proteínas de membrana. El modelado por homología se puede utilizar para construir un modelo de resolución atómica de la proteína integral "objetivo" a partir de su secuencia de aminoácidos y una estructura tridimensional experimental de una proteína homóloga relacionada. Este procedimiento se ha utilizado ampliamente para los receptores acoplados a proteína G ( GPCR) y sus complejos. [6]
Los IMP incluyen transportadores , enlazadores, canales , receptores , enzimas , dominios estructurales de anclaje a la membrana, proteínas involucradas en la acumulación y transducción de energía y proteínas responsables de la adhesión celular . La clasificación de los transportadores se puede encontrar en la Base de datos de clasificación de transportadores . [7]
A continuación se muestra un ejemplo de la relación entre el IMP (en este caso, el pigmento fotocaptador bacteriano, bacteriorrodopsina) y la membrana formada por la bicapa de fosfolípidos. En este caso, la proteína integral de membrana abarca la bicapa de fosfolípidos siete veces. La parte de la proteína que está incrustada en las regiones hidrofóbicas de la bicapa tiene forma de hélice alfa y está compuesta predominantemente por aminoácidos hidrofóbicos. El extremo C terminal de la proteína está en el citosol, mientras que la región N terminal está en el exterior de la célula. Una membrana que contiene esta proteína en particular es capaz de funcionar en la fotosíntesis. [8]
Ejemplos de proteínas integrales de membrana: