El haplogrupo D , también conocido como D-CTS3946 , es un haplogrupo del cromosoma Y. Al igual que su hermano relativamente lejano, el haplogrupo E-M96 , D-CTS3946 tiene el polimorfismo de evento único YAP+ , que define a su padre, el haplogrupo DE . D-CTS3946 tiene dos ramas basales, D1 y D2 . D1 y D2 se encuentran principalmente en el este de Asia , con baja frecuencia en Asia central y el sudeste asiático , y con muy baja frecuencia en África occidental y Asia occidental .
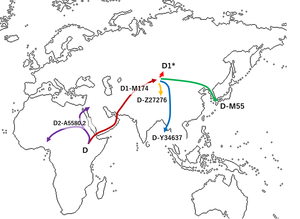
Haplogrupo D era anteriormente el nombre del linaje D D-M174 . Existen diversas propuestas con respecto al origen del haplogrupo DE , el padre de D, algunas sugieren un origen africano [2] y otras un origen asiático. [3] Pero se suponía, y generalmente se supone, que D-M174 era de origen asiático y se encuentra exclusivamente en Asia.
Haber et al. (2019) identificaron un haplogrupo, denominado "D0", en tres nigerianos. Definido por el SNP A5580.2, el haplogrupo "D0" está fuera de M174, pero pertenece al linaje D, comparte con él 7 SNP D-M174 del que carece E, y se determinó que divergió temprano de la rama D (cerca de la rama D /E división). Haber et al. (2019) consideraron varias posibilidades, incluido un origen africano y asiático para el haplogrupo, pero en parte debido al profundo arraigo del haplogrupo "D0", así como a los tiempos de divergencia temprana calculados recientemente para él y su haplogrupo padre, DE, Los autores concluyen a favor de un origen africano para D0 y DE, así como para el ancestro común (ahora conocido como D-CTS3946 o "D") de D0 y D-M174. Según Haber et al. (2019), D0 es una rama del linaje DE cerca de la división D/E pero en la rama D, divergiendo hace unos 71.000 años. Los autores encuentran tiempos de divergencia para DE*, E y D0, todos probablemente dentro de un período de hace aproximadamente 76.000-71.000 años, y una fecha probable para la salida de África de los antepasados de los euroasiáticos modernos (y la consiguiente mezcla neandertal) más tarde. , hace alrededor de 50.300-59.400 años, lo que, según ellos, también respalda un origen africano para esos haplogrupos. [1] Por lo tanto, se propone que D-CTS3946 se haya extendido tanto dentro como fuera de África: con una rama divergiendo en D0 en África, y otra rama que salió de África eventualmente divergiendo en D-M174 (es decir, con la mutación M174 que surgió más tarde de la D-CTS3946 que se había extendido a Asia).
Hallast et al. (2020), sobre haplogrupos antiguos y modernos utilizando un análisis filogenético de secuencias de haplogrupos C, D y FT, incluidos linajes de raíces profundas muy raros (como D0/D2 muestreados por Haber et al. 2019), argumenta, tomando los "raros y profundos -rooted D0", que las divisiones iniciales dentro del haplogrupo CT (antepasado de DE) ocurrieron en África. También argumentan que los análisis filogeográficos de los cromosomas Y no africanos antiguos y actuales apuntan al este y sudeste de Asia como el origen hace 55.000 a 50.000 años de todos los linajes masculinos no africanos supervivientes conocidos (aparte de los inmigrantes recientes) poco después. una migración inicial hace entre 70 y 55.000 años desde África del haplogrupo D basal y otros linajes y basales. Argumentan que estos linajes luego se expandieron rápidamente por Eurasia, luego se diversificaron en el sudeste asiático y luego se expandieron hacia el oeste hace entre 55 y 50 000 años, reemplazando a otros linajes locales dentro de Eurasia, y concluyen que el haplogrupo D (como D-M174) luego experimentó rápidas expansiones dentro de Eurasia. Poblaciones de Eurasia oriental y consta de 5 ramas diferentes que se formaron hace unos 45.000 años. Encuentran que estos haplogrupos tienen actualmente su mayor diversidad en Eurasia oriental (este/sureste de Asia). Se encontró que las poblaciones tibeto-birmanas del este y sudeste de Asia tenían la mayor diversidad. [4]

FTDNA también encontró otras tres muestras de D2 (también en 2019): dos en Al Wajh en la costa oeste de Arabia Saudita [5] [6] y otra en un sirio (él es D2b-FT51782). [7] [5] También se encontró una muestra D2b-FT51782 en al-Qirbi de Bayda [8] en Yemen que resultó tener varios miles de años de relación con la de Siria. [7] Hay otra persona (apellido desconocido) en la vecina gobernación de Shabwah con TMRCA 1700 y. Hace con el primero. [9] Michael Sager de FTDNA anunció en 2020 que se había encontrado otra muestra en un afroamericano [10] y otra en otro afroamericano. [10] [11] Las muestras encontradas en el descendiente sirio y en uno de los afroamericanos son hasta la fecha las muestras más basales de D2. [10] [5] [6] [11] La otra muestra afroamericana comparte una sucursal con los tres nigerianos. [10] La evidencia reciente (como también lo propone Haber et al.) sugiere que D2 es un haplogrupo altamente divergente cercano a la división DE pero en la rama D y que carece de la mutación M174 que poseen los otros linajes D conocidos (que pertenecen a su hermano D-M174). [5]
El haplogrupo D1 es un subclado del haplogrupo D-CTS3946.
El haplogrupo D2 es un subclado del paleolítico superior del haplogrupo D-CTS3946. D0 pasó a llamarse D2 y D-M174 pasó a llamarse D1 debido a este descubrimiento. [5] [6]
ISOGG (versión: 14.151). [12]
Teniendo en cuenta un linaje D0 africano poco común y el período resumido anteriormente, hemos argumentado (Haber et al. 2019) que es probable que las divisiones iniciales dentro de CT hayan ocurrido en África antes de la salida, y que tres linajes, C , D y FT, fueron realizados por los antepasados de los actuales no africanos. Cada uno de estos tres linajes se expandió posteriormente: C y D moderadamente, y FT masivamente". También (de una parte anterior del estudio): "Los genomas de los humanos actuales fuera de África se originaron casi en su totalidad a partir de una única emigración ~ 50.000-70.000 hace años, seguido de una mezcla en la que los neandertales contribuyeron con aproximadamente el 2% de todos los no africanos. Sin embargo, los detalles de esta migración inicial siguen siendo poco conocidos porque no hay análisis de ADN antiguos disponibles de este período clave, y la interpretación de los datos autosómicos actuales es complicada debido a los movimientos/remodelaciones posteriores de la población. Sin embargo, un locus conserva información específica masculina de este período temprano: el cromosoma Y, donde se ha construido una filogenia calibrada detallada. La migración inicial transportó tres linajes Y actuales: el raro haplogrupo D, el moderadamente raro C y el muy común linaje FT que ahora domina la mayoría de las poblaciones no africanas.Este artículo incorpora texto de esta fuente, que está disponible bajo la licencia CC BY 4.0.