La conversión genética es el proceso mediante el cual una secuencia de ADN reemplaza una secuencia homóloga de modo que las secuencias se vuelven idénticas después del evento de conversión. La conversión genética puede ser alélica , lo que significa que un alelo del mismo gen reemplaza a otro alelo, o ectópica , lo que significa que una secuencia de ADN paráloga convierte a otra.
La conversión de genes alélicos ocurre durante la meiosis cuando la recombinación homóloga entre sitios heterocigóticos da como resultado un desajuste en el emparejamiento de bases. Esta falta de coincidencia es luego reconocida y corregida por la maquinaria celular, lo que hace que uno de los alelos se convierta en el otro. Esto puede provocar una segregación no mendeliana de alelos en las células germinales. [1]
La recombinación ocurre no sólo durante la meiosis, sino también como un mecanismo de reparación de roturas de doble hebra (DSB) causadas por daños en el ADN. Estos DSB generalmente se reparan utilizando la cromátida hermana del dúplex roto y no el cromosoma homólogo, por lo que no darían lugar a una conversión alélica. La recombinación también se produce entre secuencias homólogas presentes en diferentes loci genómicos (secuencias parálogas) que han resultado de duplicaciones genéticas previas. Se conjetura que la conversión genética que se produce entre secuencias parálogas ( conversión genética ectópica ) es responsable de la evolución concertada de las familias de genes. [1] [2] [3]
La conversión de un alelo a otro a menudo se debe a la reparación de un desajuste de bases durante la recombinación homóloga : si una de las cuatro cromátidas durante la meiosis se empareja con otra cromátida, como puede ocurrir debido a la homología de secuencia , puede ocurrir una transferencia de cadena de ADN seguida de una reparación del desajuste. Esto puede alterar la secuencia de uno de los cromosomas, de modo que sea idéntico al otro.
La recombinación meiótica se inicia mediante la formación de una rotura de doble cadena (DSB). Los extremos 5' de la rotura luego se degradan, dejando largos salientes 3' de varios cientos de nucleótidos. Luego, uno de estos segmentos 3' de ADN monocatenario invade una secuencia homóloga en el cromosoma homólogo, formando un intermedio que puede repararse a través de diferentes vías, lo que resulta en cruces (CO) o no cruces (NCO). En varios pasos del proceso de recombinación, se forma ADN heterodúplex (ADN bicatenario que consta de hebras simples de cada uno de los dos cromosomas homólogos que pueden ser perfectamente complementarios o no). Cuando se producen desajustes en el ADN heterodúplex, la secuencia de una cadena se reparará para unirse a la otra cadena con perfecta complementariedad, lo que lleva a la conversión de una secuencia en otra. Este proceso de reparación puede seguir cualquiera de dos vías alternativas, como se ilustra en la Figura. Por una vía, se forma una estructura llamada doble unión Holliday (DHJ), que conduce al intercambio de cadenas de ADN. Por la otra vía, denominada recocido de hebras dependiente de la síntesis (SDSA), hay intercambio de información pero no intercambio físico. La conversión genética ocurrirá durante SDSA si las dos moléculas de ADN son heterocigotas en el sitio de la reparación recombinacional. La conversión de genes también puede ocurrir durante la reparación recombinacional que involucra a un DHJ, y esta conversión de genes puede estar asociada con la recombinación física de los dúplex de ADN en los dos lados del DHJ.
La conversión genética sesgada (BGC) ocurre cuando un alelo tiene una mayor probabilidad de ser donante que el otro en un evento de conversión genética. Por ejemplo, cuando se produce una discrepancia T:G, sería más o menos probable que se corrija a un par C:G que a un par T:A. Esto le da a ese alelo una mayor probabilidad de transmisión a la siguiente generación. La conversión genética imparcial significa que ambas posibilidades ocurren con la misma probabilidad.
La conversión de genes sesgada por GC (gBGC) es el proceso mediante el cual el contenido de GC del ADN aumenta debido a la conversión de genes durante la recombinación. [2] Existe evidencia de gBGC en levaduras y humanos y la teoría se ha probado más recientemente en otros linajes eucariotas. [4] En secuencias de ADN humano analizadas, se ha descubierto que la tasa de cruce se correlaciona positivamente con el contenido de GC. [2] Las regiones pseudoautosómicas (PAR) de los cromosomas X e Y en humanos, que se sabe que tienen altas tasas de recombinación, también tienen altos contenidos de GC. [1] Ciertos genes de mamíferos que experimentan una evolución concertada (por ejemplo, operones ribosómicos, ARNt y genes de histonas) son muy ricos en GC. [1] Se ha demostrado que el contenido de GC es mayor en genes de histonas parálogos humanos y de ratón que son miembros de grandes subfamilias (presumiblemente en proceso de evolución concertada) que en genes de histonas parálogos con secuencias relativamente únicas. [5] También hay evidencia de sesgo de GC en el proceso de reparación de discordancias. [1] Se cree que esto puede ser una adaptación a la alta tasa de desaminación de metilcitosina que puede conducir a transiciones C→T.
El gen Fxy o Mid1 en algunos mamíferos estrechamente relacionados con los ratones domésticos (humanos, ratas y otras especies de Mus) se encuentra en la región ligada al sexo del cromosoma X. Sin embargo, en Mus musculus , se ha translocado recientemente de manera que el extremo 3' del gen se superpone con la región PAR del cromosoma X, que se sabe que es un punto de acceso a la recombinación. Esta porción del gen ha experimentado un aumento dramático en el contenido de GC y la tasa de sustitución en la posición del tercer codón, así como en los intrones, pero la región 5' del gen, que está ligada al cromosoma X, no. Debido a que este efecto está presente sólo en la región del gen que experimenta una mayor tasa de recombinación, debe deberse a una conversión genética sesgada y no a una presión selectiva. [2]
El contenido de GC varía ampliamente en el genoma humano (40-80%), pero parece haber grandes secciones del genoma donde el contenido de GC es, en promedio, mayor o menor que en otras regiones. [1] Estas regiones, aunque no siempre muestran límites claros, se conocen como isocoras. Una posible explicación para la presencia de isocoras ricas en GC es que evolucionaron debido a la conversión de genes sesgada por GC en regiones con altos niveles de recombinación.
Los estudios de conversión de genes han contribuido a nuestra comprensión de la función adaptativa de la recombinación meiótica. El patrón de segregación ordinario de un par de alelos (Aa) entre los 4 productos de la meiosis es 2A:2a. La detección de eventos de conversión de genes poco frecuentes (por ejemplo, patrones de segregación 3:1 o 1:3 durante las meiosis individuales) proporciona información sobre las vías alternativas de recombinación que conducen a cromosomas cruzados o no cruzados. Se cree que los eventos de conversión genética surgen cuando los alelos "A" y "a" se encuentran cerca de la ubicación exacta de un evento de recombinación molecular. Por lo tanto, es posible medir la frecuencia con la que los eventos de conversión genética se asocian con el cruce o no cruce de regiones cromosómicas adyacentes al evento de conversión inmediata, pero fuera de él. Se han llevado a cabo numerosos estudios sobre la conversión de genes en diversos hongos (que son especialmente adecuados para tales estudios), y Whitehouse ha revisado los resultados de estos estudios. [6] De esta revisión se desprende claramente que la mayoría de los eventos de conversión de genes no están asociados con el intercambio de marcadores externos. Por lo tanto, la mayoría de los eventos de conversión de genes en los diferentes hongos estudiados están asociados con el no cruce de marcadores externos. Los eventos de conversión de genes sin cruce se producen principalmente mediante el recocido de hebras dependientes de la síntesis (SDSA). [7] Este proceso implica un intercambio de información limitado, pero no un intercambio físico de ADN, entre los dos cromosomas homólogos participantes en el sitio del evento de conversión, y se produce poca variación genética. Por tanto, las explicaciones de la función adaptativa de la recombinación meiótica que se centran exclusivamente en el beneficio adaptativo de producir nueva variación genética o intercambio físico parecen inadecuadas para explicar la mayoría de los eventos de recombinación durante la meiosis. Sin embargo, la mayoría de los eventos de recombinación meiótica pueden explicarse mediante la propuesta de que son una adaptación para reparar el daño en el ADN que se transmitirá a los gametos. [8]
De particular interés, desde el punto de vista de que la recombinación es una adaptación para la reparación del ADN, son los estudios en levaduras que muestran que la conversión de genes en las células mitóticas aumenta con los rayos UV [9] [10] y la radiación ionizante [11].
En las discusiones sobre enfermedades genéticas en humanos, las conversiones de genes mediadas por pseudogenes que introducen mutaciones patógenas en genes funcionales es un mecanismo de mutación bien conocido. Por el contrario, es posible que los pseudogenes sirvan como plantillas. Durante el curso de la evolución, genes fuente funcionales que son potencialmente ventajosos se han derivado de múltiples copias en su único gen fuente. Los cambios provocados por pseudogenes podrían llegar a arreglarse siempre y cuando no poseyeran efectos nocivos. [12] Entonces, de hecho, los pseudogenes pueden actuar como fuentes de variantes de secuencia que pueden transferirse a genes funcionales en nuevas combinaciones y sobre las que se puede actuar mediante selección . La lectina 11 ( SIGLEC 11), una inmunoglobulina humana que se une al ácido siálico, puede considerarse un ejemplo de un evento de conversión genética que ha desempeñado un papel importante en la evolución. Al comparar los genes homólogos del SIGLEC11 humano y su pseudogén en el chimpancé , el gorila y el orangután , parece que hubo una conversión genética de la secuencia de las regiones 5' aguas arriba y los exones que codifican el dominio de reconocimiento del ácido siálico, aproximadamente a 2 kbp de la estrecha flanqueando el pseudogén hSIGLECP16 (Hayakawa et al., 2005). Las tres pruebas relativas a este evento han sugerido en conjunto que se trata de un cambio adaptativo que es muy importante desde el punto de vista evolutivo en el género Homo . Esto incluye que solo en el linaje humano ocurrió esta conversión genética, la corteza cerebral ha adquirido una expresión importante de SIGLEC11 específicamente en el linaje humano y la exhibición de un cambio en la unión del sustrato en el linaje humano en comparación con la de su contraparte en los chimpancés. Por supuesto, la frecuencia de la contribución de este mecanismo de conversión de genes mediado por pseudogenes a los cambios funcionales y adaptativos en la evolución humana aún se desconoce y hasta ahora apenas se ha explorado. [13] A pesar de ello, la introducción de cambios genéticos positivamente selectivos mediante dicho mecanismo puede ponerse a consideración con el ejemplo de SIGLEC11. A veces, debido a la interferencia de elementos transponibles en algunos miembros de una familia de genes, se produce una variación entre ellos y, finalmente, también puede detener la tasa de conversión de genes debido a la falta de similitud de secuencia, lo que conduce a una evolución divergente .
A partir de varios análisis del genoma, se concluyó que las roturas de doble cadena (DSB) pueden repararse mediante recombinación homóloga mediante al menos dos vías diferentes pero relacionadas. [12] En caso de una vía principal, se emplearán secuencias homólogas en ambos lados de la DSB, lo que parece ser análogo al modelo conservador de reparación de DSB [14] que se propuso originalmente para la recombinación meiótica en levadura. [15] donde la vía menor está restringida a un solo lado del DSB como lo postula el modelo de invasión unilateral no conservador. [16] Sin embargo, en ambos casos la secuencia de los socios de recombinación se conservará absolutamente. En virtud de su alto grado de homología, las nuevas copias de genes que surgieron después de la duplicación del gen tienden naturalmente a eventos de cruce desigual o de conversión de genes unidireccionales. En el último proceso, existen las secuencias aceptora y donante y la secuencia aceptora será reemplazada por una secuencia copiada del donante, mientras que la secuencia del donante permanece sin cambios. [13]
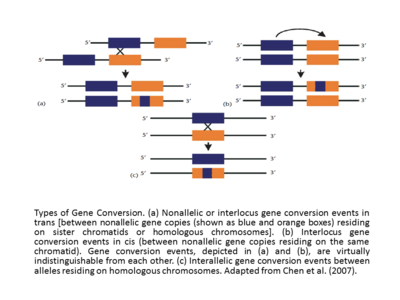
La homología efectiva entre las secuencias que interactúan hace que el evento de conversión del gen sea exitoso. Además, la frecuencia de conversión de genes es inversamente proporcional a la distancia entre las secuencias que interactúan en cis, [17] [12] y la tasa de conversión de genes suele ser directamente proporcional a la longitud del tracto de secuencia ininterrumpida en la región supuesta convertida. Parece que los tractos de conversión que acompañan al cruce son más largos (longitud media = ~460 pb) que los tractos de conversión sin cruce (longitud media = 55–290 pb). [18] En los estudios de genes de globulina humana, se ha apoyado durante mucho tiempo que el evento de conversión de genes o los eventos de migración de ramas pueden ser promovidos o inhibidos por motivos específicos que existen en las proximidades de la secuencia de ADN (Papadakis y Patrinos, 1999). ). [12] Otra clasificación básica de los eventos de conversión de genes es la conversión de genes interlocus (también llamada no alélica) e interalélica. Los eventos de conversión de genes cis o trans no alélicos o interlocus ocurren entre copias de genes no alélicos que residen en cromátidas hermanas o cromosomas homólogos y, en el caso de interalélicos, los eventos de conversión de genes tienen lugar entre alelos que residen en cromosomas homólogos (Adaptado de Chen et al., (2007) [13] [12] Si se comparan los eventos de conversión de genes interlocus, con frecuencia se revelará que exhiben una direccionalidad sesgada. A veces, como en el caso de los genes de globina humana (Papadakis y Patrinos, 1999), [12 ] la dirección de conversión de genes se correlaciona con los niveles de expresión relativos de los genes que participan en el evento, con el gen expresado en un nivel más alto, llamado gen 'maestro', convirtiendo el de menor expresión, llamado gen 'esclavo'. En un contexto evolutivo, la regla del "gen maestro/esclavo" debe explicarse con cautela. De hecho, el aumento en la transcripción del gen muestra no sólo el aumento en la probabilidad de que se utilice como donante sino también como aceptor. [12] [19]
Normalmente, un organismo que ha heredado diferentes copias de un gen de cada uno de sus padres se denomina heterocigoto. Esto se representa genéricamente como genotipo: Aa (es decir, una copia de la variante ( alelo ) 'A' y una copia del alelo 'a'). Cuando un heterocigoto crea gametos por meiosis , los alelos normalmente se duplican y terminan en una proporción de 2:2 en las 4 células resultantes que son productos directos de la meiosis. Sin embargo, en la conversión de genes, se observa una proporción distinta a la esperada 2A:2a, en la que A y a son los dos alelos. Algunos ejemplos son 3A:1a y 1A:3a. En otras palabras, puede haber, por ejemplo, tres veces más alelos A que alelos a expresados en las células hijas, como es el caso en 3A:1a.
La conversión genética que resulta en la mutación del gen CYP21A2 es una causa genética subyacente común de hiperplasia suprarrenal congénita . La conversión de genes somáticos es uno de los mecanismos que puede dar lugar al retinoblastoma familiar , un cáncer congénito de la retina , y se teoriza que la conversión de genes puede desempeñar un papel en el desarrollo de la enfermedad de Huntington .