La malato deshidrogenasa (oxaloacetato-descarboxilante) (NADP + ) ( EC 1.1.1.40) o enzima NADP-málica (NADP-ME) es una enzima que cataliza la reacción química en presencia de un ion metálico bivalente: [1]
Así, los dos sustratos de esta enzima son el (S)-malato y el NADP + , mientras que sus tres productos son el piruvato , el CO2 y el NADPH . El malato se oxida a piruvato y CO2 , y el NADP + se reduce a NADPH.
Esta enzima pertenece a la familia de las oxidorreductasas , en concreto aquellas que actúan sobre el grupo CH-OH del donador con NAD + o NADP + como aceptor. El nombre sistemático de esta clase de enzimas es (S)-malato:NADP + oxidorreductasa (oxaloacetato-descarboxilante) . Esta enzima participa en el metabolismo del piruvato y la fijación de carbono . La enzima NADP-málica es una de las tres enzimas de descarboxilación utilizadas en los mecanismos de concentración de carbono inorgánico de las plantas C4 y CAM . Las otras son la enzima NAD-málica y la carboxiquinasa PEP . [2] [3] Aunque a menudo predomina una de las tres descarboxilasas fotosintéticas, también se ha demostrado que existe el funcionamiento simultáneo de las tres. [4]
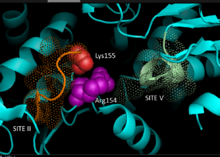
Basándose en datos cristalográficos de enzimas málicas dependientes de NADP homólogas de origen mamífero, se ha desarrollado un modelo 3D para la vía C 4 NADP-ME en plantas, identificando los residuos clave implicados en la unión al sustrato o la catálisis. La unión de dinucleótidos implica dos motivos GXGXXG ricos en glicina , un surco hidrofóbico que implica al menos seis residuos de aminoácidos y un residuo cargado negativamente al final de la cadena βB. [5] [6] La secuencia primaria del primer motivo, 240 GLGDLG 245 , es un marcador de consenso para la unión de fosfato, evidenciando la participación en la unión de NADP, mientras que el otro motivo rico en glicina adopta un pliegue de Rossmann clásico , también un marcador típico para la unión del cofactor NADP . [7] Los experimentos de mutagénesis en NADP-ME de maíz han apoyado el modelo actual. [1] La sustitución de valina por glicina en cualquiera de las regiones del motivo dejó a la enzima completamente inactiva, mientras que el análisis espectral no indicó cambios importantes con respecto a la forma de tipo salvaje. Los datos sugieren un deterioro directo en un residuo clave involucrado en la unión o catálisis en lugar de un residuo entre dominios que influye en la estabilidad conformacional. Además, se ha demostrado que un residuo clave de arginina en el sitio 237 interactúa tanto con sustratos de malato como de NADP + , formando interacciones electrostáticas favorables clave para el ácido carboxílico cargado negativamente y el grupo fosfato respectivamente. Aún está por determinar si el residuo desempeña un papel en la unión del sustrato o en el posicionamiento del sustrato para la catálisis. [8] El residuo de lisina 255 se ha implicado como una base catalítica para la reactividad de las enzimas; sin embargo, aún se requieren más estudios para establecer de manera concluyente su papel bioquímico. [1]
A partir de 2007 [actualizar], se han resuelto 3 estructuras para esta clase de enzimas, con los códigos de acceso PDB 1GQ2, 1GZ4 y 2AW5. [ cita requerida ]
En un contexto más amplio, las enzimas málicas se encuentran en una amplia gama de organismos eucariotas , desde hongos hasta mamíferos, y más allá de eso, se ha demostrado que se localizan en una variedad de ubicaciones subcelulares, incluido el citosol , las mitocondrias y los cloroplastos . C 4 NADP-ME, específicamente, se localiza en las plantas en los cloroplastos de la vaina del haz . [1]
Durante la fotosíntesis de C 4 , una vía evolucionada para aumentar las concentraciones localizadas de CO 2 bajo la amenaza de una mayor fotorrespiración , el CO 2 se captura dentro de las células del mesófilo , se fija como oxaloacetato , se convierte en malato y se libera internamente dentro de las células de la vaina del haz para alimentar directamente la actividad de RuBisCO . [9] Esta liberación de CO 2 fijado , desencadenada por la descarboxilación favorable del malato en piruvato , está mediada por la enzima málica dependiente de NADP. De hecho, la importancia de la actividad de NADP-ME en la conservación de CO 2 se evidencia en un estudio realizado con plantas transgénicas que exhiben una mutación de pérdida de función de NADP-ME. Las plantas con la mutación experimentaron un 40% de la actividad de NADP-ME de tipo salvaje y lograron una absorción de CO 2 significativamente reducida incluso a altos niveles intercelulares de CO 2 , lo que evidencia la importancia biológica de NADP-ME en la regulación del flujo de carbono hacia el ciclo de Calvin . [10] [11]
Se ha demostrado que la expresión de NADP-ME está regulada por factores de estrés abiótico . En las plantas CAM , las condiciones de sequía hacen que el estoma permanezca cerrado en gran medida para evitar la pérdida de agua por evapotranspiración , lo que desafortunadamente conduce a la falta de CO2 . En compensación, el estoma cerrado activa la traducción de NADP-ME para reforzar la alta eficiencia de la asimilación de CO2 durante los breves intervalos de ingesta de CO2 , lo que permite que continúe la fijación de carbono .
Además de la regulación a escala de tiempo más larga por medio del control de la expresión, la regulación a escala de tiempo corta puede ocurrir a través de mecanismos alostéricos . Se ha demostrado que C 4 NADP-ME es parcialmente inhibido por su sustrato , el malato, lo que sugiere dos sitios de unión independientes: uno en el sitio activo y otro en un sitio alostérico. Sin embargo, el efecto inhibidor exhibe dependencia del pH , existente a un pH de 7 pero no a un pH de 8. El control de la actividad enzimática debido a los cambios de pH se alinea con la hipótesis de que NADP-ME es más activo mientras la fotosíntesis está en progreso: las reacciones de luz activas conducen a un aumento de la basicidad dentro del estroma del cloroplasto , la ubicación de NADP-ME, lo que conduce a un efecto inhibidor disminuido del malato sobre NADP-ME y, por lo tanto, promueve un estado más activo. Por el contrario, las reacciones de luz más lentas conducen a un aumento de la acidez dentro del estroma, lo que promueve la inhibición de NADP-ME por malato. Dado que los productos de alta energía de las reacciones de la luz , NADPH y ATP , son necesarios para que se lleve a cabo el ciclo de Calvin , una acumulación de CO 2 sin ellos no es útil, lo que explica la necesidad del mecanismo regulador. [12]
Esta proteína puede utilizar el modelo de morfeína de regulación alostérica . [13]
La enzima NADP-málica, como todas las demás descarboxilasas C4 , no evolucionó de novo para la acumulación de CO2 para ayudar a RuBisCO . [14] En cambio, NADP-ME se transformó directamente a partir de una especie C3 en la fotosíntesis , e incluso orígenes anteriores de un ancestro cistólico antiguo . En el citosol , la enzima existía como una serie de isoformas de mantenimiento destinadas a una variedad de funciones, incluido el mantenimiento del nivel de malato durante la hipoxia , la separación de microsporas y la defensa de patógenos . Con respecto al mecanismo de evolución, se cree que la funcionalidad C4 se originó a partir de un error de duplicación de genes tanto dentro de las regiones promotoras , lo que desencadenó la sobreexpresión en las células de la vaina del haz, como dentro de la región codificante, lo que generó neofuncionalización . [15] La selección para la función de preservación de CO2 , así como la utilización mejorada de agua y nitrógeno en condiciones de estrés, fue moldeada por presiones naturales. [16]