
La formilación se refiere a cualquier proceso químico en el que un compuesto se funcionaliza con un grupo formilo (-CH=O). En química orgánica, el término se utiliza más comúnmente con respecto a compuestos aromáticos (por ejemplo, la conversión de benceno en benzaldehído en la reacción de Gattermann-Koch ). En bioquímica, la reacción está catalizada por enzimas como las formiltransferasas .
La formilación generalmente implica el uso de agentes de formilación, reactivos que dan lugar al grupo CHO. Entre los muchos reactivos de formilación, particularmente importantes son el ácido fórmico y el monóxido de carbono . [1] Una reacción de formilación en química orgánica se refiere a reacciones orgánicas en las que un compuesto orgánico se funcionaliza con un grupo formilo (-CH=O). La reacción es una ruta hacia aldehídos ( C -CH=O), formamidas ( N -CH=O) y ésteres de formato ( O -CH=O).
Un reactivo que libera el grupo formilo se llama agente formilante . [2]
Un proceso de formilación particularmente importante es la hidroformilación , que convierte los alquenos en el aldehído homologado.

Las reacciones de formilación son una forma de sustitución aromática electrofílica y, por lo tanto, funcionan mejor con materiales de partida ricos en electrones. Los fenoles son un sustrato común, ya que se desprotonan fácilmente para convertirse en excelentes nucleófilos fenóxidos . También se puede esperar que reaccionen otros sustratos ricos en electrones, como el mesitileno , el pirrol o los anillos aromáticos fusionados. El benceno reaccionará en condiciones agresivas, pero los anillos desactivados, como la piridina, son difíciles de formilar de manera efectiva.
Muchas reacciones de formilación seleccionarán solo el producto orto (p. ej. , salicilaldehído ), atribuido a la atracción entre el fenóxido y el reactivo de formilación. Se han invocado interacciones iónicas para los centros de nitrógeno catiónicos en la reacción de Vilsmeier-Haack y la reacción de Duff , y el carbeno deficiente en electrones en la reacción de Reimer-Tiemann ; se ha invocado la coordinación con metales de alta oxidación en las formilaciones de Casiraghi y Rieche (cf. reacción de Kolbe-Schmitt ).
La reacción directa entre el fenol y el paraformaldehído es posible a través de la formilación de Casiraghi, [4] pero otros métodos aplican formas enmascaradas de formaldehído, en parte para limitar la formación de resinas de fenol formaldehído . Los aldehídos son fuertemente desactivantes y, como tales, los fenoles generalmente solo reaccionan una vez. Sin embargo, ciertas reacciones, como la reacción de Duff , pueden dar una doble adición. [5]
La formilación se puede aplicar a otros anillos aromáticos. Como generalmente comienza con el ataque nucleofílico del grupo aromático, la densidad electrónica del anillo es un factor importante. Se sabe que algunos compuestos aromáticos, como el pirrol, se formilan de forma regioselectiva. [6]
La formilación de los anillos de benceno se puede lograr mediante la reacción de Gattermann y la reacción de Gattermann-Koch . Estas implican una catálisis ácida fuerte y se desarrollan de manera similar a la reacción de Friedel-Crafts .
La hidroformilación de alquenos es el método más importante para obtener formilos alifáticos (es decir, aldehídos). La reacción está restringida en gran medida a entornos industriales. Existen varios métodos especializados para la síntesis a escala de laboratorio, incluida la reacción de Sommelet , la síntesis de aldehídos de Bouveault o la síntesis de aldehídos de Bodroux-Chichibabin .
En bioquímica, la adición de un grupo funcional formilo se denomina "formilación". Un grupo funcional formilo consiste en un carbonilo unido a hidrógeno. Cuando está unido a un grupo R , un grupo formilo se denomina aldehído .
La formilación se ha identificado en varios procesos biológicos críticos. Marcker y Sanger descubrieron por primera vez que la metionina estaba formilada en E. coli en 1964 [7] y más tarde se identificó que estaba involucrada en el inicio de la síntesis de proteínas en bacterias y orgánulos. [8] La formación de N -formilmetionina es catalizada por la enzima metionil-ARNt Met transformilasa . [9] Además, ocurren dos reacciones de formilación en la biosíntesis de novo de purinas . Estas reacciones son catalizadas por las enzimas glicinamida ribonucleótido (GAR) transformilasa y 5-aminoimidazol-4-carboxiamida ribótido (AICAR) transformilasa . [10] Más recientemente, se ha descubierto que la formilación es una modificación de histonas , que puede modular la expresión génica.
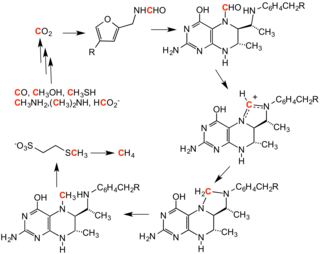
La formilación del metanofurano inicia el ciclo de la metanogénesis . El grupo formilo se deriva del dióxido de carbono y se convierte en metano .

En bacterias y orgánulos, el inicio de la síntesis de proteínas está señalado por la formación de formil-metionil-ARNt (ARNt fMet ). Esta reacción depende del 10-formiltetrahidrofolato y de la enzima metionil-ARNt formiltransferasa. [9] Esta reacción no es utilizada por eucariotas o Archaea, ya que la presencia de ARNt fMet en células no bacterianas se considera material intrusivo y se elimina rápidamente. Después de su producción, el ARNt fMet se entrega a la subunidad 30S del ribosoma para iniciar la síntesis de proteínas. fMet posee la misma secuencia de codones que la metionina. Sin embargo, fMet solo se utiliza para el inicio de la síntesis de proteínas y, por lo tanto, se encuentra solo en el extremo N de la proteína. La metionina se utiliza durante la traducción restante. En E. coli , el ARNt fMet es reconocido específicamente por el factor de iniciación IF-2 , ya que el grupo formilo bloquea la formación del enlace peptídico en el extremo N de la metionina. [9]
Una vez que se logra la síntesis de proteínas, el grupo formilo de la metionina puede ser eliminado por la péptido deformilasa . El residuo de metionina puede ser eliminado aún más por la enzima metionina aminopeptidasa .

Se requieren dos reacciones de formilación en la síntesis de novo de once pasos del monofosfato de inosina (IMP) , el precursor de los ribonucleótidos de purina AMP y GMP. La transformilasa del ribonucleótido de glicinamida (GAR) cataliza la formilación de GAR a formilglicinamidina ribótida (FGAR) en la cuarta reacción de la vía. En el penúltimo paso de la biosíntesis de novo de purina, la 5-aminoimidazol-4-carboxamida ribótida (AICAR) se formilan a 5-formaminoimidazol-4-carboxamida ribótida (FAICAR) por la transformilasa AICAR . [10]
La transformilasa PurN GAR se encuentra en eucariotas y procariotas. Sin embargo, se ha identificado una segunda transformilasa GAR, la transformilasa PurT GAR, en E. coli . Si bien las dos enzimas no tienen conservación de secuencia y requieren diferentes donantes de formilo, la actividad específica y la Km para GAR son las mismas en las transformilasas PurT y PurN GAR.
La transformilasa PurN GAR 1CDE utiliza la coenzima N10-formiltetrahidrofolato (N10-formil-THF) como donante de formilo para formilar el grupo α-amino de GAR. En eucariotas, la transformilasa PurN GAR es parte de una proteína multifuncional grande, pero se encuentra como proteína única en procariotas. [12]

Se propone que la reacción de formilación se produce a través de una reacción de transferencia directa en la que el grupo amino de GAR ataca nucleofílicamente al N10-formil-THF creando un intermedio tetraédrico. [10] Como el grupo α-amino de GAR es relativamente reactivo, se propone que la desprotonación del nucleófilo se produce por el disolvente. En el sitio activo, Asn 106, His 108 y Asp 144 están posicionados para ayudar con la transferencia de formilo. [12] Sin embargo, los estudios de mutagénesis han indicado que estos residuos no son esenciales individualmente para la catálisis, ya que solo las mutaciones de dos o más residuos inhiben la enzima. Con base en la estructura, se cree que el Asp144 cargado negativamente aumenta el pKa de His108, lo que permite que el grupo imidazolio protonado de His108 mejore la electrofilicidad del grupo formilo del N10-formil-THF. Además, se cree que His108 y Asn106 estabilizan el oxianión formado en el estado de transición. [13]

La transformilasa PurT GAR requiere formato como donante de formilo y ATP para la catálisis. Se ha estimado que la transformilasa PurT GAR lleva a cabo entre el 14 y el 50 % de las formilaciones de GAR en E. coli . La enzima es miembro de la superfamilia de proteínas que captan ATP . [14]
Se ha propuesto un mecanismo secuencial para la transformilasa PurT GAR en el que se propone que primero se forme un intermediario de fosfato de formil de vida corta. Luego, este intermediario de fosfato de formil sufre un ataque nucleofílico por parte de la amina GAR para la transferencia del grupo formilo. Se ha detectado un intermediario de fosfato de formil en experimentos de mutagénesis, en los que la transformilasa PurT GAR mutante tenía una afinidad débil por el formato. [12] La incubación de la transformilasa PurT GAR con fosfato de formil, ADP y GAR produce ATP y FGAR. Esto indica además que el fosfato de formil puede ser un intermediario, ya que es cinética y químicamente competente para llevar a cabo la reacción de formilación en la enzima. [15] También se ha propuesto que se forme un intermediario de fosfato enzimático que precede al intermediario de fosfato de formil basándose en estudios de intercambio de isótopos posicionales. [15] Sin embargo, los datos estructurales indican que el formato puede estar posicionado para un ataque directo al fosfato γ de ATP en el sitio activo de la enzima para formar el intermediario formilfosfato. [14]

La transformilasa AICAR requiere la coenzima N10-formiltetrahidrofolato (N10-formil-THF) como donante de formilo para la formilación de AICAR a FAICAR. Sin embargo, la transformilasa AICAR y la transformilasa GAR no comparten una gran similitud de secuencia ni homología estructural. [13]

La amina en AICAR es mucho menos nucleófila que su contraparte en GAR debido a la deslocalización de electrones en AICAR a través de la conjugación. Por lo tanto, el nucleófilo N5 de AIRCAR debe activarse para que se produzca la reacción de formilación. Se ha descubierto que la histidina 268 y la lisina 267 son esenciales para la catálisis y se conservan en todas las transformilasas AICAR. La histidina 268 está involucrada en la desprotonación del nucleófilo N5 de AICAR, mientras que se propone que la lisina 267 estabiliza el intermediario tetraédrico. [13]
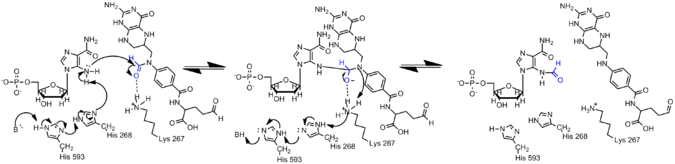

La ε-formilación es una de las muchas modificaciones postraduccionales que ocurren en las proteínas histonas, que se ha demostrado que modulan las conformaciones de la cromatina y la activación genética.

La formilación se ha identificado en el Nε de los residuos de lisina en histonas y proteínas. Esta modificación se ha observado en las histonas de enlace y en las proteínas del grupo de alta movilidad , es muy abundante y se cree que tiene un papel en la epigenética de la función de la cromatina. Se ha demostrado que las lisinas que están formiladas desempeñan un papel en la unión del ADN. Además, se ha detectado formilación en lisinas de histonas que también se sabe que están acetiladas y metiladas. Por lo tanto, la formilación puede bloquear otras modificaciones postraduccionales. [16] La formilación se detecta con mayor frecuencia en 19 sitios de modificación diferentes en la histona H1. La expresión genética de la célula se ve muy alterada por la formilación, lo que puede causar enfermedades como el cáncer. El desarrollo de estas modificaciones puede deberse al estrés oxidativo. [16]
En las proteínas histonas, la lisina es modificada típicamente por las histonas acetiltransferasas (HAT) y las histonas desacetilasas (HDAC o KDAC). La acetilación de la lisina es fundamental para la regulación y expresión de ciertos genes. El estrés oxidativo crea un entorno significativamente diferente en el que la acetillisina puede ser rápidamente desplazada por la formación de formillisina debido a la alta reactividad de las especies de formilfosfato. Actualmente se cree que esta situación es causada por daño oxidativo del ADN. Se ha propuesto un mecanismo para la formación de formilfosfato, que depende en gran medida del ADN dañado por oxidación y está impulsado principalmente por la química radical dentro de la célula. [17] El formilfosfato producido puede luego usarse para formilar lisina. Se cree que el estrés oxidativo juega un papel en la disponibilidad de residuos de lisina en la superficie de las proteínas y la posibilidad de ser formiladas.

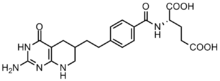
La inhibición de las enzimas implicadas en la biosíntesis de purina se ha explotado como un posible objetivo farmacológico para la quimioterapia.
Las células cancerosas requieren altas concentraciones de purinas para facilitar la división [12] y tienden a depender de la síntesis de novo en lugar de la vía de rescate de nucleótidos . [13] [18] Se han desarrollado varios inhibidores basados en folato para inhibir las reacciones de formilación por la transformilasa GAR y la transformilasa AICAR. [19] El primer inhibidor de la transformilasa GAR, Lometrexol [(6R)5,10-didesazatetrahidrofolato], se desarrolló en la década de 1980 a través de una colaboración entre Eli Lilly y laboratorios académicos. [20] Aunque similar en estructura al N10-formil-THF, el lometrexol es incapaz de llevar a cabo reacciones de transferencia de un carbono. [19] Además, también se han sintetizado varios inhibidores basados en GAR de la transformilasa GAR. [19] Se ha descubierto que el desarrollo de inhibidores basados en folato es particularmente complicado, ya que los inhibidores también regulan negativamente la enzima folipoliglutamato sintasa , que agrega γ-glutamatos adicionales a los folatos monoglutamato y antifolatos después de ingresar a la célula para aumentar la afinidad enzimática. Esta mayor afinidad puede conducir a una resistencia a los antifolatos. [18]
El síndrome de Leigh es un trastorno neurodegenerativo que se ha relacionado con un defecto en una reacción de formilación enzimática. El síndrome de Leigh se asocia típicamente con defectos en la fosforilación oxidativa, que ocurre en las mitocondrias. [21] La secuenciación del exoma se ha utilizado para identificar una mutación en el gen que codifica para la metionil-ARNt formiltransferasa mitocondrial (MTFMT) en pacientes con síndrome de Leigh. Se cree que la mutación c.626C>T identificada en MTFMT que produce síntomas del síndrome de Leigh altera el empalme de exones, lo que conduce a una mutación de cambio de marco y un codón de terminación prematuro. Se encontró que los individuos con la mutación MTFMT c.626C>T tenían niveles reducidos de fMet-ARNtMet y cambios en el nivel de formilación de COX1 traducido mitocondrialmente. Este vínculo proporciona evidencia de la necesidad de metionina formilada en el inicio de la expresión de ciertos genes mitocondriales. [22]