
Los análogos de ácidos nucleicos son compuestos análogos ( estructuralmente similares) al ARN y ADN naturales , utilizados en medicina y en la investigación de biología molecular. Los ácidos nucleicos son cadenas de nucleótidos, que se componen de tres partes: una cadena principal de fosfato , un azúcar pentosa, ya sea ribosa o desoxirribosa , y una de cuatro nucleobases . Un análogo puede tener cualquiera de estos alterados. [1] Normalmente, las nucleobases análogas confieren, entre otras cosas, diferentes propiedades de apareamiento y apilamiento de bases. Los ejemplos incluyen bases universales, que pueden emparejarse con las cuatro bases canónicas, y análogos de la cadena principal de fosfato-azúcar como PNA , que afectan las propiedades de la cadena (PNA puede incluso formar una triple hélice ). [2] Los análogos de ácidos nucleicos también se denominan xenoácidos nucleicos y representan uno de los pilares principales de la xenobiología , el diseño de formas de vida nuevas en la naturaleza basadas en bioquímicas alternativas.
Los ácidos nucleicos artificiales incluyen los ácidos nucleicos peptídicos (PNA), los ácidos nucleicos morfolino y bloqueados (LNA), así como los ácidos nucleicos glicolados (GNA), los ácidos nucleicos treosa (TNA) y los ácidos nucleicos hexitol (HNA). Cada uno de ellos se distingue del ADN o ARN naturales por los cambios en la estructura principal de la molécula. Sin embargo, la teoría polielectrolítica del gen propone que una molécula genética requiere una estructura principal cargada para funcionar.
En mayo de 2014, los investigadores anunciaron que habían introducido con éxito dos nuevos nucleótidos artificiales en el ADN bacteriano y que, al incluir nucleótidos artificiales individuales en el medio de cultivo, habían logrado que la bacteria pasara 24 veces; no crearon ARNm ni proteínas capaces de utilizar los nucleótidos artificiales. Los nucleótidos artificiales presentaban dos anillos aromáticos fusionados.
Varios análogos de nucleósidos se utilizan como agentes antivirales o anticancerígenos. La polimerasa viral incorpora estos compuestos con bases no canónicas. Estos compuestos se activan en las células al convertirse en nucleótidos y se administran como nucleósidos, ya que los nucleótidos cargados no pueden atravesar fácilmente las membranas celulares. [ cita requerida ]

Los análogos de ácidos nucleicos se utilizan en biología molecular para varios propósitos:
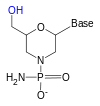
El grupo hidroxi 2' de la ribosa reacciona con el grupo hidroxi 3' unido al fosfato, lo que hace que el ARN sea demasiado inestable para ser utilizado o sintetizado de manera confiable. Para superar esto, se puede utilizar un análogo de la ribosa. Los análogos de ARN más comunes son el ARN 2'-O-metil-sustituido, el ácido nucleico bloqueado (LNA) o el ácido nucleico puenteado (BNA), el morfolino [5] [ 6] y el ácido nucleico peptídico ( PNA ). Aunque estos oligonucleótidos tienen un azúcar de cadena principal diferente (o, en el caso del PNA, un residuo de aminoácido en lugar del fosfato de ribosa), aún se unen al ARN o al ADN según el emparejamiento de Watson y Crick mientras son inmunes a la actividad de las nucleasas. No se pueden sintetizar enzimáticamente y solo se pueden obtener sintéticamente utilizando la estrategia de la fosforamidita o, para el PNA, otros métodos de síntesis de péptidos . [ cita requerida ]
Los didesoxinucleótidos se utilizan en la secuenciación . Estos trifosfatos de nucleósidos poseen un azúcar no canónico, la didesoxirribosa, que carece del grupo hidroxilo 3' normalmente presente en el ADN y, por lo tanto, no puede unirse con la siguiente base. La falta del grupo hidroxilo 3' termina la reacción en cadena, ya que las polimerasas del ADN lo confunden con un desoxirribonucleótido normal. Otro análogo de terminación de cadena que carece de un hidroxilo 3' e imita a la adenosina se llama cordicepina . La cordicepina es un fármaco contra el cáncer que tiene como objetivo la replicación del ARN . Otro análogo en la secuenciación es un análogo de nucleobase, 7-deaza-GTP, y se utiliza para secuenciar regiones ricas en CG; en cambio, la 7-deaza-ATP se llama tubercidina, un antibiótico. [ cita requerida ]
Se ha sugerido que el mundo del ARN puede haber sido precedido por un "mundo similar al ARN" donde existían otros ácidos nucleicos con una estructura diferente, como GNA , PNA y TNA , sin embargo, la evidencia de esta hipótesis se ha calificado de "tenue". [7]
Las bases naturales se pueden dividir en dos clases según su estructura:
Se han insertado nucleótidos artificiales ( pares de bases no naturales (UBP) denominados d5SICS UBP y dNaM UBP ) en el ADN bacteriano, pero estos genes no sirvieron de modelo para el ARNm ni indujeron la síntesis de proteínas. Los nucleótidos artificiales presentaban dos anillos aromáticos fusionados que formaban un complejo (d5SICS–dNaM) que imitaba el par de bases natural (dG–dC). [8] [9] [10]
Uno de los análogos de base más comunes es el 5-bromouracilo (5BU), la base anormal que se encuentra en el análogo de nucleótido mutagénico BrdU. Cuando un nucleótido que contiene 5-bromouracilo se incorpora al ADN, es más probable que se aparee con la adenina; sin embargo, puede cambiar espontáneamente a otro isómero que se aparea con una nucleobase diferente , la guanina . Si esto sucede durante la replicación del ADN, se insertará una guanina como el análogo de base opuesto y, en la siguiente replicación del ADN, esa guanina se apareará con una citosina. Esto da como resultado un cambio en un par de bases del ADN, específicamente una mutación de transición . [ cita requerida ]
Además, el ácido nitroso (HNO2) es un potente mutágeno que actúa sobre el ADN replicante y no replicante. Puede causar la desaminación de los grupos amino de la adenina, la guanina y la citosina. La adenina se desamina a hipoxantina , que se empareja con la citosina en lugar de la timina. La citosina se desamina a uracilo, que se empareja con la adenina en lugar de la guanina. La desaminación de la guanina no es mutagénica. Las mutaciones inducidas por el ácido nitroso también se inducen a mutar de nuevo al tipo salvaje. [ cita requerida ]

Comúnmente, los fluoróforos (como la rodamina o la fluoresceína ) se unen al anillo unido al azúcar (en para) a través de un brazo flexible, que presumiblemente sale del surco mayor de la hélice. Debido a la baja procesividad de los nucleótidos unidos a aductos voluminosos como los floróforos por las [Taq polimerasas], la secuencia se copia típicamente utilizando un nucleótido con un brazo y luego se acopla con un fluoróforo reactivo (marcaje indirecto):
Los fluoróforos encuentran una variedad de usos en la medicina y la bioquímica.
El análogo de base fluorescente más comúnmente utilizado y disponible comercialmente, la 2-aminopurina (2-AP), tiene un alto rendimiento cuántico de fluorescencia libre en solución (0,68) que se reduce considerablemente (aproximadamente 100 veces, pero altamente dependiente de la secuencia de bases) cuando se incorpora a ácidos nucleicos. [11] La sensibilidad de emisión de 2-AP al entorno inmediato es compartida por otros análogos de base fluorescentes prometedores y útiles como 3-MI, 6-MI, 6-MAP, [12] pirrolo-dC (también disponible comercialmente), [13] derivados modificados y mejorados de pirrolo-dC, [14] bases modificadas con furano [15] y muchas otras (ver revisiones recientes). [16] [17] [ 18] [19] [20] Esta sensibilidad al microambiente se ha utilizado en estudios de, por ejemplo, estructura y dinámica dentro del ADN y el ARN, dinámica y cinética de la interacción ADN-proteína y transferencia de electrones dentro del ADN. [ cita requerida ]
Un grupo recientemente desarrollado y muy interesante de análogos de bases fluorescentes que tiene un rendimiento cuántico de fluorescencia que es casi insensible a su entorno inmediato es la familia de citosinas tricíclicas. 1,3-Diaza-2-oxofenotiazina, tC, tiene un rendimiento cuántico de fluorescencia de aproximadamente 0,2 tanto en cadenas simples como en cadenas dobles, independientemente de las bases circundantes. [21] [22] También el oxo-homólogo de tC llamado tC O (ambos disponibles comercialmente), 1,3-diaza-2-oxofenoxazina, tiene un rendimiento cuántico de 0,2 en sistemas de doble cadena. [23] Sin embargo, es algo sensible a las bases circundantes en cadenas simples (rendimientos cuánticos de 0,14-0,41). Los altos y estables rendimientos cuánticos de estos análogos de base los hacen muy brillantes y, en combinación con sus buenas propiedades de análogo de base (dejan la estructura y estabilidad del ADN casi inalteradas), son especialmente útiles en anisotropía de fluorescencia y mediciones de FRET, áreas donde otros análogos de base fluorescentes son menos precisos. Además, en la misma familia de análogos de citosina, se ha desarrollado un análogo de base aceptor de FRET, tC nitro . [24] Junto con tC O como donante de FRET, esto constituye el primer par FRET de análogo de base de ácido nucleico jamás desarrollado. La familia tC se ha utilizado, por ejemplo, en estudios relacionados con la unión del ADN de la polimerasa y los mecanismos de polimerización del ADN.
En una célula, hay varias bases no canónicas presentes: islas CpG en el ADN (a menudo metiladas), todos los ARNm eucariotas (protegidos con una metil-7-guanosina) y varias bases de ARNr (metiladas). A menudo, los ARNt se modifican en gran medida postranscripcionalmente para mejorar su conformación o apareamiento de bases, en particular en o cerca del anticodón: la inosina puede aparearse con C, U e incluso con A, mientras que la tiouridina (con A) es más específica que el uracilo (con una purina). [25] Otras modificaciones de bases de ARNt comunes son la pseudouridina (que da su nombre al bucle TΨC ), la dihidrouridina (que no se apila porque no es aromática), la queuosina, la wyosina, etc. Sin embargo, todas estas son modificaciones de bases normales y no las coloca una polimerasa. [25]
Las bases canónicas pueden tener un grupo carbonilo o un grupo amina en los carbonos que rodean al átomo de nitrógeno más alejado del enlace glucosídico, lo que les permite aparearse ( emparejamiento de bases Watson-Crick) a través de enlaces de hidrógeno (amina con cetona, purina con pirimidina). La adenina y la 2-aminoadenina tienen uno o dos grupos amina, mientras que la timina tiene dos grupos carbonilo, y la citosina y la guanina son aminas y carbonilos mixtos (invertidos entre sí). [ cita requerida ]
La razón precisa por la que solo hay cuatro nucleótidos es objeto de debate, pero hay varias posibilidades que no se utilizan. Además, la adenina no es la opción más estable para el emparejamiento de bases: en Cyanophage S-2L, se utiliza diaminopurina (DAP) en lugar de adenina. [26] La diaminopurina se empareja perfectamente con la timina, ya que es idéntica a la adenina, pero tiene un grupo amino en la posición 2 que forma 3 enlaces de hidrógeno intramoleculares, lo que elimina la principal diferencia entre los dos tipos de pares de bases (AT débil frente a CG fuerte). Esta estabilidad mejorada afecta a las interacciones de unión a proteínas que dependen de esas diferencias. Otras combinaciones incluyen:
Sin embargo, la estructura correcta del ADN puede formarse incluso cuando las bases no están emparejadas a través de enlaces de hidrógeno; es decir, las bases se emparejan gracias a la hidrofobicidad, como han demostrado los estudios con isósteros de ADN (análogos con el mismo número de átomos) como el análogo de timina 2,4-difluorotolueno (F) o el análogo de adenina 4-metilbencimidazol (Z). [28] Un par hidrofóbico alternativo podría ser la isoquinolina y la pirrolo[2,3-b]piridina [29]
Otros pares de bases dignos de mención:
En el apareamiento de bases metálicas, los enlaces de hidrógeno de Watson-Crick se reemplazan por la interacción entre un ion metálico con nucleósidos que actúan como ligandos. Las posibles geometrías del metal que permitirían la formación de dúplex con dos nucleósidos bidentados alrededor de un átomo metálico central son tetraédrica , dodecaédrica y plana cuadrada . La formación de complejos de metales con ADN puede ocurrir por la formación de pares de bases no canónicos a partir de nucleobases naturales con participación de iones metálicos y también por el intercambio de los átomos de hidrógeno que forman parte del apareamiento de bases de Watson-Crick por iones metálicos. [33] Se ha demostrado que la introducción de iones metálicos en un dúplex de ADN tiene propiedades magnéticas [34] o conductoras potenciales, [35] así como una mayor estabilidad. [36]
Se ha demostrado que la formación de complejos metálicos ocurre entre nucleobases naturales . Un ejemplo bien documentado es la formación de T-Hg-T, que involucra dos nucleobases de timina desprotonadas que se unen mediante Hg 2+ y forman un par de base metálica conectado. [37] Este motivo no da cabida a Hg 2+ apilado en un dúplex debido a un proceso de formación de horquilla intracatenaria que se favorece sobre la formación de dúplex. [38] Dos timinas una frente a la otra no forman un par de bases Watson-Crick en un dúplex; este es un ejemplo en el que un desajuste de pares de bases Watson-Crick se estabiliza mediante la formación del par de base metálica. Otro ejemplo de formación de complejos metálicos con nucleobases naturales es la formación de A-Zn-T y G-Zn-C a pH alto; Co 2+ y Ni 2+ también forman estos complejos. Estos son pares de bases Watson-Crick en los que el catión divalente está coordinado con las nucleobases. La unión exacta es motivo de debate. [39]
Se ha desarrollado una gran variedad de nucleobases artificiales para su uso como pares de bases metálicas. Estas nucleobases modificadas exhiben propiedades electrónicas ajustables, tamaños y afinidades de enlace que se pueden optimizar para un metal específico. Por ejemplo, se ha demostrado que un nucleósido modificado con un piridina-2,6-dicarboxilato se une firmemente a Cu 2+ , mientras que otros iones divalentes solo se unen de manera débil. El carácter tridentado contribuye a esta selectividad. El cuarto sitio de coordinación en el cobre está saturado por una nucleobase de piridina dispuesta de manera opuesta. [40] El sistema de apareamiento de bases metálicas asimétrico es ortogonal a los pares de bases de Watson-Crick. Otro ejemplo de una nucleobase artificial es aquella con nucleobases de hidroxipiridona, que pueden unirse a Cu 2+ dentro del dúplex de ADN. Se incorporaron cinco pares de bases de cobre-hidroxipiridona consecutivos en una doble cadena, que estaban flanqueados por una sola nucleobase natural en ambos extremos. Los datos de EPR mostraron que la distancia entre los centros de cobre se estimó en 3,7 ± 0,1 Å, mientras que un dúplex de ADN de tipo B natural es solo ligeramente más grande (3,4 Å). [41] El atractivo de apilar iones metálicos dentro de un dúplex de ADN es la esperanza de obtener cables metálicos autoensamblables nanoscópicos, aunque esto aún no se ha hecho realidad.
Un par de bases no naturales (UBP) es una subunidad diseñada (o nucleobase ) de ADN que se crea en un laboratorio y no ocurre en la naturaleza. En 2012, un grupo de científicos estadounidenses dirigido por Floyd Romesberg, un biólogo químico del Instituto de Investigación Scripps en San Diego, California, publicó que su equipo había diseñado dos pares de bases no naturales llamados d5SICS y dNaM . [42] Más técnicamente, estos nucleótidos artificiales que llevan nucleobases hidrófobas presentan dos anillos aromáticos fusionados que forman un complejo o par de bases d5SICS–dNaM en el ADN. [10] [43] En 2014, el mismo equipo informó que habían sintetizado un plásmido que contenía pares de bases TA y CG naturales junto con el UBP de mejor rendimiento que el laboratorio de Romesberg había diseñado y lo había insertado en células de la bacteria común E. coli , que replicó con éxito los pares de bases no naturales a través de múltiples generaciones. [44] Este es el primer ejemplo conocido de un organismo vivo que transmite un código genético ampliado a generaciones posteriores. [10] [45] Esto se logró en parte mediante la adición de un gen de alga de apoyo que expresa un transportador de trifosfato de nucleótidos que importa de manera eficiente los trifosfatos de d5SICSTP y dNaMTP a las bacterias E. coli . [10] Luego, las vías de replicación bacterianas naturales los utilizan para replicar con precisión el plásmido que contiene d5SICS–dNaM. [ cita requerida ]
La incorporación exitosa de un tercer par de bases es un avance significativo hacia el objetivo de expandir en gran medida el número de aminoácidos que pueden ser codificados por el ADN, de los 20 aminoácidos existentes a 172 teóricamente posibles, expandiendo así el potencial de los organismos vivos para producir nuevas proteínas . [44] Anteriormente, las cadenas artificiales de ADN no codificaban nada, pero los científicos especularon que podrían diseñarse para fabricar nuevas proteínas que podrían tener usos industriales o farmacéuticos. [46] La transcripción de ADN que contiene pares de bases no naturales y la traducción del ARNm correspondiente se lograron recientemente. En noviembre de 2017, el mismo equipo del Instituto de Investigación Scripps que introdujo por primera vez dos nucleobases adicionales en el ADN bacteriano informó haber construido una bacteria E. coli semisintética capaz de producir proteínas utilizando dicho ADN. Su ADN contenía seis nucleobases diferentes : cuatro canónicas y dos añadidas artificialmente, dNaM y dTPT3 (estas dos forman un par). Las bacterias tenían dos bases de ARN correspondientes incluidas en dos nuevos codones, ARNt adicionales que reconocían estos nuevos codones (estos ARNt también contenían dos nuevas bases de ARN dentro de sus anticodones) y aminoácidos adicionales, lo que permitía a las bacterias sintetizar proteínas "no naturales". [47] [48]
Otra demostración de UBP fue lograda por el grupo de Ichiro Hirao en el instituto RIKEN en Japón. En 2002, desarrollaron un par de bases no natural entre 2-amino-8-(2-tienil)purina (s) y piridina-2-ona (y) que funciona in vitro en la transcripción y traducción, para la incorporación específica del sitio de aminoácidos no estándar en proteínas. [49] En 2006, crearon 7-(2-tienil)imidazo[4,5-b]piridina (Ds) y pirrol-2-carbaldehído (Pa) como un tercer par de bases para la replicación y la transcripción. [50] Posteriormente, se descubrió que Ds y 4-[3-(6-aminohexanamido)-1-propinil]-2-nitropirrol (Px) eran un par de alta fidelidad en la amplificación por PCR. [51] [52] En 2013, aplicaron el par Ds-Px a la generación de aptámeros de ADN mediante selección in vitro (SELEX) y demostraron que la expansión del alfabeto genético aumenta significativamente las afinidades de los aptámeros de ADN con las proteínas objetivo. [53]
Se ha propuesto y estudiado, tanto teórica como experimentalmente, la posibilidad de implementar un sistema ortogonal en el interior de las células independientemente del material genético celular para conseguir un sistema totalmente seguro [54] , con el posible aumento de los potenciales de codificación [55] . Varios grupos se han centrado en diferentes aspectos: