El ADN ribosómico (abreviado como ADNr ) consiste en un grupo de genes que codifican ARN ribosómico y elementos reguladores relacionados, y está ampliamente distribuido en una configuración similar en todos los dominios de la vida. El ADN ribosómico codifica el ARN ribosómico no codificante , elementos estructurales integrales en el ensamblaje de los ribosomas , su importancia lo convierte en la sección más abundante de ARN que se encuentra en las células de los eucariotas . [1] Además, estos segmentos incluyen secciones reguladoras , como un promotor específico de la ARN polimerasa I , así como segmentos espaciadores transcritos y no transcritos .
Debido a su gran importancia en el ensamblaje de los ribosomas para la biosíntesis de proteínas , los genes del ADNr están generalmente muy conservados en la evolución molecular . El número de copias puede variar considerablemente según la especie. [1] El ADN ribosómico se utiliza ampliamente para estudios filogenéticos . [2] [3]
El ADN ribosómico incluye todos los genes que codifican las moléculas de ARN ribosómico estructural no codificante . En todos los dominios de la vida, estas son las secuencias estructurales de la subunidad pequeña ( ARNr 16S o 18S ) y la subunidad grande ( ARNr 23S o 28S ). El ensamblaje de esta última también incluye el ARNr 5S, así como el ARNr 5.8S adicional en eucariotas.
Los genes de ADNr suelen estar presentes en múltiples copias en el genoma, donde se organizan en grupos enlazados en la mayoría de las especies, separados por un espaciador transcrito interno (ITS) y precedidos por el espaciador transcrito externo (ETS) . El ARNr 5S también está enlazado a estas regiones de ADNr en procariotas, mientras que se encuentra en regiones repetitivas separadas en la mayoría de los eucariotas. [4] Se transcriben juntos a un ARN precursor que luego se procesa para obtener cantidades iguales de cada ARNr.
Las moléculas de ARNr estructural primario en bacterias y arqueas son más pequeñas que sus contrapartes en eucariotas, agrupadas como ARNr 16S y ARNr 23S . Mientras tanto, el ARNr 5S también presente en procariotas, es de un tamaño similar al de los eucariotas.
Una cantidad notable de bacterias y arqueas divergen de la estructura canónica del operón que contiene los genes del ADNr, llevando los genes "no enlazados" en diferentes lugares de su genoma. [5]
El ADN ribosómico en los cloroplastos sigue la estructura del ADN ribosómico procariota.

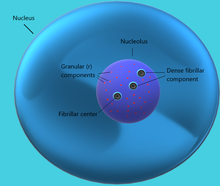
El grupo de genes del ADNr de los eucariotas consta de los genes para el ARNr 18S , 5.8S y 28S , separados por los dos espaciadores ITS-1 e ITS-2 . El genoma activo de los eucariotas contiene varios cientos de copias de la unidad transcripcional del ADNr como repeticiones en tándem , que están organizadas en regiones organizadoras del nucléolo (NOR) , [4] que a su vez pueden estar presentes en múltiples loci en el genoma. [6]
De manera similar a la estructura de los procariotas, el ARNr 5S se agrega al grupo de ADNr en los Saccharomycetes (Hemiascomycetes) [6] como Saccharomyces cerevisiae . [4] Sin embargo, la mayoría de los eucariotas llevan el gen del ARNr 5S en repeticiones genéticas separadas en diferentes loci del genoma. [6] [4]
El ADNr 5S también está presente en repeticiones en tándem independientes como en Drosophila . [6] Como las regiones de ADN repetitivas a menudo experimentan eventos de recombinación, las repeticiones de ADNr tienen muchos mecanismos reguladores que evitan que el ADN sufra mutaciones, [ ejemplo necesario ] manteniendo así el ADNr conservado. [1]
En el núcleo, las regiones organizadoras del nucléolo dan lugar al nucléolo , donde las regiones de ADNr del cromosoma forman bucles cromosómicos expandidos, accesibles para la transcripción del ARNr . En el ADNr, las repeticiones en tándem se encuentran principalmente en el nucléolo; pero el ADNr heterocromático se encuentra fuera del nucléolo. Sin embargo, el ADNr transcripcionalmente activo reside dentro del propio nucléolo. [1]
El genoma humano contiene un total de 560 copias [4] de la unidad transcripcional del ADNr, distribuidas en cinco cromosomas con regiones organizadoras del nucléolo . Los grupos de repeticiones se encuentran en los cromosomas acrocéntricos 13 ( RNR1 ), 14 ( RNR2 ), 15 ( RNR3 ), 21 ( RNR4 ) y 22 ( RNR5 ). [7]
En los ciliados , la presencia de un micronúcleo generativo junto al macronúcleo vegetativo permite la reducción de los genes de ADNr en la línea germinal. El número exacto de copias en el genoma central del micronúcleo varía desde varias copias en Paramecium [8] hasta una sola copia en Tetrahymena thermophila [4] y otras especies de Tetrahymena . Durante la formación del macronúcleo , las regiones que contienen los grupos de genes de ADNr se amplifican, lo que aumenta drásticamente la cantidad de plantillas disponibles para la transcripción hasta varios miles de copias. En algunos géneros de ciliados, como Tetrahymena o el género Hypotrich Oxytricha , [8] la fragmentación extensa del ADN amplificado conduce a la formación de microcromosomas, centrados en la unidad transcripcional del ADNr. [8] Se informan procesos similares de Glaucoma chattoni y, en menor medida, de Paramecium . [8]
En la gran matriz de ADNr, los polimorfismos entre las unidades de repetición de ADNr son muy bajos, lo que indica que las matrices en tándem de ADNr están evolucionando a través de una evolución concertada . [6] Sin embargo, el mecanismo de la evolución concertada es imperfecto, de modo que los polimorfismos entre repeticiones dentro de un individuo pueden ocurrir en niveles significativos y pueden confundir los análisis filogenéticos para organismos estrechamente relacionados. [9] [10]
Se compararon entre sí las secuencias repetidas en tándem 5S en varias Drosophila ; el resultado reveló que las inserciones y deleciones ocurrían con frecuencia entre especies y a menudo flanqueadas por secuencias conservadas. [11] Podrían ocurrir por deslizamiento de la cadena recién sintetizada durante la replicación del ADN o por conversión genética. [11]
Los tractos de transcripción del ADNr tienen una baja tasa de polimorfismo entre especies, lo que permite la comparación interespecífica para dilucidar la relación filogenética utilizando solo unos pocos especímenes. Las regiones codificantes del ADNr están altamente conservadas entre especies, pero las regiones ITS son variables debido a inserciones, deleciones y mutaciones puntuales. Entre especies remotas como humanos y ranas, la comparación de secuencias en los tractos ITS no es apropiada. [12] Las secuencias conservadas en las regiones codificantes del ADNr permiten comparaciones de especies remotas, incluso entre levaduras y humanos. El ARNr 5.8S humano tiene un 75% de identidad con el ARNr 5.8S de levadura. [13] En los casos de especies hermanas, la comparación del segmento de ADNr que incluye los tractos ITS entre especies y el análisis filogenético se realizan satisfactoriamente. [14] [15] Las diferentes regiones codificantes de las repeticiones de ADNr generalmente muestran distintas tasas evolutivas. Como resultado, este ADN puede proporcionar información filogenética de especies que pertenecen a amplios niveles sistemáticos. [2]
Un fragmento de ADNr de levadura que contiene el gen 5S, ADN espaciador no transcrito y parte del gen 35S tiene actividad estimuladora de recombinación mitótica localizada que actúa en cis. [16] Este fragmento de ADN contiene un punto caliente de recombinación mitótica , denominado HOT1. HOT1 expresa actividad estimulante de la recombinación cuando se inserta en nuevas ubicaciones en el genoma de la levadura . HOT1 incluye un promotor de transcripción de la ARN polimerasa I (PolI) que cataliza la transcripción del gen ARNr ribosomal 35S . En un mutante defectuoso de PolI, la actividad estimulante de la recombinación del punto caliente HOT1 se elimina. El nivel de transcripción de PolI en HOT1 parece determinar el nivel de recombinación . [17]
Las enfermedades pueden estar asociadas a mutaciones del ADN donde el ADN puede expandirse, como la enfermedad de Huntington , o perderse debido a mutaciones por deleción. Lo mismo es cierto para las mutaciones que ocurren en las repeticiones del ADNr; se ha encontrado que si los genes que están asociados con la síntesis de ribosomas se interrumpen o mutan, puede dar lugar a varias enfermedades asociadas con el esqueleto o la médula ósea. Además, cualquier daño o interrupción de las enzimas que protegen las repeticiones en tándem del ADNr, puede dar lugar a una menor síntesis de ribosomas, lo que también conduce a otros defectos en la célula. Las enfermedades neurológicas también pueden surgir de mutaciones en las repeticiones en tándem del ADNr, como el síndrome de Bloom , que ocurre cuando el número de repeticiones en tándem aumenta cerca de cien veces; en comparación con el del número normal de repeticiones en tándem. Varios tipos de cánceres también pueden nacer de mutaciones de las repeticiones en tándem en el ADN ribosómico. Las líneas celulares pueden volverse malignas debido a un reordenamiento de las repeticiones en tándem o a una expansión de las repeticiones en el ADNr. [18]