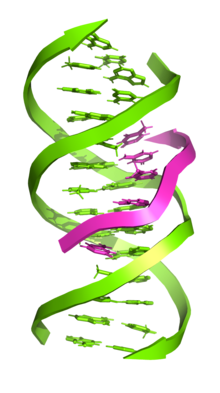
El ADN tricótico (también conocido como ADN-H o ADN-Tríplex ) es una estructura de ADN en la que tres oligonucleótidos se enrollan entre sí y forman una triple hélice . En el ADN tricótico, la tercera hebra se une a una doble hélice de ADN en forma B (a través del apareamiento de bases Watson-Crick ) mediante la formación de pares de bases de Hoogsteen o enlaces de hidrógeno de Hoogsteen invertidos.
Se han descrito ejemplos de ADN de triple cadena de fuentes naturales con la combinación necesaria de composición de bases y elementos estructurales, por ejemplo en Satellite DNA . [1]
Una nucleobase de timina (T) puede unirse a un apareamiento de bases Watson-Crick de TA mediante la formación de un enlace de hidrógeno de Hoogsteen . El enlace de hidrógeno de timina con la adenosina (A) del ADN bicatenario original crea un triplete de bases TA*T. [2]
[3]

Existen dos clases de ADN tríplex: formaciones intermoleculares e intramoleculares. Un tríplex intermolecular se refiere a la formación de tríplex entre un dúplex y una tercera hebra de ADN diferente. La tercera hebra puede ser de un cromosoma vecino o de un oligonucleótido formador de tríplex (TFO). El ADN tríplex intramolecular se forma a partir de un dúplex con hebras de homopurina y homopirimidina con simetría de repetición especular. [4] El grado de superenrollamiento en el ADN influye en la cantidad de formación de tríplex intramolecular que ocurre. [5] Existen dos tipos diferentes de ADN tríplex intramolecular: ADN-H y ADN-H*. La formación de ADN-H se estabiliza en condiciones ácidas y en presencia de cationes divalentes como Mg 2+ . En esta conformación, la hebra de homopirimidina en el dúplex se dobla hacia atrás para unirse a la hebra de purina de manera paralela. Las tríadas de bases que se utilizan para estabilizar esta conformación son TA*T y CG*A + . La citosina de esta tríada de bases necesita ser protonada para formar esta triple hélice intramolecular, por lo que esta conformación se estabiliza en condiciones ácidas. [6] El H*-ADN tiene condiciones de formación favorables a pH neutro y en presencia de cationes divalentes. [5] Esta conformación intramolecular se forma a partir de la unión de la cadena de homopurina y purina del dúplex de manera antiparalela. Se estabiliza mediante los tripletes de bases TA*A y CG*G. [4] [6]
Los TFO son cadenas cortas de ácido nucleico (≈15-25 nt) que se unen en el surco mayor del ADN bicatenario para formar estructuras intramoleculares de ADN tríplex. Existe cierta evidencia de que también pueden modular la actividad genética in vivo . En el ácido nucleico peptídico (PNA), la cadena principal de azúcar-fosfato del ADN se reemplaza por una cadena principal similar a la de una proteína. Los PNA forman bucles P mientras interactúan con el ADN dúplex, formando un tríplex con una cadena de ADN mientras desplazan la otra. Se ha asumido que se forman recombinaciones muy inusuales o tríplex paralelos, o ADN-R, bajo la proteína RecA en el curso de la recombinación homóloga. [7]
Los TFO se unen específicamente a las regiones de homopurina-homopirimidina que suelen ser comunes en las secuencias promotoras e intrónicas de los genes, lo que influye en la señalización celular. [8] Los TFO pueden inhibir la transcripción uniéndose con alta especificidad a la hélice de ADN, bloqueando así la unión y la función de los factores de transcripción para secuencias particulares. Al introducir TFO en una célula (a través de transfección u otros medios), se puede controlar la expresión de ciertos genes. [9] Esta aplicación tiene implicaciones novedosas en la mutagénesis específica del sitio y la terapia génica. En las células de cáncer de próstata humano, se sobreexpresa un factor de transcripción Ets2 y se cree que impulsa el crecimiento y la supervivencia de las células en tal exceso. Carbone et al. diseñaron un TFO específico de secuencia para la secuencia promotora Ets2 que reguló a la baja la expresión génica y condujo a una desaceleración del crecimiento celular y la muerte celular. [10] Changxian et al. también han presentado un TFO dirigido a la secuencia promotora de bcl-2, un gen que inhibe la apoptosis. [11]
La inhibición observada de la transcripción también puede tener efectos negativos para la salud, como su papel en el gen autosómico recesivo de la ataxia de Friedreich . [12] En la ataxia de Friedreich, la formación de ADN triple perjudica la expresión del intrón 1 del gen FXN . Esto da como resultado la degeneración del sistema nervioso y la médula espinal, lo que perjudica el movimiento de las extremidades. [13] Para combatir esta inestabilidad triple, se ha demostrado que las proteínas de reparación por escisión de nucleótidos (NER) reconocen y reparan las estructuras de ADN de triple cadena, restableciendo la disponibilidad total del gen previamente inhibido e inestable. [14]

Los ácidos nucleicos peptídicos son oligonucleótidos sintéticos que resisten la degradación de las proteasas y se utilizan para inducir la reparación en regiones de formación de triplex en sitios específicos de los sitios genómicos del ADN. Los PNA pueden unirse con alta afinidad y especificidad de secuencia a una secuencia de ADN complementaria a través de la unión por apareamiento de bases Watson-Crick y pueden formar triples hélices a través de enlaces Hoogsteen de orientación paralela con el PNA orientado hacia el extremo 5' de la cadena de ADN. [15] El triplex PNA-ADN es estable porque los PNA consisten en una cadena principal pseudopeptídica con carga neutra que se une para unirse a la secuencia de ADN bicatenario (dsADN). [16] De manera similar a la homopirimidina en los TFO, la homopirimidina en los PNA puede formar un enlace con la homopurina complementaria en la secuencia diana del dsADN. Estos análogos de ADN pueden unirse al dsADN explotando las condiciones ambientales del ADN y diferentes modos de predicción de reconocimiento. Esto es diferente de los TFO que se unen a través del reconocimiento del surco mayor del dsADN. [15]
Uno de los modos de predicción de reconocimiento utilizados para el reconocimiento es a través de una invasión dúplex. [17] [16] Dentro de la secuencia mixta A–T/G–C dsADN es el objetivo de un par de PNA pseudocomplementarios (pc) que pueden unirse a los dsADN a través de una doble invasión mediante la formación simultánea de diaminopurina (D) y tiouracilo (U s ) que sustituyen a la adenina y la timina, respectivamente. [17] El par pc PNA forma una hélice de PNA-ADN emparejada DT y U s -A y GC o CG Watson-Crick con cada una de las hebras de ADN complementarias. Otra forma de invasión dúplex reconocida en la secuencia objetivo puede ocurrir en dsADN que contiene secuencias mixtas T–C. [18] Esta forma de invasión dúplex se logra a través de una secuencia complementaria de oligómeros de PNA de homopurina. Este triplex se forma a partir de un híbrido PNA-ADN que se une de forma antiparalela a la secuencia de ADN complementaria y da como resultado una cadena de ADN no complementaria desplazada. [16]
Además, el PNA puede modificarse para formar estructuras triplex de “pinza” en el sitio diana. [17] Un tipo de “pinza” formada es una estructura bis-PNA, en la que dos moléculas de PNA se mantienen juntas mediante un conector flexible como el ácido 8-amino-3,6-dioxaoctanoico (O). [19] La estructura bis-PNA forma un triplex PNA-ADN-PNA en el sitio diana, donde una hebra forma pares de bases Watson-Crick con el ADN en una orientación antiparalela y la otra hebra forma pares de bases Hoogsteen con la hebra de ADN de homopurina en el dúplex ADN-PNA. [18] Una pinza de cola de PNA (tcPNA) es también otra forma de pinza triple que también puede formarse. Los tcPNA contienen una cola extendida de 5-10 pb que forma un dúplex PNA/ADN además de una “pinza” PNA-ADN-PNA. Esto permite una unión más específica del PNA sin la necesidad de un tramo de homopirimidia/piridina. [16] Se ha demostrado que estas estructuras de pinza tienen una alta afinidad y especificidad. La adición de residuos de lisina a uno o ambos extremos del PNA podría utilizarse para aumentar la captación y la unión celular. [17]
El ADN de triple cadena se ha visto implicado en la regulación de varios genes. Por ejemplo, el gen c- myc ha sido ampliamente mutado para examinar el papel que desempeña el ADN tríplex, frente a la secuencia lineal, en la regulación génica. Un elemento promotor de c- myc , denominado elemento sensible a la nucleasa o NSE, puede formar tríplex intramoleculares en tándem del tipo H-ADN y tiene un motivo de secuencia repetitiva (ACCCTCCCC) 4 . El NSE mutado se examinó en busca de actividad transcripcional y de su capacidad de formación de tríplex intra e intermolecular. La actividad transcripcional de los NSE mutantes se puede predecir por la capacidad del elemento de formar H-ADN y no por el número de repeticiones, la posición o el número de pares de bases mutantes. Por tanto, el ADN puede ser un participante dinámico en la transcripción del gen c-myc. [20]
Según varios artículos publicados, el H-ADN tiene la capacidad de regular la expresión génica dependiendo de factores como la ubicación y las secuencias cercanas. Aunque las regiones intergénicas del genoma procariota han mostrado pocos rastros de H-ADN natural o motivos triplex, las estructuras de H-ADN han demostrado ser más frecuentes en el genoma eucariota. Se ha demostrado que el H-ADN es especialmente abundante en células de mamíferos, incluidos los humanos (1 de cada 50.000 pb). [15] Las secuencias genéticas implicadas en la regulación génica se encuentran normalmente en las regiones promotoras del genoma eucariota. [15]
En consecuencia, la región promotora ha mostrado la capacidad de formar H-ADN con una frecuencia más alta. [15] Un análisis bioinformático del genoma de S. cerevisiae observó la aparición de H-ADN y otros motivos de ADN triplete en cuatro regiones organizativas: intrones, exones, regiones promotoras y regiones misceláneas. La bioinformática mostró un total de 148 posibles estructuras de H-ADN o ADN triplete. La región promotora representó la frecuencia más alta con 71 estructuras tripletes, mientras que los exones representaron 57 estructuras tripletes y los intrones y misceláneos representaron 2 y 18 estructuras. [21]
Estudios in vitro e in vivo de la expresión del genoma eucariota dieron como resultado uno de tres resultados: regulación positiva, regulación negativa o ningún cambio en la presencia de motivos de H-ADN. [15] Kato et al. informaron sobre la expresión positiva de lacZ cuando se introdujo H-ADN en el promotor de B-lactamasa . [22] [15] Por otro lado, un estudio similar (Brachmachari et al. ) no informó ninguna inhibición estadísticamente significativa del gen reportero lacZ cuando se insertó H-ADN en el genoma de células COS de mamíferos. [15] Aunque los estudios sugieren la regulación de H-ADN, el mecanismo aún está bajo investigación. Potaman et al . asocia el mecanismo de regulación génica a las interacciones entre el H-ADN y la caja TATA que se encuentra en la región promotora de Na,K-ATPasa . En las formaciones de H-ADN adyacentes a una caja TATA, la estructura de H-ADN desestabiliza los enlaces TA esenciales para la transcripción. La interferencia con la caja TATA inhibe la maquinaria transcripcional y la iniciación de la transcripción, lo que interfiere con la expresión génica. [15] [23] Otros mecanismos asociados con la expresión genómica de una secuencia genética en presencia de H-ADN involucran a los TFO. Estudios in vitro han resaltado una disminución en la expresión génica en presencia de TFO en células de mamíferos. [24] Otro posible mecanismo presentado por Valentina et al. sugiere que el complejo triplex de oligonucleótidos con motivo AG de 13 meros (complejo TFO) regula negativamente la transcripción de ARNm a través de inhibición competitiva. [25] La inhibición directa de la expresión génica del H-ADN es clave para la mutagénesis, inhibición de la replicación e incluso recombinación de ADN en el genoma. [15]

Se ha demostrado que los motivos H-ADN estimulan la recombinación homóloga con diferentes mecanismos. Las implicaciones iniciales para el papel del H-ADN en la recombinación llegaron a principios de la década de 1990 cuando se observó RecA , una proteína de recombinación de ADN bacteriano compuesta de ADN de triple hélice. RecA exhibe una actividad enzimática esencial para la recombinación. [7] [26] También se ha encontrado recombinación homóloga que involucra motivos H-ADN en eucariotas. Se ha demostrado que RadA, una proteína homóloga a RecA, tiene la misma actividad enzimática en la recombinación que RecA. [27] La proteína tiene la capacidad de promover e intercambiar hebras homólogas a través de hélices de triple hebra paralelas. [28] [29] El ADN monocatenario (ssADN) y el ADN bicatenario complementario (dsADN) formarán una estructura de bucle D. [30] [15] Otro mecanismo posible para RecA involucra el ssDNA de dos estructuras H-DNA separadas para formar pares de bases Watson-Crick. La nueva estructura se conoce como unión Holliday, un intermediario en la recombinación homóloga. [15] El H-DNA también se encuentra en otras formas de recombinación. En células de mamíferos, las secuencias H-DNA mostraron una alta frecuencia de recombinación. Por ejemplo, un estudio realizado en la línea celular de mieloma de ratones encontró estructuras H-DNA en Cγ2a y Cγ2b, que participan en el intercambio de cromátidas hermanas. [15]
Se han realizado numerosas investigaciones sobre las implicaciones biológicas relacionadas con la presencia de ADN-H en las regiones de puntos de ruptura principales (Mbr) y en los puntos de ruptura de doble cadena de ciertos genes. Trabajos recientes han vinculado la presencia de estructuras que no son de ADN-B con casos de inestabilidad genética. [31]
Se encontraron secuencias formadoras de H-ADN con repetición en espejo de polipurina vecinas al promotor P1 del gen c-MYC y están asociadas con los principales puntos de ruptura de esta región. También se observaron casos de inestabilidad genética en la descendencia F1 de ratones transgénicos después de la incorporación de secuencias formadoras de H-ADN humanas emparejadas con secuencias de Z-ADN en sus genomas donde no se había informado previamente de inestabilidad. [32] Además, se ha observado la formación de conformaciones de H-ADN R. RY en el Mbr del gen bcl-2 . Se ha postulado que la formación de estas estructuras causa la translocación t(14;18) observada en muchos cánceres y la mayoría de los linfomas foliculares . Esta observación ha llevado a una investigación que indicó que se puede observar una disminución sustancial en los eventos de translocación después de bloquear la formación de H-ADN alterando ligeramente la secuencia de esta región. [32] [33] También se ha observado que largos tramos de GAA·TTC forman estructuras de H-ADN muy estables. Se ha demostrado que las interacciones entre estas dos estructuras de ADN-H, denominadas ADN pegajoso, interrumpen la transcripción del gen X25 o de la frataxina . Como la disminución de los niveles de la proteína frataxina está asociada con la ataxia de Friedreich , se ha sugerido que la formación de esta inestabilidad es la base de esta enfermedad genética. [34] [35] Se ha observado ADN de triple cadena en el ADN satélite superenrollado en regiones donde los números de copias de microsatélites son muy variables, junto con estructuras de ADN-Z de repetición invertida dentro de una unidad de repetición de ADN satélite más grande de 2,1 kb. [36]
Además, se ha demostrado que el H-ADN causa mutaciones relacionadas con procesos celulares críticos como la replicación y transcripción del ADN . [37] La importancia de estos procesos para la supervivencia ha llevado al desarrollo de mecanismos complejos de reparación del ADN que permiten a las células reconocer y reparar el daño del ADN. Las estructuras de ADN no canónicas pueden ser percibidas como daño por la célula, y trabajos recientes han demostrado una mayor prevalencia de mutaciones cerca de secuencias que no forman B-ADN. [37] Algunas de estas mutaciones se deben a las interacciones entre el H-ADN y las enzimas involucradas en la replicación y transcripción del ADN, donde el H-ADN interfiere con estos procesos y desencadena varios mecanismos de reparación del ADN. Esto puede causar inestabilidad genética e implica al H-ADN en la formación del cáncer. [37]
Se ha demostrado que la replicación del ADN afecta la función de varias enzimas de reparación del ADN. La formación de H-ADN implica la formación de ADN monocatenario (ssADN), que es más susceptible al ataque de las nucleasas . [37] Se ha demostrado que varias nucleasas interactúan con H-ADN de una manera dependiente o independiente de la replicación. [37]
Un estudio con células humanas descubrió que las nucleasas de reparación por escisión de nucleótidos (NER) ERCC1-XPF y ERCC1-XPG inducían inestabilidad genética. [38] Estas enzimas escinden el ADN-H en el bucle formado por las dos hebras unidas por hidrógeno de Hoogsteen y el extremo 5' de la otra hebra unida por hidrógeno de Watson-Crick, respectivamente. [38] Se ha demostrado que esta escisión induce grandes deleciones que causan roturas de doble cadena (DSB) en el ADN que pueden conducir a inestabilidad genética. [37] [38] En células deficientes en ERCC1-XPF y ERCC1-XPG, estas deleciones fueron menos frecuentes cerca de las secuencias formadoras de ADN-H. [38] Además, se encontraron más mutaciones en células deficientes en ERCC1-XPF y ERCC1-XPG en ausencia de replicación de ADN, lo que sugiere que procesan el ADN-H de una manera independiente de la replicación. [38]
Como alternativa, se descubrió que la nucleasa de reparación de replicación de ADN FEN1 suprime la inestabilidad genética. [38] De manera similar a ERCC1-XPG, FEN1 corta el H-ADN en el extremo 5' de la cadena que no está involucrada en la unión de hidrógeno de Hoogsteen. [38] Las células HeLa deficientes en FEN1 mostraron una mayor prevalencia de deleciones cerca de las secuencias formadoras de H-ADN, pero la mutagénesis inducida por H-ADN fue más pronunciada en las células deficientes en FEN1 en presencia de replicación de ADN. [38] Esto sugiere que FEN1 suprime la mutagénesis inducida por H-ADN de una manera dependiente de la replicación. [38]
El H-ADN se ha visto implicado en la etiología del cáncer humano debido a la prevalencia de secuencias formadoras de H-ADN cerca de los puntos de corte de translocación en los genomas del cáncer. [38] La actividad de nucleasa mediada por replicación con H-ADN resalta otra forma en que la mutagénesis inducida por H-ADN conduce al crecimiento del cáncer.
Las secuencias formadoras de ADN-H también pueden causar inestabilidad genética al interferir con la transcripción y detenerla prematuramente. [37] El desenrollado del ADN involucrado en la transcripción lo hace más susceptible a sufrir daños. En la reparación acoplada a la transcripción (TCR), una lesión en la cadena de ADN molde detiene la función de la ARN polimerasa y envía señales a los factores TCR para que resuelvan el daño extirpándolo. [39] El ADN-H puede percibirse como una de estas lesiones.
Un estudio en el que se observó la transcripción por la ARN polimerasa T7 en un análogo de secuencia estable formador de ADN-H encontró un bloqueo de la transcripción en la unión dúplex-tríplex. En este caso, la cadena molde era la cadena central del ADN-H y la dificultad de interrumpir sus enlaces de hidrógeno Watson-Crick y Hoogsteen impidió que la transcripción avanzara. [40]
Cuando se observó la transcripción por T7 en el promotor P0 del gen c-MYC , los productos de transcripción acortados que se encontraron indicaron que la transcripción se detuvo en las proximidades de la secuencia formadora de H-ADN aguas abajo del promotor. La formación de H-ADN en esta región evita que T7 viaje por la cadena molde debido al impedimento estérico que causa. Esto detiene la transcripción y envía señales a los factores TCR para que resuelvan el H-ADN, lo que da como resultado la escisión del ADN que puede causar inestabilidad genética. [39] La simetría especular y la prevalencia de residuos de guanina en el gen c-MYC le confieren una alta propensión a la formación de estructuras de ADN no canónicas. [41] Esto, junto con la actividad de los factores TCR durante la transcripción, lo hace altamente mutagénico, y desempeña un papel en el desarrollo del linfoma de Burkitt y la leucemia . [39] [41]
Las regiones de ADN tricrónico se pueden generar mediante la asociación de oligonucleótidos formadores de triple cadena (TFO) y ácidos nucleicos peptídicos (PNA). Históricamente, se ha demostrado que la unión de TFO inhibe la transcripción, replicación y unión de proteínas al ADN. [17] También se ha demostrado que los TFO unidos a mutágenos promueven el daño del ADN e inducen mutagénesis. [15] Aunque se sabe que los TFO obstaculizan la transcripción y replicación del ADN, estudios recientes han demostrado que los TFO se pueden utilizar para mediar modificaciones genéticas específicas del sitio tanto in vitro como in vivo . [17] Otro estudio reciente también ha demostrado que los TFO se pueden utilizar para la supresión de oncogenes y protooncogenes para reducir el crecimiento de células cancerosas. Por ejemplo, un estudio reciente ha utilizado TFO para reducir la muerte celular en células de hepatoma mediante la disminución de la expresión de MET.
Los TFO de PNA tienen la capacidad de mejorar las frecuencias de recombinación, lo que conduce a una edición específica y dirigida de genes. La triple hélice PNA-ADN-PNA puede ser reconocida por el propio mecanismo de reparación del ADN de la célula, que sensibiliza al ADN circundante para la recombinación homóloga. Para que una estructura de PNA específica del sitio medie la recombinación dentro de una secuencia de ADN, una estructura bis-PNA se puede acoplar con un fragmento de ADN de 40 nt que sea homólogo a una región adyacente en el gen objetivo. [18] Se ha demostrado que la unión de un TFO a una cadena de ADN donante induce la recombinación del gen objetivo y la región objetivo del gen adyacente. [42] El mecanismo para esta forma de recombinación y reparación se ha vinculado a la vía de reparación por escisión de nucleótidos (NER) que desempeña un papel en el reconocimiento y la reparación de estructuras triples. [18] [17] Varias investigaciones sugieren que el grupo A del xeroderma pigmentosum (XPA) y la proteína de replicación A (RPA), que son factores NER, son capaces de unirse específicamente como un complejo a estructuras triples reticuladas. Se sabe que este mecanismo, junto con otros, desempeña un papel en el reconocimiento y la reparación de estructuras triples.
La administración in vivo de TFO ha sido una barrera importante en el uso de TFO para la modificación genética. [43] Un estudio sobre la focalización in vivo de células madre hematopoyéticas propuso una técnica novedosa de conjugación de moléculas de PNA con péptidos penetrantes celulares (CPP) junto con nanopartículas de ácido poli(láctico-co-glicólico) ( PLGA ) para permitir modificaciones de 6 pb en el gen CCR5 . [42] La edición del gen CCR5 se ha relacionado con la resistencia al VIH-1. [44] Los CPP son proteínas que pueden transportar "carga", como pequeñas proteínas o moléculas, con éxito a las células. Los PGLA son materiales biodegradables que encapsulan moléculas de PNA como nanopartículas para modificaciones genómicas específicas del sitio. [42] El estudio descubrió que las nanopartículas de PGLA de ADN-PNA podían editar eficazmente las células madre hematopoyéticas con menor toxicidad y sin virus, y la conjugación con CPP ofrecía una focalización directa de los genes para la mutagénesis específica del sitio en las células madre.
En un nuevo estudio de terapia génica para la fibrosis quística (FQ), se diseñaron tres ácidos nucleicos peptídicos (PNA) de pinza de cola junto con una molécula de ADN donante para que se administraran mediante nanopartículas para corregir las mutaciones F508 del en el regulador de conductancia transmembrana de la fibrosis quística ( CFTR ) en células epiteliales bronquiales humanas in vivo e in vitro. [45] La mutación F508 del es la mutación que se produce con más frecuencia y que lleva a una persona a tener FQ. [46] La mutación F508 conduce a una pérdida de la función del CFTR, que es un canal de cloruro de la membrana plasmática que está regulado por un monofosfato de adenosina cíclico (cAMP). En este estudio, pudieron crear el nuevo enfoque de tratamiento para la FQ mediante el uso de nanopartículas para corregir la mutación F508 del CFTR tanto in vitro en células epiteliales bronquiales humanas (HBE) como in vivo en un modelo de ratón con FQ que resultó en la aparición de transporte de cloruro dependiente de CFTR. [45]

Las estructuras de ADN de triple cadena eran hipótesis comunes en la década de 1950, cuando los científicos luchaban por descubrir la verdadera forma estructural del ADN. Watson y Crick (que más tarde ganaron el Premio Nobel por su modelo de doble hélice) consideraron originalmente un modelo de triple hélice, al igual que Pauling y Corey , quienes publicaron una propuesta para su modelo de triple hélice en 1953, [47] [48] así como su colega científico Fraser. [49] Sin embargo, Watson y Crick pronto identificaron varios problemas con estos modelos:
El modelo de Fraser se diferenciaba del de Pauling y Corey en que en su modelo los fosfatos se encuentran en el exterior y las bases en el interior, unidos entre sí por enlaces de hidrógeno. Sin embargo, Watson y Crick consideraron que el modelo de Fraser estaba demasiado mal definido como para comentar específicamente sus deficiencias.
En 1957 se describió una estructura de ADN tricótica alternativa. [50] Felsenfeld, Davies y Rich predijeron que si una cadena contenía solo purinas y la otra cadena solo purinas, la cadena sufriría un cambio conformacional para formar una hélice de ADN tricótica. Se predijo que el ADN tricótica (ADN-H) estaría compuesto por una cadena de polipurina y dos cadenas de polipirimidina. [7] [50] Se pensaba que esto ocurría solo en un proceso biológico in vivo : como un producto intermedio durante la acción de la enzima de recombinación RecA de E. coli . [50] Los primeros modelos de la década de 1960 predijeron la formación de complejos entre oligonucleótidos policetilínicos y de guanina. Los modelos sugirieron interacciones conocidas como emparejamiento de Hoogsten (interacciones no Watson-Crick) ubicadas en el surco mayor. [7] Poco después, se predijeron hélices triples compuestas por una cadena de pirimidina y dos de purina. [7] El descubrimiento de tramos de H-ADN en plásmidos superenrollados despertó el interés moderno en la función potencial de las estructuras triplex en células vivas. [51] Además, pronto se descubrió que la homopirimidina y algunos oligonucleótidos ricos en purina pueden formar una estructura de H-ADN estable con las estructuras específicas de la secuencia de unión de homopurina-homopirimidina en los dúplex de ADN. [52]