
El complejo de piruvato deshidrogenasa ( PDC ) es un complejo de tres enzimas que convierte el piruvato en acetil-CoA mediante un proceso llamado descarboxilación de piruvato . [1] Luego, el acetil-CoA puede usarse en el ciclo del ácido cítrico para llevar a cabo la respiración celular , y este complejo vincula la vía metabólica de la glucólisis con el ciclo del ácido cítrico . La descarboxilación del piruvato también se conoce como "reacción de piruvato deshidrogenasa" porque también implica la oxidación del piruvato. [2]
Este complejo multienzimático está relacionado estructural y funcionalmente con los complejos multienzimáticos de oxoglutarato deshidrogenasa y oxoácido deshidrogenasa de cadena ramificada .
La reacción catalizada por el complejo piruvato deshidrogenasa es:

La subunidad E1, llamada subunidad de piruvato deshidrogenasa , es un homodímero (que comprende dos cadenas “ɑ”, por ejemplo en Escherichia coli ) o un heterotetrámero de dos cadenas diferentes (dos cadenas “ɑ” y dos cadenas “ꞵ”). Un ion magnesio forma un complejo de 4 coordenadas con tres residuos de aminoácidos polares (Asp, Asn y Tyr) ubicados en la cadena alfa, y el cofactor difosfato de tiamina (TPP) directamente involucrado en la descarboxilación del piruvato . [3] [4]
La subunidad E2, o dihidrolipoil acetiltransferasa, tanto para procariotas como para eucariotas, generalmente se compone de tres dominios. El dominio N-terminal (el dominio lipoilo) consta de 1 a 3 grupos lipoilo de aproximadamente 80 aminoácidos cada uno. El dominio de unión a la subunidad periférica (PSBD) sirve como sitio de unión selectivo para otros dominios de las subunidades E1 y E3. Finalmente, el dominio C-terminal (catalítico) cataliza la transferencia de grupos acetilo y la síntesis de acetil-CoA. [5] En las gammaproteobacterias, 24 copias de E2 forman el núcleo cúbico del complejo de piruvato deshidrogenasa, en el que 8 homotrímeros de E2 se encuentran en los vértices de la partícula del núcleo cúbico.

La subunidad E3, llamada enzima dihidrolipoil deshidrogenasa , se caracteriza como una proteína homodímera en la que dos residuos de cisteína , involucrados en enlaces disulfuro , y el cofactor FAD en el sitio activo facilitan su propósito principal como catalizador oxidante. Un ejemplo de estructura E3, que se encuentra en Pseudomonas putida , se forma de manera que cada subunidad homodímera individual contiene dos dominios de unión responsables de la unión de FAD y la unión de NAD, así como un dominio central y un dominio de interfaz. [6] [7]
Una proteína auxiliar exclusiva de la mayoría de los eucariotas es la proteína de unión a E3 (E3BP), que sirve para unir la subunidad E3 al complejo PDC. En el caso de la E3BP humana, los residuos hidrófobos de prolina y leucina en la BP interactúan con el sitio de reconocimiento de la superficie formado por la unión de dos monómeros E3 idénticos. [8]
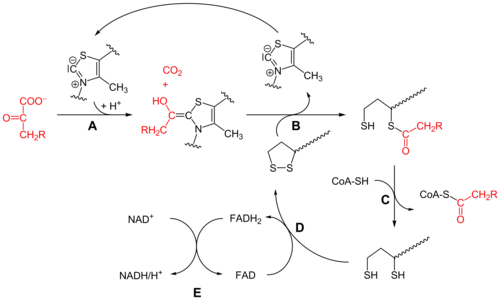
Inicialmente, el piruvato y el pirofosfato de tiamina (TPP o vitamina B 1 ) están unidos por subunidades de piruvato deshidrogenasa . [1] El anillo de tiazolio del TPP está en forma zwitteriónica y el carbono C2 aniónico realiza un ataque nucleofílico al carbonilo C2 (cetona) del piruvato. El intermedio resultante se somete a descarboxilación para producir un equivalente de anión acilo (ver química de cianohidrina o aldehído -ditiano , así como condensación de benjuí ). Este anión ataca al S1 de una especie de lipoato oxidado que está unido a un residuo de lisina . En un mecanismo similar a S N 2 de apertura de anillo, S2 se desplaza como un resto sulfuro o sulfhidrilo. El colapso posterior del intermedio tetraédrico expulsa tiazol, liberando el cofactor TPP y generando un tioacetato en S1 de lipoato. El proceso catalizado por E1 es el paso limitante de la velocidad de todo el complejo de piruvato deshidrogenasa.
En este punto, la funcionalidad lipoato-tioéster se transloca al sitio activo de la dihidrolipoil transacetilasa (E2), [1] donde una reacción de transacilación transfiere el acetilo del "brazo oscilante" del lipoilo al tiol de la coenzima A. Esto produce acetil-CoA , que se libera del complejo enzimático y posteriormente ingresa en el ciclo del ácido cítrico . E2 también puede conocerse como lipoamida reductasa-transacetilasa.
El dihidrolipoato , unido covalentemente a un residuo de lisina del complejo, luego se transfiere al sitio activo de la dihidrolipoil deshidrogenasa (E3), [1] donde sufre una oxidación mediada por flavina , similar en química a, por ejemplo, la tiorredoxina reductasa. Primero, el FAD oxida el dihidrolipoato y lo devuelve a su estado de reposo de lipoato (disulfuro), produciendo FADH 2 . Luego, el sustrato NAD + oxida FADH 2 de nuevo a su estado de reposo FAD, produciendo NADH y H + .
PDC es un gran complejo compuesto por múltiples copias de 3 o 4 subunidades según la especie.
En las bacterias Gram negativas , por ejemplo, Escherichia coli , la PDC consta de un núcleo cúbico central formado por 24 moléculas de dihidrolipoil transacetilasa (E2). Hasta 16 homodímeros de piruvato deshidrogenasa (E1) y 8 homodímeros de dihidrolipoil deshidrogenasa (E3) se unen al PSBD E2 alrededor del núcleo E2. En Gammaproteobacteria , la especificidad de PSBD para unirse a E1 o E3 está determinada por el estado oligomérico de PSBD. En cada homotrímero E2, dos de los tres PSBD se dimerizan. Mientras que dos homodímeros E1 se unen cooperativamente al PSBD dimérico, el PSBD no apareado restante interactúa específicamente con un homodímero E3. La dimerización de PSBD determina así la composición de subunidades del complejo de piruvato deshidrogenasa cuando está completamente saturado con las subunidades periféricas E1 y E3, que tiene una estequiometría de E1:E2:E3 (monómeros) = 32:24:16 [9]
Por el contrario, en las bacterias Gram positivas (por ejemplo, Bacillus stearothermophilus ) y en los eucariotas, el núcleo central del PDC contiene 60 moléculas de E2 dispuestas en un icosaedro. Este “núcleo” de la subunidad E2 coordina 30 subunidades de E1 y 12 copias de E3.
Los eucariotas también contienen 12 copias de una proteína central adicional, la proteína de unión a E3 (E3BP), que une las subunidades E3 al núcleo E2. [10] La ubicación exacta de E3BP no está completamente clara. La microscopía crioelectrónica ha establecido que E3BP se une a cada una de las caras icosaédricas de la levadura. [11] Sin embargo, se ha sugerido que reemplaza un número equivalente de moléculas E2 en el núcleo de PDC bovino.
Hasta 60 moléculas E1 o E3 pueden asociarse con el núcleo E2 de bacterias Gram positivas; la unión es mutuamente excluyente. En eucariotas, E1 está específicamente unido a E2, mientras que E3 se asocia con E3BP. Se cree que están presentes hasta 30 enzimas E1 y 6 E3, aunque el número exacto de moléculas puede variar in vivo y a menudo refleja los requisitos metabólicos del tejido en cuestión.
La piruvato deshidrogenasa se inhibe cuando se incrementan una o más de las tres proporciones siguientes: ATP / ADP , NADH /NAD + y acetil-CoA / CoA . [ cita necesaria ]
En eucariotas, la PDC está estrechamente regulada por su propia piruvato deshidrogenasa quinasa (PDK) y piruvato deshidrogenasa fosfatasa (PDP), desactivándola y activándola respectivamente. [12]
Los productos de la reacción actúan como inhibidores alostéricos de la PDC porque activan la PDK. Los sustratos, a su vez, inhiben la PDK y reactivan la PDC.
Durante la inanición , la cantidad de PDK aumenta en la mayoría de los tejidos, incluido el músculo esquelético , a través de una mayor transcripción genética . En las mismas condiciones, la cantidad de PDP disminuye. La inhibición resultante de la PDC impide que los músculos y otros tejidos catabolicen la glucosa y los precursores de la gluconeogénesis . El metabolismo se desplaza hacia la utilización de grasas , mientras que se minimiza la descomposición de las proteínas musculares para suministrar precursores de la gluconeogénesis y se ahorra la glucosa disponible para que la utilice el cerebro . [ cita necesaria ]
Los iones de calcio tienen un papel en la regulación de la PDC en el tejido muscular, porque activan la PDP, estimulando la glucólisis en su liberación al citosol, durante la contracción muscular . Algunos productos de estas transcripciones liberan H2 en los músculos. Esto puede hacer que los iones de calcio se descompongan con el tiempo. [ cita necesaria ]
En las células eucariotas la descarboxilación del piruvato se produce en el interior de la matriz mitocondrial, tras el transporte del sustrato, el piruvato, desde el citosol . El transporte de piruvato a las mitocondrias se realiza a través de la proteína de transporte piruvato translocasa. La piruvato translocasa transporta el piruvato de forma simportante con un protón (a través de la membrana mitocondrial interna), lo que puede considerarse una forma de transporte activo secundario, pero es posible que se necesite mayor confirmación/apoyo para el uso del desciptor de "transporte activo secundario". aquí (Nota: el método de transporte de piruvato a través de la translocasa de piruvato parece estar acoplado a un gradiente de protones según S. Papa et al., 1971, aparentemente coincidiendo con el transporte activo secundario en definición). [13]
Fuentes alternativas dicen que "el transporte de piruvato a través de la membrana mitocondrial externa parece lograrse fácilmente a través de grandes canales no selectivos, como los canales aniónicos dependientes de voltaje , que permiten la difusión pasiva" y el transporte a través de la membrana mitocondrial interna está mediado por el transportador de piruvato mitocondrial 1 ( MPC1) y portador de piruvato mitocondrial 2 (MPC2). [14]
Al entrar en la matriz mitocondrial, el piruvato se descarboxila, produciendo acetil-CoA (y dióxido de carbono y NADH). Esta reacción irreversible atrapa el acetil-CoA dentro de las mitocondrias (el acetil-CoA sólo puede transportarse fuera de la matriz mitocondrial en condiciones de alto contenido de oxaloacetato a través de la lanzadera de citrato, un intermedio de TCA que normalmente es escaso). El dióxido de carbono producido por esta reacción no es polar y es pequeño, y puede difundirse fuera de las mitocondrias y fuera de la célula. [ cita necesaria ]
En los procariotas , que no tienen mitocondrias, esta reacción se lleva a cabo en el citosol o no se lleva a cabo en absoluto. [ cita necesaria ]
Se descubrió que la enzima piruvato deshidrogenasa que se encuentra en las mitocondrias de las células eucariotas se parece mucho a una enzima del Geobacillus stearothermophilus , que es una especie de bacteria grampositiva . A pesar de las similitudes del complejo piruvato deshidrogenasa con las bacterias grampositivas, existe poca semejanza con las de las bacterias gramnegativas . Las similitudes de las estructuras cuaternarias entre la piruvato deshidrogenasa y las enzimas en las bacterias grampositivas apuntan a una historia evolutiva compartida que se distingue de la historia evolutiva de las enzimas correspondientes que se encuentran en las bacterias gramnegativas. A través de un evento endosimbiótico, la piruvato deshidrogenasa que se encuentra en las mitocondrias eucariotas apunta a vínculos ancestrales que se remontan a las bacterias grampositivas. [15]
Los complejos de piruvato deshidrogenasa comparten muchas similitudes con la 2-oxoácido deshidrogenasa de cadena ramificada (BCOADH), particularmente en su especificidad de sustrato por los alfa-cetoácidos. Específicamente, BCOADH cataliza la degradación de aminoácidos y estas enzimas habrían prevalecido durante los períodos en la Tierra prehistórica dominados por ambientes ricos en aminoácidos. La subunidad E2 de la piruvato deshidrogenasa evolucionó a partir del gen E2 que se encuentra en BCOADH, mientras que ambas enzimas contienen subunidades E3 idénticas debido a la presencia de un solo gen E3. Dado que las subunidades E1 tienen una especificidad distintiva por sustratos particulares, las subunidades E1 de piruvato deshidrogenasa y BCOADH varían pero comparten similitudes genéticas. Las bacterias grampositivas y cianobacterias que luego darían lugar a las mitocondrias y los cloroplastos que se encuentran en las células eucariotas conservaron las subunidades E1 que están genéticamente relacionadas con las que se encuentran en las enzimas BCOADH. [16] [17]
La deficiencia de piruvato deshidrogenasa (PDCD) puede deberse a mutaciones en cualquiera de las enzimas o cofactores utilizados para formar el complejo. Su principal hallazgo clínico es la acidosis láctica . [18] Estas mutaciones de PDCD, que conducen a deficiencias posteriores en la producción de NAD y FAD, dificultan los procesos de fosforilación oxidativa que son clave en la respiración aeróbica. Por lo tanto, el acetil-CoA se reduce mediante mecanismos anaeróbicos a otras moléculas como el lactato, lo que provoca un exceso de lactato corporal y patologías neurológicas asociadas. [19]
Si bien la deficiencia de piruvato deshidrogenasa es poco común, existe una variedad de genes diferentes que, cuando están mutados o no son funcionales, pueden inducir esta deficiencia. Primero, la subunidad E1 de la piruvato deshidrogenasa contiene cuatro subunidades diferentes: dos subunidades alfa designadas como E1-alfa y dos subunidades beta designadas como E1-beta. El gen PDHA1 que se encuentra en las subunidades E1-alfa, cuando muta, causa el 80% de los casos de deficiencia de piruvato deshidrogenasa porque esta mutación reduce la proteína E1-alfa. La disminución de la E1 alfa funcional impide que la piruvato deshidrogenasa se una suficientemente al piruvato, reduciendo así la actividad del complejo general. [20] Cuando el gen PDHB que se encuentra en la subunidad beta E1 del complejo sufre una mutación, esto también conduce a una deficiencia de piruvato deshidrogenasa. [21] Asimismo, mutaciones encontradas en otras subunidades del complejo, como el gen DLAT que se encuentra en la subunidad E2, el gen PDHX que se encuentra en la subunidad E3, así como una mutación en un gen de la piruvato deshidrogenasa fosfatasa, conocido como PDP1, han todos se remontan a la deficiencia de piruvato deshidrogenasa, mientras que se desconoce su contribución específica al estado de la enfermedad. [22] [23] [24]