

Un cladograma (del griego clados "rama" y gramma "carácter") es un diagrama utilizado en cladística para mostrar las relaciones entre organismos. Sin embargo, un cladograma no es un árbol evolutivo porque no muestra cómo se relacionan los ancestros con los descendientes, ni muestra cuánto han cambiado, por lo que muchos árboles evolutivos diferentes pueden ser consistentes con el mismo cladograma. [1] [2] [3] [4] [5] Un cladograma utiliza líneas que se ramifican en diferentes direcciones y terminan en un clado , un grupo de organismos con un último ancestro común . Hay muchas formas de cladogramas, pero todas tienen líneas que se ramifican de otras líneas. Las líneas se remontan hasta donde se bifurcan. Estos puntos de ramificación representan un ancestro hipotético (no una entidad real) del que se puede inferir que exhibe los rasgos compartidos entre los taxones terminales por encima de él. [4] [6] Este ancestro hipotético podría entonces proporcionar pistas sobre el orden de evolución de diversas características, adaptación y otras narrativas evolutivas sobre los ancestros. Aunque tradicionalmente estos cladogramas se generaban en gran medida sobre la base de caracteres morfológicos, los datos de secuenciación de ADN y ARN y la filogenética computacional se utilizan ahora con mucha frecuencia en la generación de cladogramas, ya sea solos o en combinación con la morfología.
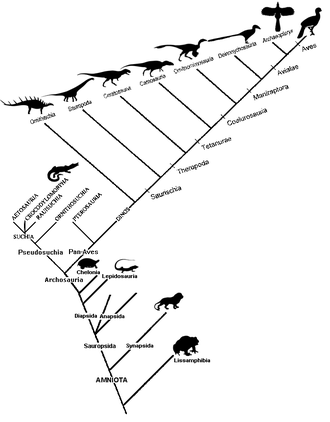
Las características utilizadas para crear un cladograma se pueden clasificar a grandes rasgos como morfológicas (cráneo sinápsido, de sangre caliente, notocorda , unicelular, etc.) o moleculares (ADN, ARN u otra información genética). [7] Antes de la llegada de la secuenciación de ADN, el análisis cladístico utilizaba principalmente datos morfológicos. También se pueden utilizar datos de comportamiento (para animales). [8]
A medida que la secuenciación del ADN se ha vuelto más barata y sencilla, la sistemática molecular se ha convertido en una forma cada vez más popular de inferir hipótesis filogenéticas. [9] Usar un criterio de parsimonia es sólo uno de varios métodos para inferir una filogenia a partir de datos moleculares. Enfoques como el de máxima verosimilitud , que incorporan modelos explícitos de evolución de secuencias, son formas no hennigianas de evaluar datos de secuencias. Otro método poderoso para reconstruir filogenias es el uso de marcadores de retrotransposones genómicos , que se cree que son menos propensos al problema de reversión que afecta a los datos de secuencia. En general, también se supone que tienen una baja incidencia de homoplasias porque alguna vez se pensó que su integración en el genoma era completamente aleatoria; Sin embargo, al menos a veces parece que este no es el caso.

Los investigadores deben decidir qué estados de carácter son "ancestrales" ( plesiomorfias ) y cuáles son derivados ( sinapomorfias ), porque sólo los estados de carácter sinapomórficos proporcionan evidencia de agrupación. [10] Esta determinación generalmente se realiza mediante comparación con los estados de carácter de uno o más grupos externos . Los estados compartidos entre el exogrupo y algunos miembros del endogrupo son simplesiomorfias; Los estados que están presentes sólo en un subconjunto del grupo interno son sinapomorfias. Tenga en cuenta que los estados de caracteres exclusivos de un único terminal (autapomorfias) no proporcionan evidencia de agrupación. La elección de un exogrupo es un paso crucial en el análisis cladístico porque diferentes exogrupos pueden producir árboles con topologías profundamente diferentes.
Una homoplasia es un estado de carácter compartido por dos o más taxones debido a alguna causa distinta a la ascendencia común. [11] Los dos tipos principales de homoplasia son la convergencia (evolución del "mismo" carácter en al menos dos linajes distintos) y la reversión (el regreso a un estado de carácter ancestral). Los caracteres que son obviamente homoplásticos, como el pelaje blanco en diferentes linajes de mamíferos árticos, no deberían incluirse como caracteres en un análisis filogenético, ya que no aportan nada a nuestra comprensión de las relaciones. Sin embargo, la homoplasia a menudo no es evidente a partir de la inspección del carácter en sí (como en la secuencia de ADN, por ejemplo), y luego se detecta por su incongruencia (distribución poco parsimoniosa) en un cladograma de lo más parsimonioso. Tenga en cuenta que los caracteres que son homoplásticos aún pueden contener señales filogenéticas . [12]
Un ejemplo bien conocido de homoplasia debido a la evolución convergente sería el personaje "presencia de alas". Aunque las alas de las aves, los murciélagos y los insectos cumplen la misma función, cada uno evolucionó de forma independiente, como se puede comprobar por su anatomía . Si se calificara a un pájaro, un murciélago y un insecto alado para el carácter "presencia de alas", se introduciría una homoplasia en el conjunto de datos, y esto podría confundir el análisis, lo que posiblemente daría como resultado una hipótesis falsa de relaciones. Por supuesto, la única razón por la que una homoplasia es reconocible es porque hay otros caracteres que implican un patrón de relaciones que revelan su distribución homoplástica.
Un cladograma es el resultado esquemático de un análisis que agrupa taxones basándose únicamente en sinapomorfias. Hay muchos otros algoritmos filogenéticos que tratan los datos de manera algo diferente y dan como resultado árboles filogenéticos que parecen cladogramas pero no son cladogramas. Por ejemplo, los algoritmos fenéticos, como UPGMA y Neighbor-Joining, agrupan por similitud general y tratan tanto las sinapomorfias como las simplesiomorfias como evidencia de agrupación. Los diagramas resultantes son fenogramas, no cladogramas. De manera similar, los resultados de los métodos basados en modelos (Máximo Los enfoques de probabilidad o bayesianos) que tienen en cuenta tanto el orden de ramificación como la "longitud de la rama", cuentan tanto las sinapomorfias como las autapomorfias como evidencia a favor o en contra de la agrupación. Los diagramas resultantes de ese tipo de análisis tampoco son cladogramas. [13]
Hay varios algoritmos disponibles para identificar el "mejor" cladograma. [14] La mayoría de los algoritmos utilizan una métrica para medir qué tan consistente es un cladograma candidato con los datos. La mayoría de los algoritmos de cladogramas utilizan técnicas matemáticas de optimización y minimización.
En general, los algoritmos de generación de cladogramas deben implementarse como programas de computadora, aunque algunos algoritmos pueden realizarse manualmente cuando los conjuntos de datos son modestos (por ejemplo, sólo unas pocas especies y un par de características).
Algunos algoritmos son útiles sólo cuando los datos característicos son moleculares (ADN, ARN); otros algoritmos son útiles sólo cuando los datos característicos son morfológicos. Se pueden utilizar otros algoritmos cuando los datos característicos incluyen datos tanto moleculares como morfológicos.
Los algoritmos para cladogramas u otros tipos de árboles filogenéticos incluyen mínimos cuadrados , unión de vecinos , parsimonia , máxima verosimilitud e inferencia bayesiana .
Los biólogos a veces utilizan el término parsimonia para un tipo específico de algoritmo de generación de cladogramas y, a veces, como término general para todos los algoritmos filogenéticos. [15]
Los algoritmos que realizan tareas de optimización (como la construcción de cladogramas) pueden ser sensibles al orden en que se presentan los datos de entrada (la lista de especies y sus características). Ingresar los datos en varios órdenes puede hacer que el mismo algoritmo produzca diferentes "mejores" cladogramas. En estas situaciones, el usuario debe ingresar los datos en varios órdenes y comparar los resultados.
El uso de diferentes algoritmos en un único conjunto de datos a veces puede producir diferentes "mejores" cladogramas, porque cada algoritmo puede tener una definición única de lo que es "mejor".
Debido a la cantidad astronómica de cladogramas posibles, los algoritmos no pueden garantizar que la solución sea la mejor en general. Se seleccionará un cladograma no óptimo si el programa se fija en un mínimo local en lugar del mínimo global deseado. [16] Para ayudar a resolver este problema, muchos algoritmos de cladograma utilizan un enfoque de recocido simulado para aumentar la probabilidad de que el cladograma seleccionado sea el óptimo. [17]
La posición basal es la dirección de la base (o raíz) de un cladograma o árbol filogenético enraizado. Un clado basal es el clado más antiguo (de un rango taxonómico determinado [a]) que se ramifica dentro de un clado más grande.
La prueba de diferencia de longitud de incongruencia (ILD) es una medida de cómo la combinación de diferentes conjuntos de datos (por ejemplo, genes morfológicos y moleculares, de plástidos y nucleares) contribuye a un árbol más largo. Se mide calculando primero la longitud total del árbol de cada partición y sumándolas. Luego se hacen réplicas haciendo particiones ensambladas al azar que constan de las particiones originales. Las longitudes se suman. Se obtiene un valor de p de 0,01 para 100 réplicas si 99 réplicas tienen longitudes de árbol combinadas más largas.
Algunas medidas intentan medir la cantidad de homoplasia en un conjunto de datos con referencia a un árbol, [18] aunque no está necesariamente claro exactamente qué propiedad pretenden cuantificar estas medidas [19]
El índice de consistencia (CI) mide la consistencia de un árbol con un conjunto de datos, una medida de la cantidad mínima de homoplasia que implica el árbol. [20] Se calcula contando el número mínimo de cambios en un conjunto de datos y dividiéndolo por el número real de cambios necesarios para el cladograma. [20] También se puede calcular un índice de coherencia para un carácter individual i , denominado c i .
Además de reflejar la cantidad de homoplasia, la métrica también refleja la cantidad de taxones en el conjunto de datos, [21] (en menor medida) la cantidad de caracteres en un conjunto de datos, [22] el grado en que cada carácter lleva información filogenética, [ 23] y la forma en que se codifican los caracteres aditivos, lo que los hace inadecuados para su propósito. [24]
c i ocupa un rango de 1 a 1/[ n.taxa /2] en caracteres binarios con una distribución de estado uniforme; su valor mínimo es mayor cuando los estados no están distribuidos uniformemente. [23] [18] En general, para un carácter binario o no binario con , ci ocupa un rango de 1 a . [23]
El índice de retención (RI) se propuso como una mejora del CI "para ciertas aplicaciones" [25]. Esta métrica también pretende medir la cantidad de homoplasia, pero también mide qué tan bien las sinapomorfias explican el árbol. Se calcula tomando (el número máximo de cambios en un árbol menos el número de cambios en el árbol) y dividiendo por (el número máximo de cambios en el árbol menos el número mínimo de cambios en el conjunto de datos).
El índice de consistencia reescalado (RC) se obtiene multiplicando el IC por el RI; de hecho, esto amplía el rango del IC de modo que su valor mínimo teóricamente alcanzable se reescala a 0, y su máximo permanece en 1. [18] [25] El índice de homoplasia (HI) es simplemente 1 − IC.
Esto mide la cantidad de homoplasia observada en un árbol en relación con la cantidad máxima de homoplasia que teóricamente podría estar presente - 1 - (exceso de homoplasia observado) / (exceso máximo de homoplasia). [22] Un valor de 1 indica que no hay homoplasia; 0 representa tanta homoplasia como la que habría en un conjunto de datos completamente aleatorio, y los valores negativos indican aún más homoplasia (y tienden a ocurrir solo en ejemplos artificiales). [22] El HER se presenta como la mejor medida de homoplasia disponible actualmente. [18] [26]