La secuenciación de ADN mediante microscopía electrónica de transmisión es una tecnología de secuenciación de moléculas individuales que utiliza técnicas de microscopía electrónica de transmisión . El método fue concebido y desarrollado en los años 60 y 70, [1] pero perdió popularidad cuando se reconoció la magnitud del daño a la muestra. [2]
Para que el ADN se pueda visualizar claramente con un microscopio electrónico , debe estar marcado con átomos pesados. Además, las técnicas de obtención de imágenes especializadas y la óptica con corrección de aberraciones son beneficiosas para obtener la resolución necesaria para obtener imágenes de la molécula de ADN marcada. En teoría, la secuenciación del ADN mediante microscopía electrónica de transmisión podría proporcionar longitudes de lectura extremadamente largas, pero el problema del daño del haz de electrones aún puede persistir y la tecnología aún no se ha desarrollado comercialmente.
Sólo unos pocos años después de que James Watson y Francis Crick dedujeran la estructura del ADN , y casi dos décadas antes de que Frederick Sanger publicara el primer método para la secuenciación rápida del ADN , Richard Feynman , un físico estadounidense, imaginó el microscopio electrónico como la herramienta que un día permitiría a los biólogos "ver el orden de las bases en la cadena de ADN ". [3] Feynman creía que si el microscopio electrónico pudiera hacerse lo suficientemente potente, entonces sería posible visualizar la estructura atómica de todos y cada uno de los compuestos químicos, incluido el ADN.
En 1970, Albert Crewe desarrolló la técnica de obtención de imágenes de campo oscuro anular de ángulo alto (HAADF) en un microscopio electrónico de transmisión de barrido . Utilizando esta técnica, visualizó átomos pesados individuales en películas delgadas de carbono amorfo. [4] En 2010, Krivanek y sus colegas informaron sobre varias mejoras técnicas del método HAADF, incluida una combinación de óptica electrónica con corrección de aberración y bajo voltaje de aceleración. Este último es crucial para obtener imágenes de objetos biológicos, ya que permite reducir el daño causado por el haz y aumentar el contraste de la imagen para los átomos ligeros. Como resultado, se pudieron obtener imágenes de sustituciones de un solo átomo en una monocapa de nitruro de boro. [5]
A pesar de la invención de una multitud de tecnologías de secuenciación química y fluorescente, la microscopía electrónica todavía se está explorando como un medio para realizar la secuenciación de ADN de una sola molécula. Por ejemplo, en 2012 una colaboración entre científicos de la Universidad de Harvard , la Universidad de New Hampshire y ZS Genetics demostró la capacidad de leer secuencias largas de ADN utilizando la técnica, [6] sin embargo, la tecnología de secuenciación de ADN mediante microscopía electrónica de transmisión aún está lejos de estar disponible comercialmente. [7]
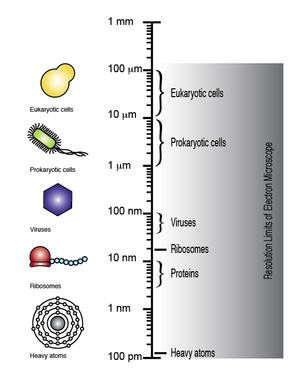
El microscopio electrónico tiene la capacidad de obtener una resolución de hasta 100 pm, con lo que se pueden observar biomoléculas y estructuras microscópicas como virus, ribosomas, proteínas, lípidos, pequeñas moléculas e incluso átomos individuales. [8]
Aunque el ADN es visible cuando se observa con el microscopio electrónico, la resolución de la imagen obtenida no es lo suficientemente alta como para permitir descifrar la secuencia de las bases individuales , es decir , la secuenciación del ADN . Sin embargo, al etiquetar diferencialmente las bases del ADN con átomos pesados o metales, es posible tanto visualizar como distinguir entre las bases individuales. Por lo tanto, la microscopía electrónica junto con el etiquetado diferencial de átomos pesados del ADN podría usarse para obtener imágenes directas del ADN con el fin de determinar su secuencia. [7] [9] [10] [11]
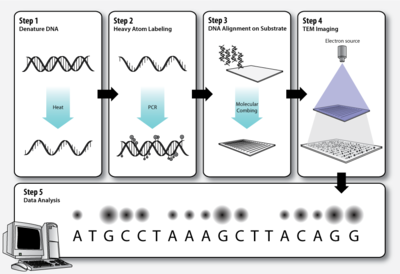
Al igual que en una reacción en cadena de la polimerasa (PCR) estándar , las moléculas de ADN de doble cadena que se van a secuenciar deben desnaturalizarse antes de que se pueda sintetizar la segunda cadena con nucleótidos marcados.
Los elementos que forman las moléculas biológicas ( C , H , N , O , P , S ) son demasiado ligeros ( número atómico bajo, Z ) para poder visualizarse claramente como átomos individuales mediante microscopía electrónica de transmisión . Para evitar este problema, las bases de ADN pueden marcarse con átomos más pesados (Z más alto). Cada nucleótido se marca con una etiqueta pesada característica, de modo que se puedan distinguir en la micrografía electrónica de transmisión.
Las moléculas de ADN deben extenderse sobre un sustrato sólido y delgado para que el orden de las bases marcadas sea claramente visible en la micrografía electrónica. El peinado molecular es una técnica que utiliza la fuerza de una interfaz aire-agua en retroceso para extender las moléculas de ADN, dejándolas unidas irreversiblemente a una capa de silano una vez secas. [13] [14] Este es un medio por el cual se puede lograr la alineación del ADN sobre un sustrato sólido.

La microscopía electrónica de transmisión (MET) produce imágenes de alta resolución y gran aumento al pasar un haz de electrones a través de una muestra muy delgada. Mientras que la resolución atómica se ha demostrado con la MET convencional, una mejora adicional en la resolución espacial requiere corregir las aberraciones esféricas y cromáticas de las lentes del microscopio . Esto solo ha sido posible en la microscopía electrónica de transmisión de barrido , donde la imagen se obtiene escaneando el objeto con un haz de electrones finamente enfocado, de manera similar a un tubo de rayos catódicos . Sin embargo, la mejora lograda en la resolución viene acompañada de la irradiación del objeto estudiado con intensidades de haz mucho más altas, el daño concomitante de la muestra y los artefactos de imagen asociados. [15] Se aplican diferentes técnicas de imagen dependiendo de si la muestra contiene átomos pesados o ligeros:
Los puntos oscuros y brillantes en la micrografía electrónica, correspondientes a las bases de ADN marcadas diferencialmente, se analizan mediante un software informático.
La secuenciación de ADN mediante microscopía electrónica de transmisión aún no está disponible comercialmente, pero las largas longitudes de lectura que esta tecnología podría proporcionar algún día la harán útil en una variedad de contextos.
Al secuenciar un genoma, se debe dividir en partes lo suficientemente cortas como para secuenciarlas en una sola lectura. Luego, estas lecturas se deben volver a unir como un rompecabezas alineando las regiones que se superponen entre las lecturas; este proceso se denomina ensamblaje de genoma de novo . Cuanto mayor sea la longitud de lectura que proporciona una plataforma de secuenciación, más largas serán las regiones superpuestas y más fácil será ensamblar el genoma. Desde una perspectiva computacional, la secuenciación de Sanger microfluídica sigue siendo la forma más eficaz de secuenciar y ensamblar genomas para los que no existe una secuencia genómica de referencia . Las longitudes de lectura relativamente largas proporcionan una superposición sustancial entre las lecturas de secuenciación individuales, lo que permite una mayor confianza estadística en el ensamblaje. Además, las lecturas de Sanger largas pueden abarcar la mayoría de las regiones de secuencia de ADN repetitiva que, de lo contrario, confunden el ensamblaje de secuencias al causar alineaciones falsas. Sin embargo, el ensamblaje de genoma de novo mediante secuenciación de Sanger es extremadamente costoso y requiere mucho tiempo. Las tecnologías de secuenciación de segunda generación , [19] si bien son menos costosas, generalmente no son aptas para el ensamblaje de genomas de novo debido a las longitudes de lectura cortas. En general, las tecnologías de secuenciación de tercera generación, [11] incluida la secuenciación de ADN por microscopía electrónica de transmisión, apuntan a mejorar la longitud de lectura manteniendo al mismo tiempo un bajo costo de secuenciación. Por lo tanto, a medida que las tecnologías de secuenciación de tercera generación mejoren, el ensamblaje de genomas de novo rápido y económico se convertirá en una realidad.
Un haplotipo es una serie de alelos ligados que se heredan juntos en un solo cromosoma. La secuenciación de ADN se puede utilizar para genotipar todos los polimorfismos de un solo nucleótido (SNP) que constituyen un haplotipo. Sin embargo, las lecturas de secuenciación de ADN cortas a menudo no se pueden escalonar; es decir, las variantes heterocigóticas no se pueden asignar con confianza al haplotipo correcto. De hecho, la haplotipificación con datos de secuenciación de ADN de lectura corta requiere una cobertura muy alta (cobertura promedio >50x de cada base de ADN) para identificar con precisión los SNP, así como datos de secuenciación adicionales de los padres para que se pueda utilizar la transmisión mendeliana para estimar los haplotipos. [20] Las tecnologías de secuenciación que generan lecturas largas, incluida la secuenciación de ADN por microscopía electrónica de transmisión, pueden capturar haplobloques completos en una sola lectura. Es decir, los haplotipos no se dividen entre múltiples lecturas y los alelos genéticamente ligados permanecen juntos en los datos de secuenciación. Por lo tanto, las lecturas largas hacen que la haplotipificación sea más fácil y precisa, lo que es beneficioso para el campo de la genética de poblaciones .
Los genes normalmente están presentes en dos copias en el genoma humano diploide ; los genes que se desvían de este número de copias estándar se denominan variantes del número de copias (CNV) . La variación del número de copias puede ser benigna (normalmente son variantes comunes, llamadas polimorfismos del número de copias) o patógena. [21] Las CNV se detectan mediante hibridación in situ fluorescente (FISH) o hibridación genómica comparativa (CGH) . Para detectar los puntos de ruptura específicos en los que se produce una deleción, o para detectar lesiones genómicas introducidas por un evento de duplicación o amplificación, la CGH se puede realizar utilizando una matriz de mosaico ( matriz CGH ), o se puede secuenciar la región variante. Las lecturas de secuenciación largas son especialmente útiles para analizar duplicaciones o amplificaciones, ya que es posible analizar la orientación de los segmentos amplificados si se capturan en una única lectura de secuenciación.
La genómica del cáncer, u oncogenómica , es un campo emergente en el que se está aplicando tecnología de secuenciación de ADN de segunda generación de alto rendimiento para secuenciar genomas completos del cáncer. El análisis de estos datos de secuenciación de lectura corta abarca todos los problemas asociados con el ensamblaje de genomas de novo utilizando datos de lectura corta. [22] Además, los genomas del cáncer a menudo son aneuploides . [23] Estas aberraciones, que son esencialmente variantes del número de copias a gran escala, se pueden analizar mediante tecnologías de secuenciación de segunda generación utilizando la frecuencia de lectura para estimar el número de copias. [22] Sin embargo, lecturas más largas proporcionarían una imagen más precisa del número de copias, la orientación de las regiones amplificadas y los SNP presentes en los genomas del cáncer.
El microbioma se refiere al conjunto total de microbios presentes en un microambiente y sus respectivos genomas. Por ejemplo, se estima que 100 billones de células microbianas colonizan el cuerpo humano en un momento dado. [24] El microbioma humano es de particular interés, ya que estas bacterias comensales son importantes para la salud y la inmunidad humanas. La mayoría de los genomas bacterianos de la Tierra aún no han sido secuenciados; emprender un proyecto de secuenciación del microbioma requeriría un extenso ensamblaje de genomas de novo , una perspectiva que es desalentadora con tecnologías de secuenciación de ADN de lectura corta. [25] Lecturas más largas facilitarían en gran medida el ensamblaje de nuevos genomas microbianos.
En comparación con otras tecnologías de secuenciación de ADN de segunda y tercera generación, la secuenciación de ADN mediante microscopía electrónica de transmisión tiene una serie de posibles fortalezas y debilidades clave, que en última instancia determinarán su utilidad y prominencia como futura tecnología de secuenciación de ADN.
Se han desarrollado o se están desarrollando actualmente muchas tecnologías de secuenciación de ADN de segunda y tercera generación distintas de Sanger con el objetivo común de aumentar el rendimiento y disminuir los costos para que la medicina genética personalizada pueda realizarse plenamente.
Tanto el Premio Archon X de Genómica de 10 millones de dólares estadounidenses apoyado por la Fundación X Prize (Santa Mónica, California, EE. UU.) como los premios en subvenciones de 70 millones de dólares estadounidenses apoyados por el Instituto Nacional de Investigación del Genoma Humano de los Institutos Nacionales de Salud (NIH-NHGRI) están impulsando el rápido aumento de la actividad de investigación en el desarrollo de nuevas tecnologías de secuenciación de ADN. [7]
Dado que cada tecnología de secuenciación de ADN se define por diferentes enfoques, técnicas y estrategias, cada una de ellas tiene sus propias fortalezas y debilidades. En la Tabla 1 se presenta una comparación de parámetros importantes entre las distintas tecnologías de secuenciación de ADN de segunda y tercera generación.