El término S/MAR ( scaffold/matrix attached region ), también llamado SAR ( scaffold-attachment region ) o MAR ( matrix-associated region ), son secuencias en el ADN de los cromosomas eucariotas donde se une la matriz nuclear . Como componentes arquitectónicos del ADN que organizan el genoma de los eucariotas en unidades funcionales dentro del núcleo celular , los S/MAR median la organización estructural de la cromatina dentro del núcleo. Estos elementos constituyen puntos de anclaje del ADN para el andamiaje de la cromatina y sirven para organizar la cromatina en dominios estructurales . Los estudios sobre genes individuales llevaron a la conclusión de que la organización dinámica y compleja de la cromatina mediada por elementos S/MAR juega un papel importante en la regulación de la expresión génica .
Se sabe desde hace muchos años que un entramado de polímeros, denominado " matriz nuclear " o "andamio nuclear", es un componente esencial de los núcleos eucariotas. Este esqueleto nuclear actúa como soporte dinámico para muchos eventos especializados relacionados con la lectura de una amplia gama de información genética (véase más abajo).
Los S/MAR se asignan a ubicaciones no aleatorias en el genoma. Se encuentran en los flancos de las regiones transcritas, en los intrones 5' y también en las regiones de grupos de puntos de ruptura de genes (BCR). Al ser puntos de asociación para proteínas estructurales nucleares comunes, los S/MAR son necesarios para la replicación y transcripción cromosómica auténtica y eficiente , para la recombinación y la condensación cromosómica. Los S/MAR no tienen una secuencia de consenso obvia. Aunque los elementos prototipo consisten en regiones ricas en AT de varios cientos de pares de bases de longitud, la composición general de bases definitivamente no es el determinante principal de su actividad. En cambio, su función requiere un patrón de "parches AT" que confieren la propensión al desapareamiento de la cadena local bajo tensión torsional .
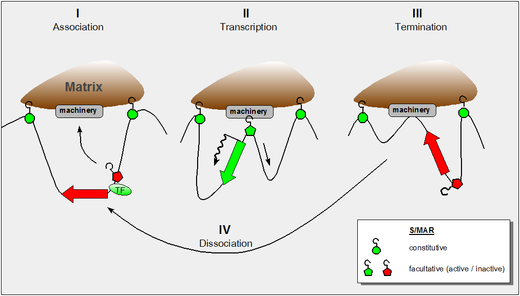
Los enfoques bioinformáticos respaldan la idea de que, por estas propiedades, las S/MAR no solo separan una unidad transcripcional dada (dominio de cromatina) de sus vecinos, sino que también proporcionan plataformas para el ensamblaje de factores que permiten eventos transcripcionales dentro de un dominio dado. Una mayor propensión a separar las cadenas de ADN (el llamado potencial de 'desestabilización dúplex inducida por estrés', SIDD ) puede servir para la formación de estructuras secundarias como estructuras cruciformes o de deslizamiento , que son características reconocibles para una serie de enzimas ( ADNasas , topoisomerasas , poli(ADP-ribosil) polimerasas y enzimas del aparato de acetilación de histonas y metilación de ADN). Las S/MAR se han clasificado como constitutivas (actúan como límites de dominio permanentes en todos los tipos de células) o facultativas (relacionadas con el tipo de célula y la actividad) dependiendo de sus propiedades dinámicas.
Aunque se ha estimado que el número de S/MAR en el genoma humano se acerca a 64.000 (dominios de cromatina) más 10.000 adicionales (focos de replicación), en 2007 todavía sólo una fracción menor (559 para todos los eucariotas) había cumplido los criterios estándar para una anotación en la base de datos S/MARt. [1]
Las opiniones actuales sobre la matriz nuclear la consideran una entidad dinámica, que cambia sus propiedades en función de los requerimientos del núcleo celular, de manera muy similar a cómo el citoesqueleto adapta su estructura y función a las señales externas. En retrospectiva, cabe señalar que el descubrimiento de las S/MAR tuvo dos vías principales:
Trabajos posteriores demostraron tanto la función constitutiva (similar a SAR) como la facultativa (similar a MAR) de los elementos dependiendo del contexto. Mientras que se encontró que los S/MAR constitutivos estaban asociados con un sitio hipersensible a la ADNasa I en "todos" los tipos de células (ya sea que se haya transcrito o no el dominio incluido), la hipersensibilidad a la ADNasa I del tipo facultativo dependía del estado transcripcional. [4] La principal diferencia entre estos dos tipos funcionales de S/MAR es su tamaño: los elementos constitutivos pueden extenderse sobre varios kilopares de bases mientras que los facultativos están en el límite de tamaño inferior alrededor de 300 pares de bases.
La figura muestra nuestra comprensión actual de estas propiedades e incorpora los siguientes hallazgos:
Como alternativa a los vectores virales, que pueden tener efectos no deseados en el organismo de los pacientes, se están estudiando métodos no virales de terapia génica. Uno de estos métodos utiliza plásmidos con propiedades especiales, los llamados episomas . Los episomas tienen la capacidad de dividirse junto con el resto del genoma eucariota durante la mitosis. En comparación con los plásmidos estándar, no se silencian epigenéticamente dentro del núcleo y no se destruyen enzimáticamente. Los episomas adquieren esta capacidad mediante la presencia de la secuencia S/MAR dentro de su construcción. [7]
En 2006, Tetko encontró una fuerte correlación de los S/MAR intragénicos con la expresión espaciotemporal de genes en Arabidopsis thaliana . [8] A escala del genoma, se han detectado patrones pronunciados de expresión de genes que contienen S/MAR específicos de tejidos y órganos y del desarrollo. En particular, los genes de factores de transcripción contienen una porción significativamente mayor de S/MAR. La marcada diferencia en las características de expresión de los genes que contienen S/MAR enfatiza su importancia funcional y la importancia de las características cromosómicas estructurales para la regulación genética en plantas, así como en otros eucariotas.