La reacción en cadena de la polimerasa por superposición y extensión (o OE-PCR ) es una variante de la PCR . También se la conoce como PCR por superposición y extensión (SOE) . Se utiliza para ensamblar múltiples fragmentos de ADN bicatenario más pequeños en una secuencia de ADN más grande. La OE-PCR se usa ampliamente para insertar mutaciones en puntos específicos de una secuencia o para ensamblar una secuencia de ADN personalizada a partir de fragmentos de ADN más pequeños en un polinucleótido más grande . [1]
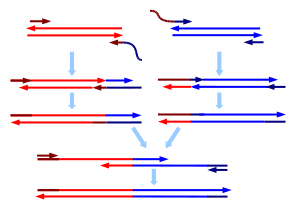
Como en la mayoría de las reacciones de PCR, se utilizan dos cebadores (uno para cada extremo) por secuencia. Para unir dos moléculas de ADN, se utilizan cebadores especiales en los extremos que se van a unir. Para cada molécula, el cebador del extremo que se va a unir se construye de forma que tenga un saliente 5' complementario al extremo de la otra molécula. Después de la hibridación, cuando se produce la replicación, el ADN se extiende con una nueva secuencia que es complementaria a la molécula a la que se va a unir. Una vez que ambas moléculas de ADN se extienden de esa manera, se mezclan y se lleva a cabo una PCR con solo los cebadores de los extremos más alejados. Las secuencias complementarias superpuestas introducidas servirán como cebadores y las dos secuencias se fusionarán. Este método tiene una ventaja sobre otras técnicas de empalme de genes en que no requiere sitios de restricción.
Para obtener mayores rendimientos, algunos cebadores se utilizan en exceso, como en la PCR asimétrica .

Para insertar una mutación en una secuencia de ADN, se diseña un cebador específico . El cebador puede contener una única sustitución o una nueva secuencia en su extremo 5'. Si se requiere una deleción , se añade una secuencia que se encuentre en el extremo 5' de la deleción, ya que el extremo 3' del cebador debe tener complementariedad con la cadena molde para que el cebador pueda anexarse lo suficiente al ADN molde.
Después de la hibridación del cebador con la plantilla, la replicación del ADN continúa hasta el final de la plantilla. El dúplex se desnaturaliza y el segundo cebador se hibrida con la cadena de ADN recién formada, que contiene la secuencia del primer cebador. La replicación continúa para producir una cadena de la secuencia requerida, que contiene la mutación.
El dúplex se desnaturaliza nuevamente y el primer cebador ahora puede unirse a la última cadena de ADN. La reacción de replicación continúa para producir un fragmento de ADN completamente dimerizado . Después de más ciclos de PCR, para amplificar el ADN, la muestra se puede separar mediante electroforesis en gel de agarosa , seguida de electroelución para su recolección.
Generar oligonucleótidos de más de ~110 nucleótidos de longitud de manera eficiente es muy difícil, por lo que para insertar una mutación más allá de lo que permite un cebador de 110 nt en una secuencia, es necesario emplear PCR de extensión por superposición. En la PCR-OE, la secuencia que se está modificando se utiliza para crear dos cadenas modificadas con la mutación en extremos opuestos, utilizando la técnica descrita anteriormente. Después de mezclar y desnaturalizar, se permite que las cadenas se asocien para producir tres combinaciones diferentes, como se detalla en el diagrama. Solo el dúplex sin superposición en el extremo 5' permitirá la extensión por la ADN polimerasa en la dirección 3' a 5'.
Después de la extensión de la reacción OE-PCR, la mezcla de PCR o los fragmentos eluidos de tamaño apropiado se someten a PCR normal, utilizando los cebadores más externos utilizados en las reacciones de PCR mutagénicas iniciales.
Además, la combinación de OE-PCR y PCR asimétrica podría utilizarse para mejorar la eficiencia de la mutagénesis dirigida al sitio . [2]

Además de la introducción de mutaciones, la PCR de extensión superpuesta se utiliza ampliamente para ensamblar secuencias de ADN complejas sin la introducción de nucleótidos no deseados en ninguna posición. Esto es posible porque la PCR de extensión superpuesta se basa en la utilización de salientes complementarios para guiar el empalme sin cicatrices de fragmentos de ADN personalizados en un orden deseado. Esta es la principal ventaja de la PCR de extensión superpuesta y otros métodos de clonación basados en homología larga como el ensamblaje de Gibson , que superan las limitaciones de los métodos tradicionales de clonación por ligadura y digestión con enzimas de restricción . [3]
El ensamblaje de secuencias de ADN personalizadas con OE-PCR consta de tres pasos principales. En primer lugar, se amplifican secuencias de ADN individuales mediante PCR a partir de diferentes plantillas y se flanquean con los salientes complementarios necesarios. En segundo lugar, los productos de PCR obtenidos anteriormente se combinan en la reacción de PCR de extensión por superposición, donde los salientes complementarios se unen por pares, lo que permite que la polimerasa extienda la cadena de ADN. Finalmente, se utilizan cebadores externos que apuntan a los salientes externos y el producto de ADN deseado se amplifica en la reacción de PCR final.
El éxito general de los ensamblajes de ADN basados en OE-PCR depende de varios factores, siendo los más relevantes las características intrínsecas de la secuencia de ADN a ensamblar, la secuencia y longitud de los salientes superpuestos, el diseño de cebadores externos para la amplificación final y las condiciones de la reacción de PCR. Normalmente, se pueden empalmar de 2 a 6 fragmentos simultáneamente en una sola reacción de OE-PCR. [4] Los salientes deben tener al menos 40 nucleótidos de longitud para asegurar una interacción adecuada entre los fragmentos. Los cebadores de amplificación final se diseñan comúnmente siguiendo pautas generales para PCR, sin embargo, se utilizan en una concentración de 2 a 5 veces menor que en las reacciones de PCR estándar, ya que se ha demostrado que esto reduce las amplificaciones no deseadas. [5] Además, se recomienda encarecidamente la utilización de ADN polimerasas correctoras .