La ADN adenina metilasa , (Dam) [1] (también ADN-metiltransferasa de sitio específico (adenina-específica) , EC 2.1.1.72, metilasa de modificación , sistema de restricción-modificación ) es una enzima que agrega un grupo metilo a la adenina de la secuencia 5'-GATC-3' en el ADN recién sintetizado . [2] [3] Inmediatamente después de la síntesis de ADN , la cadena hija permanece sin metilar durante un corto tiempo. [4] Es una metiltransferasa huérfana que no forma parte de un sistema de restricción-modificación y regula la expresión génica. [5] [6] [7] [8] Esta enzima cataliza la siguiente reacción química
Se trata de un gran grupo de enzimas exclusivas de los procariotas y los bacteriófagos. [9]
La enzima adenina metiltransferasa del ADN de E. coli (Dam) se utiliza ampliamente para la técnica de perfil de cromatina DamID , en la que la Dam se fusiona a una proteína de unión al ADN de interés y se expresa como un transgén en un organismo modelo genéticamente manejable para identificar sitios de unión de proteínas. [10]
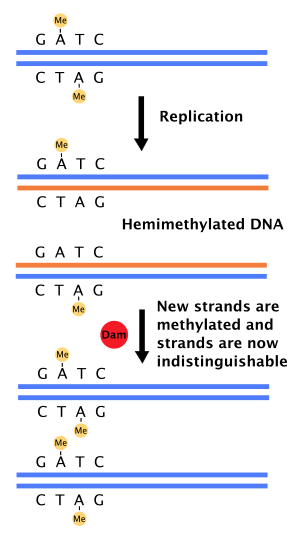
Cuando la ADN polimerasa comete un error que da como resultado un par de bases desapareado o una pequeña inserción o eliminación durante la síntesis de ADN , la célula reparará el ADN mediante una vía llamada reparación de desajustes . Sin embargo, la célula debe ser capaz de diferenciar entre la cadena molde y la cadena recién sintetizada. En algunas bacterias , las cadenas de ADN son metiladas por la metilasa Dam y, por lo tanto, inmediatamente después de la replicación , el ADN será hemimetilado. [4] Una enzima reparadora, MutS , se une a los desajustes en el ADN y recluta a MutL, que posteriormente activa la endonucleasa MutH. MutH se une a los sitios GATC hemimetilados y, cuando se activa, escindirá selectivamente la cadena hija no metilada, lo que permite que la helicasa y las exonucleasas escindan la cadena naciente en la región que rodea el desajuste. [4] [11] Luego, la cadena es resintetizada por la ADN polimerasa III .
La activación del origen de replicación (oriC) en las células bacterianas está muy controlada para garantizar que la replicación del ADN se produzca solo una vez durante cada división celular. Parte de esto se puede explicar por la hidrólisis lenta del ATP por parte de DnaA, una proteína que se une a las repeticiones en el oriC para iniciar la replicación. La metilasa Dam también desempeña un papel porque el oriC tiene 11 secuencias 5'-GATC-3' (en E. coli ). Inmediatamente después de la replicación del ADN, el oriC se hemimetila y queda secuestrado durante un período de tiempo. Solo después de esto, el oriC se libera y debe ser metilado por completo por la metilasa Dam antes de que se produzca la unión a DnaA.
Dam también desempeña un papel en la promoción y represión de la transcripción de ARN . En E. coli, las secuencias GATC descendentes están metiladas , lo que promueve la transcripción. Por ejemplo, la variación de fase de pili asociada a pielonefritis (PAP) en E. coli uropatógena está controlada por Dam a través de la metilación de los dos sitios GATC proximales y distales al promotor PAP . [12] Dado su papel de regulación de proteínas en E. coli , el gen de metilasa Dam no es esencial ya que una eliminación del gen aún deja a las bacterias viables. [13] La retención de la viabilidad a pesar de una eliminación del gen dam también se observa en Salmonella y Aggregatibacter actinomycetemcomitans . [14] [15] Sin embargo, en organismos como Vibrio cholerae y Yersinia pseudotuberculosis , el gen dam es esencial para la viabilidad. [16] La eliminación del gen dam en Aggregatibacter actinomycetemcomitans resultó en niveles desregulados de la proteína, leucotoxina, y también redujo la capacidad del microbio para invadir las células epiteliales orales. [15] Además, un estudio sobre Streptococcus mutans deficiente en metilasa Dam , un patógeno dental, reveló la desregulación de 103 genes, algunos de los cuales incluyen potencial cariogénico. [16]
La similitud en los dominios catalíticos de las metiltransferasas de C5-citosina y las metiltransferasas de N6 y N4-adenina generó un gran interés en comprender la base de las similitudes y diferencias funcionales. Las metiltransferasas o metilasas se clasifican en tres grupos (Grupos α, β y γ) según el orden secuencial de ciertos 9 motivos y el Dominio de Reconocimiento de Objetivo (TRD). [17] El Motivo I consiste en un tripéptido Gly-X-Gly y se lo conoce como bucle G y está implicado en la unión del cofactor S-adenosil metionina . [18] El Motivo II está altamente conservado entre las metilasas de N4 y N6-adenina y contiene un aminoácido cargado negativamente seguido de una cadena lateral hidrofóbica en las últimas posiciones de la cadena β2 para unirse a AdoMet . [17] El motivo III también está implicado en la unión de Adomet. El motivo IV es especialmente importante y bien conocido en las caracterizaciones de metilasas. Consiste en un componente diprolílico y está altamente conservado entre las N6-adenina metiltransferasas como el motivo DPPY, sin embargo, este motivo puede variar para las N4-adenina y C5-citosina metiltransferasas. Se ha descubierto que el motivo DPPY es esencial para la unión de AdoMet. [19] Los motivos IV-VIII desempeñan un papel en la actividad catalítica, mientras que los motivos 1-III y X desempeñan un papel en la unión del cofactor. Para las N6-adenina metilasas, el orden secuencial de estos motivos es el siguiente: N-terminal - X - I - II - III - TRD - IV - V - VI - VII - VIII - C-terminal y la metilasa Dam de E . coli sigue esta secuencia estructural. [17] Un experimento de cristalografía de 2015 mostró que E . La metilasa Dam de E. coli fue capaz de unirse a ADN no GATC con la misma secuencia de motivos analizada; los autores postulan que la estructura obtenida podría servir como base para la represión de la transcripción que no se basa en la metilación. [20]
La metiltransferasa Dam es una metiltransferasa huérfana que no forma parte de un sistema de modificación por restricción, sino que opera de forma independiente para regular la expresión génica, la reparación de errores de apareamiento y la replicación bacteriana, entre muchas otras funciones. Este no es el único ejemplo de una metiltransferasa huérfana, ya que existe la metiltransferasa regulada por el ciclo celular (CcrM), que metila el ADN hemimetilado 5'-GANTC-'3 para controlar el ciclo de vida de Caulobacter crescentus y otras especies relacionadas. [21]
A diferencia de sus contrapartes bacterianas, también existen metiltransferasas huérfanas de fagos, y más notablemente en los bacteriófagos T2, T4 y otros bacteriófagos T-even que infectan a E. coli. [5] En un estudio se identificó que a pesar de compartir cualquier homología de secuencia, las secuencias de aminoácidos de la metilasa Dam de E. coli y T4 comparten una identidad de secuencia de hasta el 64% en cuatro regiones de 11 a 33 residuos de longitud, lo que sugiere un origen evolutivo común para los genes de la metilasa bacteriana y del fago. [22] Las metilasas T2 y T4 difieren de la metilasa Dam de E. coli no solo en su capacidad para metilar 5-hidroximetilcitosina sino también para metilar sitios de ADN no canónicos. A pesar de la extensa caracterización in vitro de estas pocas metiltransferasas huérfanas de fagos seleccionadas, su propósito biológico aún no está claro. [5]