La versatilidad de la reacción en cadena de la polimerasa (PCR) ha llevado a modificaciones del protocolo básico que se utiliza en una gran cantidad de técnicas variantes diseñadas para diversos fines. Este artículo resume muchas de las variaciones más comunes utilizadas actualmente o anteriormente en los laboratorios de biología molecular ; Es necesario estar familiarizado con la premisa fundamental mediante la cual funciona la PCR y los términos y conceptos correspondientes para comprender estas variantes de las técnicas.
A menudo, sólo es necesario realizar una pequeña modificación en el protocolo de PCR estándar para lograr el objetivo deseado:
Multiplex-PCR utiliza varios pares de cebadores que se hibridan con diferentes secuencias diana. Esto permite el análisis simultáneo de múltiples objetivos en una sola muestra. Por ejemplo, al realizar pruebas de mutaciones genéticas, se pueden combinar seis o más amplificaciones. En el protocolo estándar para la toma de huellas dactilares de ADN , los objetivos analizados a menudo se amplifican en grupos de 3 o 4. La amplificación de sonda dependiente de la ligadura múltiple ( MLPA ) permite amplificar múltiples objetivos utilizando un solo par de cebadores, evitando las limitaciones de resolución de los objetivos múltiples. PCR. La PCR multiplex también se ha utilizado para el análisis de microsatélites y SNP . [1]

"La PCR de número variable de repeticiones en tándem (VNTR) se dirige a áreas repetitivas del genoma que exhiben variación de longitud ". El análisis de los genotipos de las muestras suele implicar el dimensionamiento de los productos de amplificación mediante electroforesis en gel . El análisis de segmentos VNTR más pequeños conocidos como repeticiones cortas en tándem (o STR) es la base de las bases de datos de huellas dactilares de ADN como CODIS .
La PCR asimétrica amplifica preferentemente una hebra de un objetivo de ADN de doble hebra. Se utiliza en algunos métodos de secuenciación y sondas de hibridación para generar una cadena de ADN como producto. El termociclado se realiza exactamente como en la PCR convencional, pero limitando la cantidad o omitiendo uno de los cebadores. Cuando el cebador limitante se agota, la replicación aumenta aritméticamente en lugar de exponencialmente a través de la extensión del cebador en exceso. [2] Una modificación de este proceso, denominada Linear - Después - La - Exponencial -PCR (o LATE-PCR ), utiliza un cebador limitante con una temperatura de fusión más alta (Tm ) que el exceso de cebador para mantener la eficiencia de la reacción a medida que la concentración limitante del cebador disminuye a mitad de la reacción. [3] Véase también PCR de extensión superpuesta .
Se necesitan algunas modificaciones para realizar PCR largas . El proceso de PCR original basado en Klenow no generó productos de más de aproximadamente 400 pb. Sin embargo, la polimerasa Taq puede amplificar objetivos de hasta varios miles de pb de longitud. [4] Desde entonces, los protocolos modificados con la enzima Taq han permitido amplificar objetivos de más de 50 kb. [5]
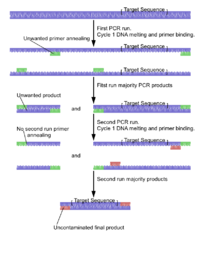
La PCR anidada se utiliza para aumentar la especificidad de la amplificación del ADN. Se utilizan dos conjuntos de cebadores en dos reacciones sucesivas. En la primera PCR, se utiliza un par de cebadores para generar productos de ADN, que pueden contener productos amplificados de áreas no objetivo. Los productos de la primera PCR se utilizan luego como plantilla en una segunda PCR, utilizando uno ("hemi-nesting") o dos cebadores diferentes cuyos sitios de unión están ubicados (anidados) dentro del primer conjunto, aumentando así la especificidad. La PCR anidada suele tener más éxito a la hora de amplificar específicamente productos de ADN largos que la PCR convencional, pero requiere un conocimiento más detallado de la secuencia del objetivo.
La PCR cuantitativa ( qPCR ) se utiliza para medir la cantidad específica de ADN (o ARN) objetivo en una muestra. Al medir la amplificación sólo dentro de la fase de verdadero aumento exponencial, la cantidad de producto medido refleja con mayor precisión la cantidad inicial de objetivo. Se utilizan termocicladores especiales que controlan la cantidad de producto durante la amplificación.
La PCR cuantitativa en tiempo real ( QRT-PCR ), a veces simplemente llamada PCR en tiempo real ( RT-PCR ), se refiere a un conjunto de métodos que utilizan colorantes fluorescentes, como Sybr Green, o sondas de ADN que contienen fluoróforos , como TaqMan. , para medir la cantidad de producto amplificado en tiempo real a medida que avanza la amplificación.
La PCR de inicio en caliente es una técnica que se realiza manualmente calentando los componentes de la reacción hasta la temperatura de fusión del ADN (por ejemplo, 95 °C) antes de agregar la polimerasa. De esta manera se evita una amplificación no específica a temperaturas más bajas. [6] Alternativamente, los reactivos especializados inhiben la actividad de la polimerasa a temperatura ambiente, ya sea mediante la unión de un anticuerpo o por la presencia de inhibidores unidos covalentemente que solo se disocian después de un paso de activación a alta temperatura. La 'PCR de inicio en caliente/acabado en frío' se logra con nuevas polimerasas híbridas que son inactivas a temperatura ambiente y solo se activan a temperaturas elevadas.
En la PCR de aterrizaje , la temperatura de recocido disminuye gradualmente en ciclos posteriores. La temperatura de recocido en los primeros ciclos suele estar entre 3 y 5 °C por encima de la Tm estándar de los cebadores utilizados, mientras que en los ciclos posteriores es una cantidad similar por debajo de la Tm . La temperatura de hibridación inicial más alta conduce a una mayor especificidad para la unión del cebador, mientras que las temperaturas más bajas permiten una amplificación más eficiente al final de la reacción. [7]
La PCR de ensamblaje (también conocida como ensamblaje cíclico de la polimerasa o PCA ) es la síntesis de estructuras largas de ADN mediante la realización de PCR en un conjunto de oligonucleótidos largos con segmentos cortos superpuestos, para ensamblar dos o más piezas de ADN en una sola pieza. Implica una PCR inicial con cebadores que se superponen y una segunda PCR que utiliza los productos como plantilla que genera el producto final completo. Esta técnica puede sustituir el ensamblaje basado en ligadura . [8]
En la PCR de colonias , las colonias bacterianas se examinan directamente mediante PCR, por ejemplo, la detección de construcciones de vectores de ADN correctas . Se toman muestras de las colonias con una punta de pipeta estéril y se transfiere una pequeña cantidad de células a una mezcla de PCR. Para liberar el ADN de las células, la PCR se inicia con un tiempo prolongado a 95 °C (cuando se utiliza la polimerasa estándar) o con un paso de desnaturalización acortado a 100 °C y una ADN polimerasa quimérica especial. [9]
La reacción en cadena de la polimerasa digital amplifica simultáneamente miles de muestras, cada una en una gota separada dentro de una emulsión o partición dentro de un micropocillo.
La PCR suicida se utiliza normalmente en paleogenética u otros estudios donde evitar falsos positivos y garantizar la especificidad del fragmento amplificado es la máxima prioridad. Se describió originalmente en un estudio para verificar la presencia del microbio Yersinia pestis en muestras dentales obtenidas de tumbas del siglo XIV de personas supuestamente muertas por la peste durante la epidemia medieval de Peste Negra . [10] El método prescribe el uso de cualquier combinación de cebadores solo una vez en una PCR (de ahí el término "suicidio"), que nunca debería haberse utilizado en ninguna reacción de PCR de control positivo, y los cebadores siempre deben apuntar a una región genómica, nunca amplificado antes en el laboratorio usando este o cualquier otro conjunto de cebadores. Esto garantiza que no haya ADN contaminante de reacciones de PCR anteriores en el laboratorio, que de otro modo podría generar falsos positivos.
COLD-PCR ( coamplificación a temperatura de desnaturalización más baja -PCR) es un protocolo modificado que enriquece alelos variantes a partir de una mezcla de muestras de ADN de tipo salvaje y que contienen mutaciones.
El proceso básico de PCR a veces puede preceder o seguir a otra técnica.
La RT-PCR (o PCR de transcripcióninversa ) se utiliza para transcribir inversamente y amplificar ARN a ADNc . La PCR va precedida de una reacción que utiliza la transcriptasa inversa , una enzima que convierte el ARN en ADNc. Las dos reacciones se pueden combinar en un tubo, utilizándose el paso de calentamiento inicial de la PCR para inactivar la transcriptasa. [4] La polimerasa Tth (descrita a continuación) tiene actividad RT y puede llevar a cabo toda la reacción. La RT-PCR se utiliza ampliamente en la elaboración de perfiles de expresión , que detecta la expresión de un gen. También se puede utilizar para obtener la secuencia de una transcripción de ARN, lo que puede ayudar a determinar los sitios de inicio y terminación de la transcripción (mediante RACE-PCR ) y facilitar el mapeo de la ubicación de exones e intrones en una secuencia genética.
La PCR de dos colas utiliza un cebador único que se une a un objetivo de microARN con extremos 3' y 5', conocidos como hemisondas. [11] Ambos extremos deben ser complementarios para que se produzca la unión. Luego, la transcriptasa inversa extiende el extremo 3' formando un ADNc largo. Luego, el ADNc se amplifica utilizando dos cebadores de PCR específicos del objetivo. La combinación de dos hemisondas, ambas dirigidas al objetivo de microARN corto, hace que el ensayo de dos colas sea extremadamente sensible y específico.
La PCR mediada por ligación utiliza pequeños 'enlazadores' (o adaptadores) de oligonucleótidos de ADN que primero se ligan a fragmentos del ADN objetivo. Luego se utilizan cebadores de PCR que se hibridan con las secuencias enlazadoras para amplificar los fragmentos diana. Este método se implementa para la secuenciación de ADN, el recorrido del genoma y la huella de ADN . [12] Una técnica relacionada es el polimorfismo de longitud de fragmento amplificado , que genera fragmentos de diagnóstico de un genoma.
La PCR específica de metilación ( MSP ) se utiliza para identificar patrones de metilación del ADN en islas de citosina-guanina (CpG) en el ADN genómico. [13] El ADN objetivo se trata primero con bisulfito de sodio , que convierte las bases de citosina no metiladas en uracilo , que es complementario a la adenosina en los cebadores de PCR. Luego se llevan a cabo dos amplificaciones en el ADN tratado con bisulfito: un conjunto de cebadores se hibrida con ADN con citosinas (correspondiente a citosina metilada) y el otro conjunto se hibrida con ADN con uracilo (correspondiente a citosina no metilada). La MSP utilizada en la PCR cuantitativa proporciona información cuantitativa sobre el estado de metilación de una isla CpG determinada. [14]
Los ajustes de los componentes en la PCR se utilizan comúnmente para lograr un rendimiento óptimo.
El ion magnesio divalente (Mg ++ ) es necesario para la actividad de la polimerasa de la PCR. Concentraciones más bajas de Mg ++ aumentarán la fidelidad de replicación, mientras que concentraciones más altas introducirán más mutaciones. [15]
Los desnaturalizantes (como el DMSO ) pueden aumentar la especificidad de la amplificación al desestabilizar la unión de cebadores no específicos. Otros productos químicos, como el glicerol , son estabilizadores de la actividad de la polimerasa durante la amplificación. Los detergentes (como Triton X-100 ) pueden evitar que la polimerasa se adhiera a sí misma o a las paredes del tubo de reacción.
Las ADN polimerasas ocasionalmente incorporan bases que no coinciden en la cadena en extensión. La PCR de alta fidelidad emplea enzimas con actividad exonucleasa 3'-5' que disminuye esta tasa de incorporación errónea. Ejemplos de enzimas con actividad correctora incluyen Pfu ; Los ajustes de las concentraciones de Mg ++ y dNTP pueden ayudar a maximizar la cantidad de productos que coinciden exactamente con el ADN objetivo original. [ cita necesaria ]
Los ajustes a los oligonucleótidos sintéticos utilizados como cebadores en la PCR son una rica fuente de modificación:
Normalmente, los cebadores de PCR se eligen de una parte invariante del genoma y podrían usarse para amplificar un área polimórfica entre ellos. En la PCR específica de alelo se hace lo contrario. Al menos uno de los cebadores se elige de un área polimórfica, con las mutaciones ubicadas en (o cerca de) su extremo 3'. En condiciones estrictas, un cebador que no coincide no iniciará la replicación, mientras que un cebador coincidente sí lo hará. Por tanto, la aparición de un producto de amplificación indica el genotipo. (Para obtener más información, consulte Genotipado de SNP ).
La PCR específica entre secuencias (o ISSR-PCR ) es un método para la toma de huellas dactilares de ADN que utiliza cebadores seleccionados de segmentos repetidos a lo largo de un genoma para producir una huella dactilar única de longitudes de productos amplificados. [16] El uso de cebadores de un segmento comúnmente repetido se llama Alu-PCR y puede ayudar a amplificar secuencias adyacentes (o entre) estas repeticiones.
Los cebadores también pueden diseñarse para que sean "degenerados", es decir, capaces de iniciar la replicación desde una gran cantidad de ubicaciones objetivo. La amplificación del genoma completo (o WGA ) es un grupo de procedimientos que permiten que se produzca la amplificación en muchos lugares de un genoma desconocido y que puede que solo esté disponible en pequeñas cantidades. Otras técnicas utilizan cebadores degenerados que se sintetizan utilizando múltiples nucleótidos en posiciones particulares (la polimerasa "elige" los cebadores que coinciden correctamente). Además, los cebadores se pueden sintetizar con el análogo de nucleósido inosina , que se hibrida con tres de las cuatro bases normales. Una técnica similar puede obligar a la PCR a realizar una mutagénesis dirigida al sitio . (ver también reacción en cadena de la polimerasa de extensión superpuesta )
Normalmente los cebadores utilizados en la PCR están diseñados para ser totalmente complementarios al objetivo. Sin embargo, la polimerasa es tolerante a desajustes lejos del extremo 3'. Los cebadores con cola incluyen secuencias no complementarias en sus extremos 5'. Un procedimiento común es el uso de enlazadores-cebadores , que finalmente colocan sitios de restricción en los extremos de los productos de la PCR, facilitando su posterior inserción en los vectores de clonación.
Una extensión del método de 'colonia-PCR' (arriba) es el uso de cebadores vectoriales . Primero se insertan fragmentos de ADN diana (o ADNc) en un vector de clonación y se diseña un único conjunto de cebadores para las áreas del vector que flanquean el sitio de inserción. La amplificación se produce para cualquier ADN que se haya insertado. [4]
La PCR se puede modificar fácilmente para producir un producto marcado para uso posterior como sonda de hibridación . Se pueden usar uno o ambos cebadores en la PCR con un marcador radiactivo o fluorescente ya adherido, o se pueden agregar marcadores después de la amplificación. Estos métodos de etiquetado se pueden combinar con la 'PCR asimétrica' (arriba) para producir sondas de hibridación eficaces.
La PCR dependiente de RNasa H (rhPCR) puede reducir la formación de dímeros de cebador y aumentar el número de ensayos en la PCR múltiple. El método utiliza cebadores con un bloque escindible en el extremo 3' que se elimina mediante la acción de una enzima RNasa HII termoestable. [17]
Hay varias ADN polimerasas que se utilizan en la PCR.
El fragmento Klenow , derivado de la ADN polimerasa I original de E. coli , fue la primera enzima utilizada en la PCR. Debido a su falta de estabilidad a altas temperaturas, es necesario reponerlo durante cada ciclo y, por lo tanto, no se usa comúnmente en PCR.
La ADN polimerasa del bacteriófago T4 (familia A) también se utilizó inicialmente en la PCR. Tiene una mayor fidelidad de replicación que el fragmento de Klenow, pero también se destruye con el calor. La ADN polimerasa T7 (familia B) tiene propiedades y propósitos similares. Se ha aplicado a la mutagénesis dirigida [18] y a la secuenciación de Sanger . [19]
La Taq polimerasa, la ADN polimerasa I de Thermus Aquaticus , fue la primera polimerasa termoestable utilizada en PCR y sigue siendo la más utilizada. La enzima se puede aislar de su fuente nativa o de su gen clonado expresado en E. coli . [4] Un fragmento de 61 kDa truncado por carecer de actividad exonucleasa 5'-3' se conoce como fragmento Stoffel y se expresa en E. coli . [20] La falta de actividad exonucleasa puede permitirle amplificar objetivos más largos que la enzima nativa. Se ha comercializado como AmpliTaq y Klentaq . [21] También se ha producido una variante diseñada para PCR de inicio en caliente llamada "polimerasa Faststart". Requiere una fuerte activación por calor, evitando así la amplificación no específica debida a la actividad de la polimerasa a baja temperatura.Se han creado muchas otras variantes . [22]
Otras Thermus polimerasas, como la Tth polimerasa I ( P52028 ) de Thermus thermophilus , han tenido algún uso. Tth tiene actividad transcriptasa inversa en presencia de iones Mn 2+ , lo que permite la amplificación por PCR a partir de objetivos de ARN. [23]
El género arcaico Pyrococcus ha demostrado ser una rica fuente de polimerasas termoestables con actividad correctora. La ADN polimerasa Pfu , aislada de P. furiosus, muestra una disminución de 5 veces en la tasa de error de replicación en comparación con Taq. [24] Dado que los errores aumentan a medida que avanza la PCR, Pfu es la polimerasa preferida cuando los productos deben clonarse individualmente para secuenciación o expresión. Otras polimerasas menos utilizadas de este género incluyen Pwo ( P61876 ) de Pyrococcus woesei , Pfx de una especie sin nombre, polimerasa "Deep Vent" ( Q51334 ) de la cepa GB-D. [25]
Vent o Tli polimerasa es una ADN polimerasa extremadamente termoestable aislada de Thermococcus litoralis . También se ha comercializado la polimerasa de Thermococcus fumicolans ( Tfu ). [25]
A veces incluso se puede modificar el mecanismo básico de la PCR.

A diferencia de la PCR normal, la PCR inversa permite la amplificación y secuenciación del ADN que rodea una secuencia conocida. Implica someter inicialmente el ADN objetivo a una serie de digestiones con enzimas de restricción y luego circularizar los fragmentos resultantes mediante autoligación . Los cebadores están diseñados para extenderse hacia afuera del segmento conocido, lo que resulta en la amplificación del resto del círculo. Esto es especialmente útil para identificar secuencias a ambos lados de varios insertos genómicos. [26]
De manera similar, la PCR entrelazada térmicamente asimétrica (o TAIL-PCR ) se utiliza para aislar secuencias desconocidas que flanquean un área conocida del genoma. Dentro de la secuencia conocida, TAIL-PCR utiliza un par anidado de cebadores con diferentes temperaturas de hibridación. Se utiliza un cebador "degenerado" para amplificar en la dirección opuesta a la secuencia desconocida. [27]
Se han desarrollado algunos protocolos de amplificación de ADN que pueden usarse como alternativa a la PCR. Son isotérmicos, lo que significa que funcionan a una temperatura constante. [28]
La amplificación dependiente de helicasa (HDA) es similar a la PCR tradicional, pero utiliza una temperatura constante en lugar de realizar ciclos a través de pasos de desnaturalización y hibridación/extensión. La ADN helicasa , una enzima que desenrolla el ADN, se utiliza en lugar de la desnaturalización térmica. [29] La amplificación isotérmica mediada por bucles es una idea similar, pero realizada con una polimerasa que desplaza las cadenas. [30]
La reacción de amplificación de la enzima de mellado (NEAR) y su prima, la amplificación por desplazamiento de cadena (SDA), son isotérmicas y replican el ADN a una temperatura constante utilizando una polimerasa y una enzima de mellado. [28]
La amplificación de la polimerasa por recombinasa (RPA) [31] utiliza una recombinasa para emparejar específicamente cebadores con ADN bicatenario sobre la base de la homología, dirigiendo así la síntesis de ADN a partir de secuencias definidas de ADN presentes en la muestra. La presencia de la secuencia objetivo inicia la amplificación del ADN y no se requiere fusión térmica o química del ADN. La reacción progresa rápidamente y da como resultado una amplificación específica del ADN desde unas pocas copias objetivo hasta niveles detectables, por lo general en 5 a 10 minutos. Todo el sistema de reacción es estable como formulación seca y no necesita refrigeración. La RPA se puede utilizar para reemplazar la PCR en una variedad de aplicaciones de laboratorio y los usuarios pueden diseñar sus propios ensayos. [32]
Otros tipos de amplificación isotérmica incluyen la amplificación del genoma completo (WGA), la amplificación basada en secuencias de ácidos nucleicos (NASBA) y la amplificación mediada por transcripción (TMA). [28]
{{cite book}}: |journal=ignorado ( ayuda )