El genotipado de SNP es la medición de variaciones genéticas de polimorfismos de un solo nucleótido (SNP) entre miembros de una especie. Es una forma de genotipado , que es la medición de una variación genética más general. Los SNP son uno de los tipos más comunes de variación genética. Un SNP es una mutación de un solo par de bases en un locus específico , que generalmente consta de dos alelos (donde la frecuencia alélica rara es> 1%). Se ha descubierto que los SNP están implicados en la etiología de muchas enfermedades humanas y están adquiriendo especial interés en la farmacogenética . Debido a que los SNP se conservan durante la evolución, se han propuesto como marcadores para su uso en análisis de loci de rasgos cuantitativos ( QTL ) y en estudios de asociación en lugar de microsatélites . El uso de SNP se está ampliando en el proyecto HapMap , cuyo objetivo es proporcionar el conjunto mínimo de SNP necesario para genotipar el genoma humano. Los SNP también pueden proporcionar una huella genética para su uso en pruebas de identidad. [1] El aumento del interés en los SNP se ha reflejado en el furioso desarrollo de una amplia gama de métodos de genotipado de SNP.
Se han desarrollado varias aplicaciones que interrogan a los SNP mediante la hibridación de sondas de ADN complementarias con el sitio del SNP. El desafío de este enfoque es reducir la hibridación cruzada entre las sondas específicas de alelo. Este desafío generalmente se supera manipulando las condiciones estrictas de hibridación. [1]

El genotipado de hibridación dinámica específica de alelo (DASH) aprovecha las diferencias en la temperatura de fusión del ADN que resultan de la inestabilidad de los pares de bases no coincidentes. El proceso puede automatizarse enormemente y abarca algunos principios simples. [ cita necesaria ]
En el primer paso, se amplifica un segmento genómico y se une a una perla mediante una reacción de PCR con un cebador biotinilado. En el segundo paso, el producto amplificado se une a una columna de estreptavidina y se lava con NaOH para eliminar la cadena no biotinilada. Luego se agrega un oligonucleótido específico de alelo en presencia de una molécula que emite fluorescencia cuando se une al ADN bicatenario. Luego se mide la intensidad a medida que aumenta la temperatura hasta que se puede determinar la temperatura de fusión (Tm) . Un SNP dará como resultado una Tm inferior a la esperada. [2]
Debido a que el genotipado DASH mide un cambio cuantificable en Tm, es capaz de medir todo tipo de mutaciones, no solo los SNP. Otros beneficios de DASH incluyen su capacidad para trabajar con sondas sin etiquetas y su diseño simple y condiciones de rendimiento. [ cita necesaria ]
La detección de SNP a través de balizas moleculares utiliza una sonda de oligonucleótidos monocatenario diseñada específicamente. El oligonucleótido está diseñado de manera que haya regiones complementarias en cada extremo y una secuencia de sonda ubicada en el medio. Este diseño permite que la sonda adopte una estructura de horquilla o bucle de vástago en su estado natural y aislado. Unido a un extremo de la sonda hay un fluoróforo y al otro extremo un extintor de fluorescencia. Debido a la estructura de vástago-bucle de la sonda, el fluoróforo está cerca del extintor, evitando así que la molécula emita fluorescencia. La molécula también está diseñada de manera que sólo la secuencia de la sonda sea complementaria al ADN genómico que se utilizará en el ensayo (Abravaya et al. 2003).
Si la secuencia de la sonda de la baliza molecular encuentra su ADN genómico objetivo durante el ensayo, se apareará e hibridará. Debido a la longitud de la secuencia de la sonda, el segmento en horquilla de la sonda se desnaturalizará a favor de formar un híbrido sonda-objetivo más largo y estable. Este cambio conformacional permite que el fluoróforo y el extintor estén libres de su estrecha proximidad debido a la asociación en horquilla, lo que permite que la molécula emita fluorescencia.
Si, por otro lado, la secuencia de la sonda encuentra una secuencia objetivo con tan solo un nucleótido no complementario, la baliza molecular permanecerá preferentemente en su estado de horquilla natural y no se observará fluorescencia, ya que el fluoróforo permanece apagado.
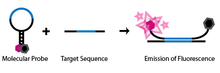
El diseño único de estas balizas moleculares permite un ensayo de diagnóstico simple para identificar SNP en una ubicación determinada. Si se diseña una baliza molecular para que coincida con un alelo de tipo salvaje y otra para que coincida con un mutante del alelo, las dos pueden usarse para identificar el genotipo de un individuo. Si durante el ensayo sólo se detecta la longitud de onda del fluoróforo de la primera sonda, entonces el individuo es homocigoto con respecto al tipo salvaje. Si sólo se detecta la longitud de onda de la segunda sonda, entonces el individuo es homocigoto al alelo mutante. Finalmente, si se detectan ambas longitudes de onda, entonces ambas balizas moleculares deben estar hibridando con sus complementos y, por lo tanto, el individuo debe contener ambos alelos y ser heterocigoto.
En las matrices de SNP de oligonucleótidos de alta densidad , cientos de miles de sondas se colocan en un pequeño chip, lo que permite interrogar muchos SNP simultáneamente. [1] Debido a que los alelos de SNP solo difieren en un nucleótido y debido a que es difícil lograr condiciones de hibridación óptimas para todas las sondas en la matriz, el ADN objetivo tiene el potencial de hibridarse con sondas que no coinciden. Esto se soluciona en cierta medida mediante el uso de varias sondas redundantes para interrogar a cada SNP. Las sondas están diseñadas para tener el sitio SNP en varias ubicaciones diferentes, además de contener discrepancias con el alelo SNP. Comparando la cantidad diferencial de hibridación del ADN diana con cada una de estas sondas redundantes, es posible determinar alelos homocigotos y heterocigotos específicos. [1] Aunque los microarrays de oligonucleótidos tienen una especificidad y sensibilidad comparativamente más bajas, la escala de SNP que se pueden interrogar es un beneficio importante. El GeneChip Affymetrix Human SNP 5.0 realiza un ensayo de todo el genoma que puede genotipar más de 500.000 SNP humanos (Affymetrix 2007).
Se ha empleado una amplia gama de enzimas, incluidas la ADN ligasa , la ADN polimerasa y las nucleasas , para generar métodos de genotipado de SNP de alta fidelidad.
El polimorfismo de longitud de fragmentos de restricción (RFLP) se considera el método más simple y temprano para detectar SNP. SNP-RFLP utiliza muchas endonucleasas de restricción diferentes y su alta afinidad por sitios de restricción únicos y específicos. Al realizar una digestión en una muestra genómica y determinar la longitud de los fragmentos mediante un ensayo en gel, es posible determinar si las enzimas cortan o no los sitios de restricción esperados. Si no se corta la muestra genómica, se obtiene un fragmento identificablemente más grande de lo esperado, lo que implica que hay una mutación en el punto del sitio de restricción que le otorga protección contra la actividad nucleasa.
Los factores combinados de la alta complejidad de la mayoría de los genomas eucariotas, el requisito de endonucleasas específicas, el hecho de que la mutación exacta no necesariamente puede resolverse en un solo experimento y la naturaleza lenta de los ensayos en gel hacen del RFLP una mala elección para análisis de alto rendimiento.
El sistema de mutación refractaria de amplificación de tetracebadores, o ARMS-PCR, emplea dos pares de cebadores para amplificar dos alelos en una reacción de PCR. Los cebadores están diseñados de manera que los dos pares de cebadores se superpongan en una ubicación de SNP pero cada uno coincida perfectamente con solo uno de los posibles SNP. La base de la invención es que, inesperadamente, los oligonucleótidos con un residuo 3' no coincidente no funcionarán como cebadores en la PCR en condiciones apropiadas. [3] Como resultado, si un alelo determinado está presente en la reacción de PCR, el par de cebadores específicos de ese alelo producirá un producto, pero no el alelo alternativo con un SNP diferente. Los dos pares de cebadores también están diseñados de manera que sus productos de PCR tengan una longitud significativamente diferente, lo que permite bandas fácilmente distinguibles mediante electroforesis en gel o análisis de temperatura de fusión. [4] [5] Al examinar los resultados, si una muestra genómica es homocigota, entonces los productos de PCR resultantes serán del cebador que coincide con la ubicación del SNP y el cebador externo de cadena opuesta, así como de los dos cebadores externos. Si la muestra genómica es heterocigota, entonces los productos resultarán del cebador de cada alelo y sus respectivas contrapartes del cebador externo, así como de los cebadores externos.
Una estrategia alternativa es ejecutar múltiples reacciones de qPCR con diferentes conjuntos de cebadores que se dirijan a cada alelo por separado. Los cebadores bien diseñados amplificarán su SNP objetivo en un ciclo mucho más temprano que los otros SNP. Esto permite distinguir más de dos alelos, aunque se requiere una reacción qPCR individual para cada SNP. Para lograr una especificidad suficientemente alta, la secuencia del cebador puede requerir la colocación de un desajuste artificial cerca de su extremo 3', lo cual es un enfoque generalmente conocido como Taq-MAMA. [6]

La endonucleasa flap (FEN) es una endonucleasa que cataliza la escisión de estructuras específicas. Esta escisión es muy sensible a los desajustes y puede usarse para interrogar a los SNP con un alto grado de especificidad [7]
En el ensayo básico Invader, un FEN llamado escisión se combina con dos sondas oligonucleotídicas específicas que, junto con el ADN objetivo, pueden formar una estructura tripartita reconocida por la escisión. [7] La primera sonda, llamada oligonucleótido invasor , es complementaria al extremo 3' del ADN objetivo. La última base del oligonucleótido Invader es una base no coincidente que se superpone al nucleótido SNP en el ADN objetivo. La segunda sonda es una sonda específica de alelo que es complementaria al extremo 5' del ADN diana, pero que también se extiende más allá del lado 3' del nucleótido SNP. La sonda específica de alelo contendrá una base complementaria al nucleótido del SNP. Si el ADN objetivo contiene el alelo deseado, las sondas invasoras y específicas de alelo se unirán al ADN objetivo formando la estructura tripartita. Esta estructura es reconocida por la escisión, que escinderá y liberará el extremo 3' de la sonda específica del alelo. Si el nucleótido SNP en el ADN diana no es complementario a la sonda específica del alelo, no se forma la estructura tripartita correcta y no se produce escisión. El ensayo Invader suele combinarse con un sistema de transferencia de energía por resonancia de fluorescencia (FRET) para detectar el evento de escisión. En esta configuración, se une una molécula extintora al extremo 3' y se une un fluoróforo al extremo 5' de la sonda específica del alelo. Si se produce una escisión, el fluoróforo se separará de la molécula extintora generando una señal detectable. [7]
Sólo se produce una escisión mínima con sondas no coincidentes, lo que hace que el ensayo Invader sea muy específico. Sin embargo, en su formato original, solo se podía interrogar un alelo SNP por muestra de reacción y se requería una gran cantidad de ADN objetivo para generar una señal detectable en un período de tiempo razonable. [7] Varios desarrollos han ampliado el ensayo Invader original . Al llevar a cabo reacciones secundarias de escisión de FEN, la reacción de amplificación de señales invasivas en serie (SISAR) permite interrogar ambos alelos de SNP en una sola reacción. El ensayo SISAR Invader también requiere menos ADN objetivo, lo que mejora la sensibilidad del ensayo Invader original . [7] El ensayo también se ha adaptado de varias maneras para su uso en un formato de alto rendimiento. En una plataforma, las sondas específicas de alelos están ancladas a microesferas. Cuando la escisión por FEN genera una señal fluorescente detectable, la señal se mide mediante citometría de flujo. La sensibilidad de la citometría de flujo elimina la necesidad de amplificación por PCR del ADN objetivo (Rao et al. 2003). Estas plataformas de alto rendimiento no han avanzado más allá de la etapa de prueba de principio y hasta ahora el sistema Invader no se ha utilizado en ningún proyecto de genotipado de SNP a gran escala. [7]
La extensión del cebador es un proceso de dos pasos que primero implica la hibridación de una sonda con las bases inmediatamente aguas arriba del nucleótido SNP, seguida de una reacción de "minisecuenciación", en la que la ADN polimerasa extiende el cebador hibridado agregando una base que es complementaria a el nucleótido SNP. Esta base incorporada se detecta y determina el alelo SNP (Goelet et al. 1999; Syvanen 2001). Debido a que la extensión del cebador se basa en la enzima ADN polimerasa de alta precisión, el método es generalmente muy confiable. La extensión del cebador es capaz de genotipar la mayoría de los SNP en condiciones de reacción muy similares, lo que también lo hace muy flexible. El método de extensión del cebador se utiliza en varios formatos de ensayo. Estos formatos utilizan una amplia gama de técnicas de detección que incluyen espectrometría de masas MALDI-TOF (ver Sequenom ) y métodos similares a ELISA . [1]
Generalmente, existen dos enfoques principales que utilizan la incorporación de didesoxinucleótidos marcados con fluorescencia (ddNTP) o desoxinucleótidos marcados con fluorescencia (dNTP). Con los ddNTP, las sondas se hibridan con el ADN objetivo inmediatamente aguas arriba del nucleótido SNP, y se agrega un único ddNTP complementario al alelo SNP al extremo 3' de la sonda (el 3'-hidroxilo faltante en el didoxinucleótido evita que se agreguen más nucleótidos). ). Cada ddNTP está etiquetado con una señal fluorescente diferente que permite la detección de los cuatro alelos en la misma reacción. Con los dNTP, las sondas específicas de alelo tienen bases 3' que son complementarias a cada uno de los alelos del SNP que se interrogan. Si el ADN objetivo contiene un alelo complementario a la base 3' de la sonda, el ADN objetivo se hibridará completamente con la sonda, permitiendo que la ADN polimerasa se extienda desde el extremo 3' de la sonda. Esto se detecta mediante la incorporación de dNTP marcados con fluorescencia en el extremo de la sonda. Si el ADN objetivo no contiene un alelo complementario a la base 3' de la sonda, el ADN objetivo producirá una falta de coincidencia en el extremo 3' de la sonda y la ADN polimerasa no podrá extenderse desde el extremo 3' de la sonda. El beneficio del segundo enfoque es que se pueden incorporar varios dNTP marcados en la cadena en crecimiento, lo que permite una mayor señal. Sin embargo, en algunos casos raros, la ADN polimerasa puede extenderse desde sondas 3' no coincidentes dando un resultado falso positivo. [1]
El método de genotipado iPLEX SNP de Sequenom utiliza un enfoque diferente , que utiliza un espectrómetro de masas MassARRAY. Las sondas de extensión están diseñadas de tal manera que se pueden amplificar y analizar 40 ensayos de SNP diferentes en un cóctel de PCR. La reacción de extensión utiliza ddNTP como se indicó anteriormente, pero la detección del alelo SNP depende de la masa real del producto de extensión y no de una molécula fluorescente. Este método es para un rendimiento bajo a medio alto y no está diseñado para escanear el genoma completo.
La flexibilidad y especificidad de la extensión del cebador la hacen susceptible de análisis de alto rendimiento. Las sondas de extensión del cebador se pueden colocar en portaobjetos, lo que permite genotipar muchos SNP a la vez. Esta tecnología, ampliamente conocida como extensión de cebadores en matriz (APEX), tiene varias ventajas sobre los métodos basados en la hibridación diferencial de sondas. Comparativamente, los métodos APEX tienen un mayor poder de discriminación que los métodos que utilizan esta hibridación diferencial, ya que a menudo es imposible obtener las condiciones óptimas de hibridación para las miles de sondas en los microarrays de ADN (normalmente esto se soluciona teniendo sondas altamente redundantes). Sin embargo, no se puede lograr la misma densidad de sondas en los métodos APEX, lo que se traduce en una menor producción por ejecución. [1]
El ensayo Infinium de Illumina Incorporated es un ejemplo de un proceso de genotipado de genoma completo que se basa en el método de extensión de cebadores. En el ensayo Infinium, se pueden genotipar más de 100.000 SNP. El ensayo utiliza nucleótidos marcados con hapteno en una reacción de extensión del cebador. El marcador hapteno es reconocido por los anticuerpos, que a su vez se acoplan a una señal detectable (Gunderson et al. 2006).
APEX-2 es un método de genotipado de extensión de cebadores en matriz que es capaz de identificar cientos de SNP o mutaciones en paralelo utilizando una PCR múltiplex homogénea eficiente (hasta 640 plex) y una extensión de base única de cuatro colores en una micromatriz. La PCR múltiple requiere dos oligonucleótidos por SNP/mutación que generan amplicones que contienen el par de bases probado. Los mismos oligonucleótidos se utilizan en el siguiente paso como cebadores de extensión de base única inmovilizados en una micromatriz (Krjutskov et al. 2008).
La actividad 5'-nucleasa de la ADN polimerasa Taq se utiliza en el ensayo TaqMan para el genotipado de SNP. El ensayo TaqMan se realiza simultáneamente con una reacción de PCR y los resultados se pueden leer en tiempo real a medida que avanza la reacción de PCR (McGuigan & Ralston 2002). El ensayo requiere cebadores de PCR directos e inversos que amplificarán una región que incluye el sitio polimórfico del SNP. La discriminación de alelos se logra utilizando FRET combinado con una o dos sondas específicas de alelo que se hibridan con el sitio polimórfico del SNP. Las sondas tendrán un fluoróforo unido a su extremo 5' y una molécula extintora unida a su extremo 3'. Mientras la sonda esté intacta, el extintor permanecerá muy cerca del fluoróforo, eliminando la señal del fluoróforo. Durante el paso de amplificación por PCR, si la sonda específica del alelo es perfectamente complementaria al alelo SNP, se unirá a la cadena de ADN objetivo y luego se degradará por la actividad 5'-nucleasa de la polimerasa Taq a medida que extiende el ADN de la PCR. cebadores. La degradación de la sonda da como resultado la separación del fluoróforo de la molécula extintora, generando una señal detectable. Si la sonda específica del alelo no es perfectamente complementaria, tendrá una temperatura de fusión más baja y no se unirá con tanta eficacia. Esto evita que la nucleasa actúe sobre la sonda (McGuigan y Ralston 2002).
Dado que el ensayo TaqMan se basa en PCR, es relativamente sencillo de implementar. El ensayo TaqMan se puede multiplexar combinando la detección de hasta siete SNP en una reacción. Sin embargo, dado que cada SNP requiere una sonda distinta, el ensayo TaqMan está limitado por la proximidad con la que se pueden situar los SNP. La escala del ensayo se puede aumentar drásticamente realizando muchas reacciones simultáneas en placas de microtitulación. Generalmente, TaqMan se limita a aplicaciones que implican interrogar una pequeña cantidad de SNP, ya que se deben diseñar sondas y condiciones de reacción óptimas para cada SNP (Syvanen 2001).
La ADN ligasa cataliza la ligación del extremo 3' de un fragmento de ADN al extremo 5' de un fragmento de ADN directamente adyacente. Este mecanismo se puede utilizar para interrogar un SNP hibridando dos sondas directamente sobre el sitio polimórfico del SNP, mediante lo cual puede producirse la ligación si las sondas son idénticas al ADN objetivo. En el ensayo de oligonucleótido ligasa, se diseñan dos sondas; una sonda específica de alelo que se hibrida con el ADN diana de modo que su base 3' esté situada directamente sobre el nucleótido del SNP y una segunda sonda que hibrida el molde aguas arriba (aguas abajo en la cadena complementaria) del sitio polimórfico del SNP proporcionando un extremo 5' para la reacción de ligación. Si la sonda específica del alelo coincide con el ADN objetivo, se hibridará completamente con el ADN objetivo y se podrá producir la ligación. La ligación generalmente no ocurre en presencia de una base 3' no coincidente. Los productos ligados o no ligados se pueden detectar mediante electroforesis en gel, espectrometría de masas MALDI-TOF o electroforesis capilar para aplicaciones a gran escala. [1] Con secuencias y etiquetas apropiadas en los oligonucleótidos, se pueden generar datos de secuencia de alto rendimiento a partir de los productos ligados y los genotipos determinados (Curry et al., 2012). El uso de una gran cantidad de índices de muestra permite generar datos de secuencia de alto rendimiento en cientos de SNP en miles de muestras en una pequeña porción de una ejecución de secuenciación de alto rendimiento. Se trata de un genotipado masivo mediante tecnología de secuenciación (MGST). [ cita necesaria ]
Las propiedades características del ADN de temperatura de fusión y conformación monocatenaria se han utilizado en varias aplicaciones para distinguir alelos de SNP. Estos métodos suelen lograr una alta especificidad, pero requieren condiciones altamente optimizadas para obtener los mejores resultados posibles.
El ADN monocatenario (ssDNA) se pliega en una estructura terciaria. La conformación depende de la secuencia y la mayoría de las mutaciones de un solo par de bases alterarán la forma de la estructura. Cuando se aplica a un gel, la forma terciaria determinará la movilidad del ssDNA, proporcionando un mecanismo para diferenciar entre alelos SNP. Este método implica primero la amplificación por PCR del ADN objetivo. Los productos de PCR de doble cadena se desnaturalizan utilizando calor y formaldehído para producir ssDNA. El ssDNA se aplica a un gel de electroforesis no desnaturalizante y se deja plegar en una estructura terciaria. Las diferencias en la secuencia del ADN alterarán la conformación terciaria y se detectarán como una diferencia en la movilidad de la cadena del ADNss (Costabile et al. 2006). Este método se utiliza ampliamente porque es técnicamente sencillo, relativamente económico y utiliza equipos comúnmente disponibles. Sin embargo, en comparación con otros métodos de genotipado de SNP, la sensibilidad de este ensayo es menor. Se ha descubierto que la conformación del ssDNA depende en gran medida de la temperatura y, en general, no es evidente cuál es la temperatura ideal. Muy a menudo el ensayo se llevará a cabo utilizando varias temperaturas diferentes. También existe una restricción en la longitud del fragmento porque la sensibilidad cae cuando se utilizan secuencias de más de 400 pb (Costabile et al. 2006).

El método de electroforesis en gel con gradiente de temperatura (TGGE) o electroforesis capilar con gradiente de temperatura (TGCE) se basa en el principio de que el ADN parcialmente desnaturalizado está más restringido y viaja más lentamente en un material poroso como un gel. Esta propiedad permite la separación del ADN por temperatura de fusión. Para adaptar estos métodos para la detección de SNP, se utilizan dos fragmentos; el ADN diana que contiene el sitio polimórfico del SNP que se está interrogando y una secuencia de ADN específica del alelo, denominada fragmento de ADN normal. El fragmento normal es idéntico al ADN diana excepto potencialmente en el sitio polimórfico del SNP, que se desconoce en el ADN diana. Los fragmentos se desnaturalizan y luego se vuelven a recocer. Si el ADN diana tiene el mismo alelo que el fragmento normal, se formarán homodúplex que tendrán la misma temperatura de fusión. Cuando se ejecuta sobre el gel con un gradiente de temperatura, solo aparecerá una banda. Si el ADN objetivo tiene un alelo distinto, se formarán cuatro productos después del paso de rehibrilación; homodúplex que consisten en ADN diana, homodúplex que consisten en ADN normal y dos heterodúplex de cada hebra de ADN diana hibridada con la hebra de ADN normal. Estos cuatro productos tendrán distintas temperaturas de fusión y aparecerán como cuatro bandas en el gel desnaturalizante. [1]
La cromatografía líquida desnaturalizante de alto rendimiento (DHPLC) utiliza HPLC de fase reversa para interrogar los SNP. La clave de la DHPLC es la fase sólida que tiene afinidad diferencial por el ADN monocatenario y bicatenario. En DHPLC, los fragmentos de ADN se desnaturalizan calentándolos y luego se les permite volver a hibridarse. La temperatura de fusión de los fragmentos de ADN rehibridados determina el tiempo que permanecen retenidos en la columna. [8] Utilizando PCR, se generan dos fragmentos; ADN diana que contiene el sitio polimórfico del SNP y una secuencia de ADN específica del alelo, denominada fragmento de ADN normal. Este fragmento normal es idéntico al ADN diana excepto potencialmente en el sitio polimórfico del SNP, que se desconoce en el ADN diana. Los fragmentos se desnaturalizan y luego se les permite reasociarse gradualmente. Los productos reanulados se añaden a la columna DHPLC. Si el alelo SNP en el ADN objetivo coincide con el fragmento de ADN normal, sólo se formarán homodúplex idénticos durante el paso de rehibrilación. Si el ADN diana contiene un alelo SNP diferente al fragmento de ADN normal, se formarán heterodúplex del ADN diana y del ADN normal que contiene un sitio polimórfico no coincidente, además de los homodúplex. Los heterodúplex no coincidentes tendrán una temperatura de fusión diferente a la de los homodúplex y no serán retenidos en la columna por tanto tiempo. Esto genera un patrón cromatográfico que se distingue del patrón que se generaría si el fragmento de ADN objetivo y los fragmentos de ADN normales fueran idénticos. El ADN eluido se detecta mediante absorción UV. [8]
DHPLC se automatiza fácilmente ya que no es necesario marcar ni purificar los fragmentos de ADN. El método también es relativamente rápido y tiene una alta especificidad. Un inconveniente importante de DHPLC es que la temperatura de la columna debe optimizarse para cada objetivo para lograr el grado correcto de desnaturalización. [1]
El análisis de fusión de alta resolución es el método basado en PCR más sencillo de entender. Básicamente, las mismas propiedades termodinámicas que permitieron que funcionaran las técnicas de gel se aplican aquí y en tiempo real. Un fluorímetro monitorea la desnaturalización post-PCR de todo el amplicón de ADNbc. Usted elabora cebadores específicos para el sitio que desea amplificar. Se "pinta" el amplicón con un tinte específico de doble hebra, incluido en la mezcla de PCR. El colorante específico de ds se integra en el producto de PCR. En esencia, todo el amplicón se convierte en una sonda. Esto abre nuevas posibilidades de descubrimiento. O bien coloca los cebadores muy cerca de cada lado del SNP en cuestión (genotipado de amplicones pequeños, Liew, 2004) o amplifica una región más grande (de 100 a 400 pb de longitud) para fines de escaneo. Para el genotipado simple de un SNP, es más fácil hacer que el amplicón sea pequeño para minimizar las posibilidades de confundir un SNP con otro. Se determina la temperatura de fusión (Tm) de todo el amplicón y la mayoría de los homocigotos son suficientemente diferentes (en los mejores instrumentos) en Tm para genotipar. Los heterocigotos son aún más fáciles de diferenciar porque tienen heterodúplex generados (consulte las explicaciones basadas en gel) que amplían la transición de fusión y generalmente dan dos picos discernibles. Se ha descrito la fusión de amplicones utilizando un cebador marcado con fluorescencia (Gundry et al., 2003), pero es menos práctica que usar tintes específicos de ds debido al costo del cebador fluorogénico.
La exploración de amplicones más grandes se basa en los mismos principios descritos anteriormente. Sin embargo, la temperatura de fusión y la forma general de la curva de fusión resultan informativas. Para amplicones >c.150 pb, a menudo hay >2 picos de fusión, cada uno de los cuales puede variar, dependiendo de la composición del molde de ADN. Numerosos investigadores han podido eliminar con éxito la mayor parte de su secuenciación mediante escaneo basado en fusión, lo que permite una genotipificación precisa basada en locus de un gran número de individuos. [9] Muchos investigadores han descubierto que la exploración en busca de mutaciones mediante fusión de alta resolución es una forma viable y práctica de estudiar genes completos.
Las proteínas de unión a errores de coincidencia del ADN pueden distinguir errores de coincidencia de un solo nucleótido y, por lo tanto, facilitar el análisis diferencial de los SNP. Por ejemplo, la proteína MutS de Thermus acuáticos se une a diferentes desajustes de un solo nucleótido con diferentes afinidades y puede usarse en electroforesis capilar para diferenciar los seis conjuntos de desajustes (Drabovich y Krylov 2006).
SNPlex es una plataforma de genotipado patentada vendida por Applied Biosystems .
La nucleasa Surveyor es una enzima endonucleasa que no coincide que reconoce todas las sustituciones de bases y pequeñas inserciones/eliminaciones (indeles) y escinde el lado 3' de los sitios no coincidentes en ambas cadenas de ADN.
Las tecnologías de secuenciación de próxima generación , como la pirosecuenciación, secuencian menos de 250 bases en una lectura, lo que limita su capacidad para secuenciar genomas completos. Sin embargo, su capacidad para generar resultados en tiempo real y su potencial para ampliarse masivamente los convierte en una opción viable para secuenciar regiones pequeñas para realizar el genotipado de SNP. En comparación con otros métodos de genotipado de SNP, la secuenciación es en particular adecuada para identificar múltiples SNP en una región pequeña, como la región del genoma del Complejo Mayor de Histocompatibilidad altamente polimórfica. [1]