Un gen gap es un tipo de gen que interviene en el desarrollo de los embriones segmentados de algunos artrópodos . Los genes gap se definen por el efecto de una mutación en ese gen, que provoca la pérdida de segmentos corporales contiguos, asemejándose a un hueco en el plan corporal normal. Por tanto, cada gen gap es necesario para el desarrollo de una sección del organismo.

Los genes gap fueron descritos por primera vez por Christiane Nüsslein-Volhard y Eric Wieschaus en 1980. [1] Utilizaron un análisis genético para identificar los genes necesarios para el desarrollo embrionario en la mosca de la fruta Drosophila melanogaster . Encontraron tres genes ( knirps, Krüppel y hunchback ) en los que las mutaciones causaron la eliminación de tramos particulares de segmentos. Un trabajo posterior identificó más genes gap en el embrión temprano de Drosophila ( gigante , huckebein y sin cola) . [2] [3] Otros genes gap, incluidos orthodenticle y buttonhead, son necesarios para el desarrollo de la cabeza de Drosophila .
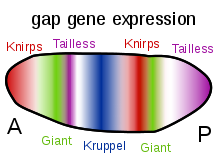
Una vez que los genes gap se habían identificado a nivel molecular, se encontró que cada gen gap se expresa en una banda en el embrión temprano generalmente correlacionada con la región que está ausente en el mutante. [4] [5] En Drosophila, los genes gap codifican factores de transcripción , y controlan directamente la expresión de otro conjunto de genes involucrados en la segmentación, los genes de regla de pares . [6] [7] Los genes gap en sí mismos se expresan bajo el control de genes de efecto materno como bicoid y nanos , y se regulan entre sí para lograr sus patrones de expresión precisos.
La expresión de tailless es activada por la proteína torso en los polos del embrión. Tailless también está regulada de manera compleja por el gen de efecto maternal bicoid.
Tanto la proteína jorobada transcrita embrionariamente como la transcrita maternamente son activadas por la proteína bicoid en la parte anterior y son inhibidas en la parte posterior por la proteína nanos. La proteína jorobada transcrita embrionariamente puede exhibir los mismos efectos en Krüppel y knirps que la proteína jorobada transcrita maternamente.
El gen Krüppel se activa cuando el gradiente de proteína bicoid disminuye abruptamente, en la parte central del embrión. Krüppel está regulado por cinco proteínas reguladoras: bicoid, hunchback, tailless, knirps y giant. Krüppel es inhibido por altos niveles de hunchback, altos niveles de giant y tailless, lo que establece el límite anterior de la expresión de Krüppel. Krüppel también es inhibido por knirps y activado por bajos niveles de bicoid y bajos niveles de hunchback, lo que establece el límite posterior de la expresión de Krüppel.
El gen knirps parece activarse espontáneamente. Es reprimido por hunchback. La represión de hunchback define así el límite anterior del gen knirps. Debido a que hunchback inhibe de manera más eficiente el gen knirps, este se expresa más posteriormente en el embrión en comparación con Krüppel. La proteína tailless inhibe la expresión del gen knirps en la parte posterior del embrión, lo que permite que la proteína knirps se exprese solo en la parte central del embrión (pero más posteriormente en comparación con Krüppel). Esto se debe a la capacidad tanto de hunchback como de tailless de unirse a las regiones potenciadoras de knirps.
Los genes gap codifican factores de transcripción que regulan la expresión de genes de reglas de pares y genes homeóticos [8] compitiendo por la unión a sus regiones potenciadoras. Se ha demostrado que la expresión de genes gap en el blastodermo de Drosophila exhibe una propiedad llamada canalización, una propiedad de los organismos en desarrollo para producir un fenotipo consistente a pesar de las variaciones en el genotipo o el entorno. Se ha propuesto que la canalización es una manifestación de la regulación cruzada de la expresión de genes gap y puede entenderse como algo que surge de las acciones de los atractores en el sistema dinámico de genes gap. [9]