El ensayo de metilación de Illumina que utiliza la plataforma Infinium I utiliza la tecnología 'BeadChip' [ aclaración necesaria ] para generar un perfil completo de todo el genoma de la metilación del ADN humano . Similar a la secuenciación de bisulfito y la pirosecuenciación , este método cuantifica los niveles de metilación en varios loci dentro del genoma . Este ensayo se utiliza para sondas de metilación en el Illumina Infinium HumanMethylation27 BeadChip (de aquí en adelante, matriz de 27k [metilación]). Las sondas en la matriz de 27k se dirigen a regiones del genoma humano para medir los niveles de metilación en 27.578 dinucleótidos CpG en 14.495 genes. [1] En 2008, Illumina lanzó la matriz Infinium HumanMethylation450 BeadChip ("matriz de 450 K"), que se dirige a más de 450.000 sitios de metilación. [2] En 2016, se lanzó el Infinium MethylationEPIC BeadChip ("EPIC"), que interroga más de 850.000 sitios de metilación en todo el genoma humano. [3]
La metilación del ADN desempeña un papel importante en la regulación epigenética de la estructura de la cromatina , que en la última década se ha reconocido como importante en la regulación de la expresión génica , el desarrollo y la impronta genética en vertebrados. [1] Se ha demostrado que los cambios en el patrón y el nivel de metilación contribuyen al cáncer y a varias enfermedades del desarrollo. [4] Por ejemplo, la hipermetilación en las islas CpG del promotor de un gen supresor de tumores , que a su vez conduce a su silenciamiento , se asocia frecuentemente con la tumorogénesis . [4] Una medición a gran escala de los patrones de metilación del ADN de una amplia selección de genes puede permitirnos comprender mejor las relaciones entre los cambios epigenéticos y la génesis de diferentes enfermedades y una mejor comprensión del papel que desempeña la epigenética en la diferenciación específica de tejidos .

El chip de metilación Illumina 27k contiene 27.578 sitios CpG individuales, distribuidos en 14.495 genes. [1] Estos genes incluyen genes RefSeq de la base de datos CCDS del NCBI, genes del cáncer que muestran patrones de metilación diferencial durante su curso de progresión y promotores de microARN. [5] Los marcadores incluidos en el chip se resumen en la Tabla 1. [5]
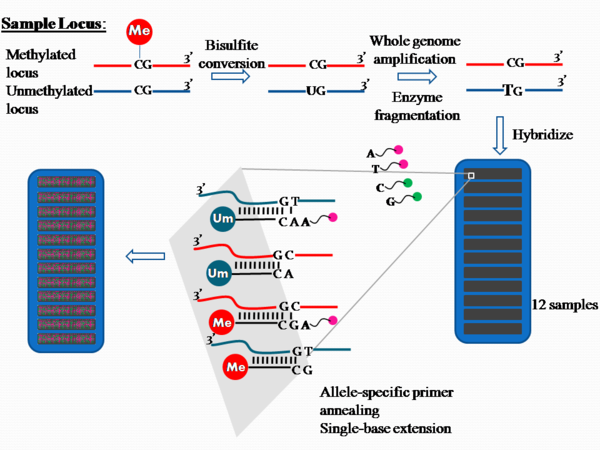
Para la tecnología de química del ensayo Infinium I, el proceso se describe en la Figura 1.
Tratamiento con bisulfito
Se utiliza aproximadamente 1 μg de ADN genómico en la conversión con bisulfito para convertir la citosina no metilada en uracilo . El producto contiene citosina no convertida donde previamente estaba metilada, pero citosina convertida en uracilo si previamente no estaba metilada.
Amplificación de ADN genómico completo
El ADN tratado con bisulfito se somete a una amplificación por desplazamiento múltiple de todo el genoma mediante cebado aleatorio de hexámeros y la ADN polimerasa Φ29 , que tiene una actividad de corrección de errores que da como resultado tasas de error 100 veces inferiores a las de la polimerasa Taq . A continuación, los productos se fragmentan enzimáticamente, [1] se purifican a partir de dNTP, cebadores y enzimas, y se aplican al chip. [6]
Hibridación y extensión de una sola base
En el chip, hay dos tipos de perlas para cada sitio CpG ( o "CG" , según la Figura 1) por locus. Cada locus probado se diferencia por diferentes tipos de perlas. [1] Ambos tipos de perlas están unidas a oligonucleótidos de ADN de 50 meros monocatenarios que difieren en secuencia solo en el extremo libre; este tipo de sonda se conoce como oligonucleótido específico de alelo . Uno de los tipos de perlas corresponderá al locus de citosina metilada y el otro corresponderá al locus de citosina no metilada, que se ha convertido en uracilo durante el tratamiento con bisulfito y luego se ha amplificado como timina durante la amplificación de todo el genoma. Los productos de ADN amplificados convertidos con bisulfito se desnaturalizan en cadenas simples y se hibridan al chip mediante hibridación específica de alelo con la sonda específica de metilación o la sonda de no metilación. La hibridación es seguida por una extensión de una sola base con didesoxinucleótidos marcados con hapteno . El ddCTP y el ddGTP están marcados con biotina, mientras que el ddATP y el ddUTP están marcados con 2,4-dinitrofenol (DNP). [7]
Tinción de fluorescencia y escaneo del chip Después de la incorporación de estos ddNTP marcados con hapteno, se realizan ensayos inmunohistoquímicos
multicapa mediante rondas repetidas de tinción con una combinación de anticuerpos para diferenciar los dos tipos. [7] Después de la tinción, se escanea el chip para mostrar las intensidades de los tipos de perlas metiladas y no metiladas. (Figura 2). [1] Los datos brutos se analizan mediante el software propietario y se calculan las proporciones de intensidad de fluorescencia entre los dos tipos de perlas. Para un individuo dado en un locus dado, un valor de proporción de 0 equivale a la no metilación del locus ( es decir , homocigoto no metilado); una proporción de 1 equivale a la metilación total ( es decir , homocigoto metilado); y un valor de 0,5 significa que una copia está metilada y la otra no ( es decir , heterocigosidad), en el genoma humano diploide. [1] [5]

Análisis de datos de metilación
Las imágenes de microarray escaneadas de datos de metilación son analizadas posteriormente por el sistema, que normaliza los datos brutos para reducir los efectos de la variación experimental, el fondo y la normalización promedio, y realiza pruebas estadísticas estándar sobre los resultados. [8] Los datos pueden luego compilarse en varios tipos de figuras para visualización y análisis. Los diagramas de dispersión se utilizan para correlacionar los datos de metilación; los diagramas de barras para visualizar los niveles relativos de metilación en cada sitio probado; los mapas de calor para agrupar los datos para comparar el perfil de metilación en los sitios probados. La Figura 2 muestra los diferentes tipos de resultados generados.
Ventajas
Desventajas