Cas9 ( proteína asociada a CRISPR 9 , anteriormente llamada Cas5 , Csn1 o Csx12 ) es una proteína de 160 kilodaltons que desempeña un papel vital en la defensa inmunológica de ciertas bacterias contra virus y plásmidos de ADN , y se utiliza ampliamente en aplicaciones de ingeniería genética . Su función principal es cortar el ADN y, por lo tanto, alterar el genoma de una célula. La técnica de edición genómica CRISPR-Cas9 contribuyó significativamente a que el Premio Nobel de Química de 2020 se otorgara a Emmanuelle Charpentier y Jennifer Doudna . [2]
Más técnicamente, Cas9 es una enzima endonucleasa de ADN guiada por ARN asociada con el sistema inmunológico adaptativo de repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas ( CRISPR ) en Streptococcus pyogenes . [3] [4] [5] S. pyogenes utiliza CRISPR para memorizar y Cas9 para luego interrogar y escindir ADN extraño, como ADN de bacteriófago invasor o ADN plasmídico. [4] [6] [7] [8] Cas9 realiza esta interrogación desenrollando el ADN extraño y verificando sitios complementarios a la región espaciadora de 20 nucleótidos del ARN guía (gRNA). Si el sustrato de ADN es complementario al ARN guía, Cas9 escinde el ADN invasor. En este sentido, el mecanismo CRISPR-Cas9 tiene una serie de paralelismos con el mecanismo de interferencia de ARN (RNAi) en eucariotas.
Aparte de su función original en la inmunidad bacteriana, la proteína Cas9 ha sido ampliamente utilizada como una herramienta de ingeniería genómica para inducir roturas de doble cadena dirigidas al sitio en el ADN. Estas roturas pueden conducir a la inactivación genética o la introducción de genes heterólogos a través de la unión de extremos no homólogos y la recombinación homóloga respectivamente en muchos organismos modelo de laboratorio. La investigación sobre el desarrollo de varias variantes de cas9 ha sido una forma prometedora de superar la limitación de la edición genómica CRISPR-Cas9 . Algunos ejemplos incluyen la nickasa Cas9 (Cas9n), una variante que induce roturas de cadena simple (SSB) o variantes que reconocen diferentes secuencias PAM . [9] Junto con las nucleasas de dedo de zinc y las proteínas nucleasas efectoras similares a activadores de la transcripción (TALEN), Cas9 se está convirtiendo en una herramienta destacada en el campo de la edición genómica.
Cas9 ha ganado fuerza en los últimos años porque puede escindir casi cualquier secuencia complementaria al ARN guía. [4] Debido a que la especificidad del objetivo de Cas9 proviene de la complementariedad del ARN guía:ADN y no de modificaciones a la proteína en sí (como TALENs y dedos de zinc ), la ingeniería de Cas9 para apuntar a nuevo ADN es sencilla. [10] Las versiones de Cas9 que se unen pero no escinden el ADN cognado se pueden utilizar para localizar activadores o represores transcripcionales en secuencias de ADN específicas para controlar la activación y represión transcripcional. [11] [12] Cas9 nativo requiere un ARN guía compuesto de dos ARN dispares que se asocian: el ARN CRISPR (crRNA) y el crRNA transactivador ( tracrRNA ). [3] La orientación de Cas9 se ha simplificado a través de la ingeniería de un ARN guía único quimérico (chiRNA). Los científicos han sugerido que los impulsores genéticos basados en Cas9 pueden ser capaces de editar los genomas de poblaciones enteras de organismos. [13] En 2015, se utilizó Cas9 para modificar el genoma de embriones humanos por primera vez. [14]
Para sobrevivir en una variedad de hábitats desafiantes e inhóspitos que están llenos de bacteriófagos , las bacterias y las arqueas han desarrollado métodos para evadir y defenderse de los virus depredadores . Esto incluye el sistema CRISPR de inmunidad adaptativa. En la práctica, los sistemas CRISPR/Cas actúan como enzimas de restricción autoprogramables. Los loci CRISPR están compuestos de repeticiones palindrómicas cortas que ocurren a intervalos regulares compuestos de repeticiones CRISPR alternas y espaciadores CRISPR variables de entre 24 y 48 nucleótidos de longitud. Estos loci CRISPR suelen estar acompañados por genes asociados a CRISPR (cas) adyacentes. En 2005, tres grupos separados descubrieron que las regiones espaciadoras eran homólogas a elementos de ADN extraños, incluidos plásmidos y virus. Estos informes proporcionaron la primera evidencia biológica de que CRISPR podría funcionar como un sistema inmunológico.
Cas9 se ha utilizado a menudo como una herramienta de edición del genoma. Cas9 se ha utilizado en desarrollos recientes para evitar que los virus manipulen el ADN de los huéspedes. Dado que CRISPR-Cas9 se desarrolló a partir de sistemas de genoma bacteriano, se puede utilizar para atacar el material genético de los virus. El uso de la enzima Cas9 puede ser una solución para muchas infecciones virales. Cas9 posee la capacidad de atacar virus específicos al apuntar a cadenas específicas de la información genética viral. Más específicamente, la enzima Cas9 se dirige a ciertas secciones del genoma viral que impiden que el virus lleve a cabo su función normal. [15] Cas9 también se ha utilizado para alterar la cadena perjudicial de ADN y ARN que causa enfermedades y cadenas mutadas de ADN. Cas9 ya ha demostrado ser prometedor en la interrupción de los efectos del VIH-1. Se ha demostrado que Cas9 suprime la expresión de las repeticiones terminales largas en el VIH-1. Cuando se introduce en el genoma del VIH-1, Cas9 ha demostrado la capacidad de mutar cadenas del VIH-1. [16] [17] Cas9 también se ha utilizado en el tratamiento de la hepatitis B mediante la orientación de los extremos de ciertas repeticiones terminales largas en el genoma del virus de la hepatitis B. [18] Cas9 se ha utilizado para reparar las mutaciones que causan cataratas en ratones.
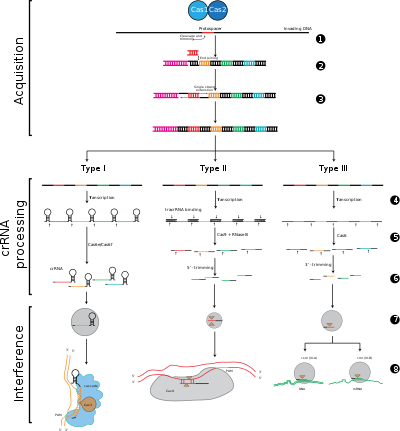
Los sistemas CRISPR-Cas se dividen en tres tipos principales (tipo I, tipo II y tipo III) y doce subtipos, que se basan en su contenido genético y diferencias estructurales. Sin embargo, las características definitorias fundamentales de todos los sistemas CRISPR-Cas son los genes cas y sus proteínas: cas1 y cas2 son universales en todos los tipos y subtipos, mientras que cas3 , cas9 y cas10 son genes característicos del tipo I, tipo II y tipo III, respectivamente.
La adaptación implica el reconocimiento y la integración de espaciadores entre dos repeticiones adyacentes en el locus CRISPR. El "protoespaciador" se refiere a la secuencia en el genoma viral que corresponde al espaciador. Existe un tramo corto de nucleótidos conservados proximal al protoespaciador, que se denomina motivo adyacente al protoespaciador (PAM). El PAM es un motivo de reconocimiento que se utiliza para adquirir el fragmento de ADN. [8] En el tipo II, Cas9 reconoce el PAM durante la adaptación para garantizar la adquisición de espaciadores funcionales. [6]
Aranaz et al. 2004 y Pourcel et al. 2007 también han observado pérdida de espaciadores e incluso grupos de varios. Esto probablemente ocurre a través de la recombinación homóloga del material entre repeticiones. [19]
La expresión de CRISPR incluye la transcripción de un transcrito primario llamado ARN CRISPR (pre-ARNcr), que se transcribe desde el locus CRISPR mediante la ARN polimerasa. Luego, endorribonucleasas específicas escinden los pre-ARNcr en pequeños ARN CRISPR (ARNcr). [20]
La interferencia involucra a los crRNA dentro de un complejo multiproteico llamado CASCADE, que puede reconocer y aparearse específicamente con regiones de inserción de ADN extraño complementario. Luego, el complejo crRNA-ácido nucleico extraño se escinde; sin embargo, si hay desajustes entre el espaciador y el ADN objetivo, o si hay mutaciones en el PAM, entonces no se iniciará la escisión. En este último escenario, el ADN extraño no es el objetivo del ataque de la célula, por lo que la replicación del virus continúa y el huésped no es inmune a la infección viral. La etapa de interferencia puede ser mecánica y temporalmente distinta de la adquisición y expresión de CRISPR, pero para que funcione completamente como un sistema de defensa, las tres fases deben ser funcionales. [21]
Etapa 1: Integración del espaciador CRISPR. Los protoespaciadores y los motivos asociados a ellos (mostrados en rojo) se adquieren en el extremo "líder" de una matriz CRISPR en el ADN del huésped. La matriz CRISPR está compuesta por secuencias espaciadoras (mostradas en recuadros de colores) flanqueadas por repeticiones (rombos negros). Este proceso requiere Cas1 y Cas2 (y Cas9 en el tipo II [6] ), que se codifican en el locus cas, que normalmente se encuentra cerca de la matriz CRISPR.
Etapa 2: Expresión CRISPR. El pre-ARNcr se transcribe comenzando en la región líder por la ARN polimerasa del huésped y luego las proteínas Cas lo escinden en ARNcr más pequeños que contienen un solo espaciador y una repetición parcial (que se muestra como una estructura en horquilla con espaciadores de colores).
Etapa 3: Interferencia de CRISPR. El ARNcr con un espaciador que tiene una fuerte complementariedad con el ADN extraño entrante comienza un evento de escisión (representado con tijeras), que requiere proteínas Cas. La escisión del ADN interfiere con la replicación viral y proporciona inmunidad al huésped. La etapa de interferencia puede ser funcional y temporalmente distinta de la adquisición y expresión de CRISPR (representada por una línea blanca que divide la célula).
dCas9 , también conocido como Cas9 deficiente en endonucleasas, se puede utilizar para editar la expresión génica cuando se aplica al sitio de unión de la transcripción de la sección deseada de un gen. La función óptima de dCas9 se atribuye a su modo de acción. La expresión génica se inhibe cuando ya no se añaden nucleótidos a la cadena de ARN y, por tanto, se termina la elongación de esa cadena, y como resultado afecta al proceso de transcripción. Este proceso se produce cuando dCas9 se produce en masa, por lo que puede afectar a la mayoría de los genes en un momento dado a través de una molécula de ARN guía específica de secuencia. Dado que dCas9 parece regular a la baja la expresión génica, esta acción se amplifica aún más cuando se utiliza junto con dominios modificadores de cromatina represivos. [22] La proteína dCas9 tiene otras funciones fuera de la regulación de la expresión génica. Se puede añadir un promotor a la proteína dCas9 que les permite trabajar entre sí para ser eficientes al iniciar o detener la transcripción en diferentes secuencias a lo largo de una cadena de ADN. Estas dos proteínas son específicas en cuanto a su acción sobre un gen. Esto es común en ciertos tipos de procariotas cuando un promotor y dCas9 se alinean para impedir la capacidad de elongación del polímero de nucleótidos que se unen para formar un fragmento de ADN transcrito. Sin el promotor, la proteína dCas9 no tiene el mismo efecto por sí sola o con un cuerpo génico. [23]
Al examinar más a fondo los efectos de la represión de la transcripción, H3K27, un componente aminoácido de una histona, se metila mediante la interacción de dCas9 y un péptido llamado FOG1. Básicamente, esta interacción provoca la represión génica en la sección terminal C + N del complejo de aminoácidos en la unión específica del gen y, como resultado, termina la transcripción. [24]
El dCas9 también demuestra ser eficaz a la hora de alterar ciertas proteínas que pueden provocar enfermedades. Cuando el dCas9 se une a una forma de ARN llamada ARN guía, impide la proliferación de codones repetidos y secuencias de ADN que podrían ser perjudiciales para el genoma de un organismo. Básicamente, cuando se producen múltiples codones repetidos, se produce una respuesta o se recluta una gran cantidad de dCas9 para combatir la sobreproducción de esos codones y se produce el cese de la transcripción. El dCas9 funciona sinérgicamente con el ARNg y afecta directamente a la ADN polimerasa II para que no continúe la transcripción.
Se puede encontrar una explicación más detallada de cómo funciona la proteína dCas9 en su utilización de genomas vegetales mediante la regulación de la producción de genes en las plantas para aumentar o disminuir ciertas características. El sistema CRISPR-CAS9 tiene la capacidad de regular al alza o a la baja los genes. Las proteínas dCas9 son un componente del sistema CRISPR-CAS9 y pueden reprimir ciertas áreas de un gen vegetal. Esto sucede cuando dCAS9 se une a dominios represores y, en el caso de las plantas, se produce la desactivación de un gen regulador como AtCSTF64. [25]
Las bacterias también son otro foco del uso de las proteínas dCas9. Dado que los eucariotas tienen una composición de ADN y un genoma más grandes, las bacterias, mucho más pequeñas, son fáciles de manipular. Como resultado, los eucariotas utilizan dCas9 para inhibir que la ARN polimerasa continúe el proceso de transcripción del material genético. [26]

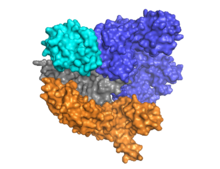
La Cas9 presenta una arquitectura bilobulada con el ARN guía ubicado entre el lóbulo alfa helicoidal (azul) y el lóbulo de la nucleasa (cian, naranja y gris). Estos dos lóbulos están conectados a través de una única hélice puente. Hay dos dominios de nucleasa ubicados en el lóbulo de nucleasa de múltiples dominios, el RuvC (gris) que corta la cadena de ADN no objetivo, y el dominio de nucleasa HNH (cian) que corta la cadena de ADN objetivo. El dominio RuvC está codificado por sitios secuencialmente dispares que interactúan en la estructura terciaria para formar el dominio de corte RuvC (ver figura de la derecha).
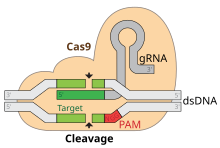
Una característica clave del ADN diana es que debe contener un motivo adyacente al protoespaciador (PAM) que consiste en la secuencia de tres nucleótidos: NGG. Este PAM es reconocido por el dominio de interacción con PAM (dominio PI, naranja) ubicado cerca del extremo C-terminal de Cas9. Cas9 sufre cambios conformacionales distintivos entre los estados apo, unido al ARN guía y unido al ARN guía:ADN.
Cas9 reconoce la arquitectura de tallo-bucle inherente al locus CRISPR, que media la maduración del complejo de ribonucleoproteína crRNA-tracrRNA. [28] Cas9 en complejo con ARN CRISPR (crRNA) y crRNA transactivador (tracrRNA) reconoce y degrada además el dsADN objetivo. [29] En la estructura de cocristal que se muestra aquí, el complejo crRNA-tracrRNA es reemplazado por un ARN guía único quimérico (sgRNA, en rojo) que ha demostrado tener la misma función que el complejo de ARN natural. [4] La base sgRNA emparejada con el ssADN objetivo está anclada por Cas9 como una arquitectura en forma de T. Esta estructura cristalina de la enzima Cas9 unida al ADN revela cambios conformacionales distintos en el lóbulo alfa-helicoidal con respecto al lóbulo de la nucleasa, así como la ubicación del dominio HNH. La proteína consta de un lóbulo de reconocimiento (REC) y un lóbulo de nucleasa (NUC). Todas las regiones, excepto el HNH, forman interacciones estrechas entre sí y con el complejo sgRNA-ssDNA, mientras que el dominio HNH forma pocos contactos con el resto de la proteína. En otra conformación del complejo Cas9 observada en el cristal, el dominio HNH no es visible. Estas estructuras sugieren la flexibilidad conformacional del dominio HNH.
Hasta la fecha, se han estudiado y publicado al menos tres estructuras cristalinas: una que representa una conformación de Cas9 en el estado apo [27] y dos que representan a Cas9 en el estado unido al ADN [30] [1]
En el complejo sgRNA-Cas9, según la estructura cristalina, los dominios REC1, BH y PI tienen contactos importantes con la cadena principal o bases tanto en la región repetida como en la espaciadora. [1] [30] Se han probado varios mutantes de Cas9 que incluyen la eliminación de los dominios REC1 o REC2 y mutaciones de residuos en BH. Los mutantes relacionados con REC1 y BH muestran una actividad menor o nula en comparación con el tipo salvaje, lo que indica que estos dos dominios son cruciales para el reconocimiento de sgRNA en la secuencia repetida y la estabilización de todo el complejo. Aunque las interacciones entre la secuencia espaciadora y Cas9, así como el dominio PI y la región repetida, necesitan más estudios, el cocristal demuestra una interfaz clara entre Cas9 y sgRNA.

Análisis de secuencias y estudios bioquímicos previos han postulado que Cas9 contiene dos dominios de nucleasa: un dominio de nucleasa HNH similar a McrA y un dominio de nucleasa similar a RuvC. [31] Estos dominios de nucleasa HNH y similar a RuvC son responsables de la escisión de las cadenas de ADN complementarias/diana y no complementarias/no diana, respectivamente. [4] A pesar de la baja similitud de secuencia, la secuencia similar a la ARNasa H tiene un pliegue RuvC (un miembro de la familia de la ARNasa H) y la región HNH se pliega como T4 Endo VII (un miembro de la familia de endonucleasas HNH). [ cita requerida ]
La Cas9 de S. pyogenes de tipo salvaje requiere cofactores de magnesio (Mg 2+ ) para la escisión del ADN mediada por ARN; sin embargo, se ha demostrado que Cas9 exhibe niveles variables de actividad en presencia de otros iones metálicos divalentes . [4] Por ejemplo, se ha demostrado que Cas9 en presencia de manganeso (Mn 2+ ) es capaz de escindir el ADN independiente del ARN. [32] La cinética de la escisión del ADN por Cas9 ha sido de gran interés para la comunidad científica, ya que estos datos proporcionan información sobre las complejidades de la reacción. Si bien se ha demostrado que la escisión del ADN por Cas9 unida al ARN es relativamente rápida ( k ≥ 700 s −1 ), la liberación de los productos de escisión es muy lenta ( t 1/2 = ln(2)/ k ≈ 43–91 h), lo que esencialmente convierte a Cas9 en una enzima de recambio único . [33] Estudios adicionales sobre la cinética de Cas9 han demostrado que la Cas9 modificada es eficaz para reducir los efectos no deseados modificando la velocidad de la reacción. [34] [35]
La eficiencia de escisión de Cas9 depende de numerosos factores. Un requisito clave es la presencia de un PAM válido en la cadena no objetivo 3 nucleótidos aguas abajo del sitio de escisión. [36] La secuencia PAM canónica para S. Pyogenes Cas9 es NGG, pero se toleran motivos alternativos con menor actividad de escisión. Los motivos PAM alternativos más eficientes para el S. Pyogenes Cas9 de tipo salvaje son NAG y NGA. [37] [38] La composición de la secuencia en el sitio de ADN objetivo complementario a la región espaciadora de 20 nucleótidos del gRNA también afecta la eficiencia de escisión. Las propiedades de composición de nucleótidos más relevantes que impactan la eficiencia son aquellas en la región proximal al PAM. [39] [40] [38] Los cambios de energía libre de los ácidos nucleicos también son muy relevantes para definir la actividad de escisión. [41] Los ARN guía que se unen al ADN formando un dúplex que cae dentro de un rango restringido de cambios de energía libre de unión que excluye uniones extremadamente débiles o estables generalmente funcionan de manera eficiente. [38] Las conformaciones de plegamiento estables del ARN guía también pueden perjudicar la escisión. [42]
La mayoría de las arqueas y bacterias se niegan obstinadamente a permitir que una Cas9 edite su genoma. Esto se debe a que pueden unir ADN extraño, que no las afecta, a su genoma. Otra forma en que estas células desafían a Cas9 es mediante el proceso del sistema de modificación por restricción (RM). Cuando un bacteriófago ingresa a una célula bacteriana o arquea, es atacado por el sistema RM. Luego, el sistema RM corta el ADN del bacteriófago en pedazos separados mediante enzimas de restricción y utiliza endonucleasas para destruir aún más las cadenas de ADN. Esto plantea un problema para la edición de Cas9 porque el sistema RM también ataca los genes extraños agregados por el proceso Cas9. [43]
Debido a la capacidad única de Cas9 de unirse a prácticamente cualquier secuencia del complemento en cualquier genoma , los investigadores querían utilizar esta enzima para reprimir la transcripción de varios loci genómicos . Para lograr esto, los dos residuos catalíticos cruciales del dominio RuvC y HNH se pueden mutar a alanina, eliminando toda la actividad endonucleasa de Cas9. La proteína resultante, denominada Cas9 "muerta" o "dCas9" para abreviar, todavía puede unirse firmemente al dsADN. Esta variante catalíticamente inactiva de Cas9 se ha utilizado tanto para estudios mecanísticos sobre la unión interrogativa de Cas9 al ADN como para un complejo ARN-proteína de unión al ADN programable general.
La interacción de dCas9 con el dsADN diana es tan estrecha que el desnaturalizante de proteína urea de alta molaridad no puede disociar completamente el complejo ARN-proteína dCas9 del dsADN diana. [44] dCas9 ha sido dirigido con ARN guía únicos diseñados a sitios de inicio de la transcripción de cualquier loci donde dCas9 puede competir con la ARN polimerasa en los promotores para detener la transcripción. [45] Además, dCas9 puede dirigirse a la región codificante de loci de modo que la inhibición de la ARN polimerasa ocurra durante la fase de elongación de la transcripción. [45] En eucariotas, el silenciamiento de la expresión génica puede extenderse dirigiendo dCas9 a secuencias potenciadoras, donde dCas9 puede bloquear el ensamblaje de factores de transcripción que conducen al silenciamiento de la expresión génica específica. [12] Además, los ARN guía proporcionados a dCas9 pueden diseñarse para incluir desajustes específicos con su secuencia cognada complementaria que debilitarán cuantitativamente la interacción de dCas9 con su secuencia cognada programada, lo que permite a un investigador ajustar el grado de silenciamiento génico aplicado a un gen de interés. [45] Esta tecnología es similar en principio al ARNi , de modo que la expresión génica se modula a nivel del ARN. Sin embargo, el enfoque dCas9 ha ganado mucha tracción ya que existen menos efectos fuera del objetivo y, en general, efectos de silenciamiento más grandes y reproducibles mediante el uso de dCas9 en comparación con los análisis de ARNi. [46] Además, debido a que el enfoque dCas9 para el silenciamiento génico se puede controlar cuantitativamente, un investigador ahora puede controlar con precisión el grado en el que se reprime un gen de interés, lo que permite responder a más preguntas sobre la regulación y la estequiometría génica .
Más allá de la unión directa de dCas9 a posiciones sensibles a la transcripción de loci, dCas9 se puede fusionar a una variedad de dominios de proteínas moduladoras para llevar a cabo una gran variedad de funciones. Recientemente, dCas9 se ha fusionado a proteínas de remodelación de cromatina (HDAC/HAT) para reorganizar la estructura de la cromatina alrededor de varios loci. [45] Esto es importante para apuntar a varios genes eucariotas de interés ya que las estructuras de heterocromatina obstaculizan la unión de Cas9. Además, debido a que Cas9 puede reaccionar a la heterocromatina , se teoriza que esta enzima se puede aplicar más al estudio de la estructura de la cromatina de varios loci. [45] Además, dCas9 se ha empleado en exámenes de represión génica de todo el genoma. Al emplear grandes bibliotecas de ARN guía capaces de apuntar a miles de genes, se han realizado exámenes genéticos de todo el genoma utilizando dCas9. [47]
Otro método para silenciar la transcripción con Cas9 es escindir directamente los productos de ARNm con la enzima Cas9 catalíticamente activa. [48] Este enfoque es posible mediante la hibridación de ssDNA con una secuencia de complemento PAM a ssRNA, lo que permite un sitio PAM dsDNA-RNA para la unión de Cas9. Esta tecnología permite aislar transcripciones de ARN endógeno en células sin la necesidad de inducir modificaciones químicas al ARN o métodos de etiquetado de ARN.
A diferencia de los genes silenciadores, dCas9 también se puede utilizar para activar genes cuando se fusiona con factores activadores de la transcripción. [45] Estos factores incluyen subunidades de la ARN polimerasa II bacteriana y factores de transcripción tradicionales en eucariotas. Recientemente, también se han logrado pruebas de activación de la transcripción en todo el genoma utilizando fusiones de dCas9 llamadas "CRISPRa" para la activación. [47]
{{cite book}}: Mantenimiento de CS1: falta la ubicación del editor ( enlace )