El alargamiento alternativo de los telómeros (también conocido como " ALT ") es un mecanismo independiente de la telomerasa mediante el cual las células cancerosas evitan la degradación de los telómeros .
En cada extremo de los cromosomas de la mayoría de las células eucariotas hay un telómero: una región de secuencias de nucleótidos repetitivas que protege el extremo del cromosoma del deterioro o de la fusión con los cromosomas vecinos. En cada división celular , los telómeros se acortan, lo que eventualmente impide una mayor división celular. Las células somáticas adultas sanas de los mamíferos no tienen enzimas telomerasa activas, por lo que las células cancerosas dejan de proliferar a menos que tengan una mutación que restablezca los telómeros. A menudo, esto se debe a la reactivación de una enzima telomerasa, pero también ocurren mecanismos alternativos.
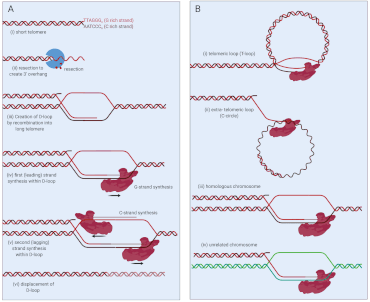
El principal mecanismo de alargamiento alternativo de los telómeros es un tipo de recombinación homóloga llamada síntesis de telómeros inducida por rotura (o BITS). [1] Normalmente, la recombinación homóloga permite que las cadenas de ADN rotas se reparen alineándolas con una secuencia coincidente de ADN no dañado, pero en BITS, este mecanismo se utiliza para extender los telómeros. Debido a que los telómeros son repetitivos por naturaleza, las secuencias coincidentes están ampliamente disponibles.
En los modelos propuestos sobre cómo funciona BITS, el proceso comienza con la resección del extremo de un telómero dañado: una de las hebras se corta para proporcionar una única hebra de ADN (la hebra rica en guanosina ) que puede unirse formando una unión (homóloga). ) plantilla, formando el llamado bucle de desplazamiento ( bucle D ) (Figura 1a). [2] En ALT, hay evidencia de que esta plantilla consta de: (i) una secuencia centrómero proximal del mismo cromosoma ( bucle T ), (ii) secuencias teloméricas extracromosómicas circulares (círculos C), (iii) cromosomas homólogos u (iv) otros cromosomas (Figura 1b). ALT puede surgir de una combinación de algunas o todas estas plantillas. [3] Es importante destacar que, debido a que los telómeros son altamente repetitivos, la invasión entre o dentro de los telómeros no está limitada por el requisito de una homología extendida en la recombinación homóloga. Después de la formación del bucle D, la ADN polimerasa δ extiende el extremo de la cadena G invadida, copiando material más allá del punto de ruptura original, lo que lleva al inicio de la síntesis de la cadena retrasada de la cadena C, también por la ADN polimerasa δ. [4]
La segunda característica de ALT es la producción de un producto de ADN no conservador en el telómero. Al finalizar la reacción de copia, ambas cadenas contienen ADN completamente nuevo. Esto es diferente de la replicación normal del ADN "semiconservadora" , donde una cadena se sintetiza recientemente y la otra proviene de la plantilla original. De esta manera, ALT permite copiar secuencias teloméricas completas de un cromosoma a otro, sin afectar la longitud o integridad de la secuencia copiada. Un trabajo reciente sugiere que la copia de ADN ALT (BITS) se produce a través de un modelo de migración de bucle D , que está respaldado por la observación de productos no conservativos en lugar de semiconservadores de replicación inducida por rotura en los telómeros ALT [5] y el D- Productos en forma de bucle observados en electroforesis en gel bidimensional en sitios sometidos a BIR. [6]