La sujeción del parche automatizada está comenzando [1] a reemplazar la sujeción del parche manual como método para medir la actividad eléctrica de las células individuales . Se utilizan diferentes técnicas para automatizar los registros de pinzamiento de parches de células en cultivo celular e in vivo . Este trabajo ha estado en curso desde finales de la década de 1990 por parte de laboratorios de investigación y empresas que intentan reducir la complejidad y el costo de la sujeción manual del parche. La fijación del parche durante mucho tiempo se consideró una forma de arte y todavía requiere mucho tiempo y es tediosa, especialmente in vivo. Las técnicas de automatización intentan reducir el error del usuario y la variabilidad en la obtención de registros electrofisiológicos de calidad a partir de células individuales.
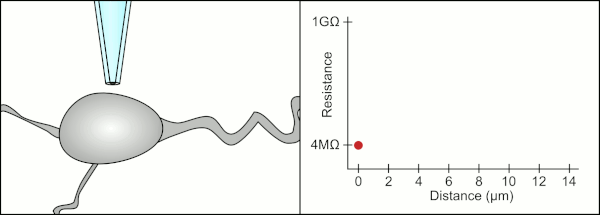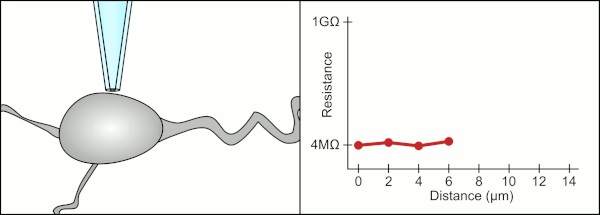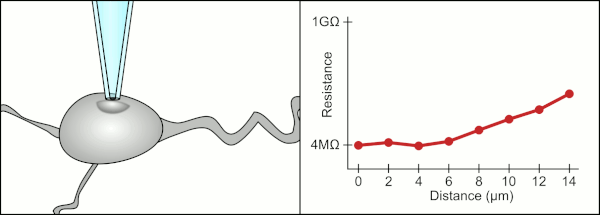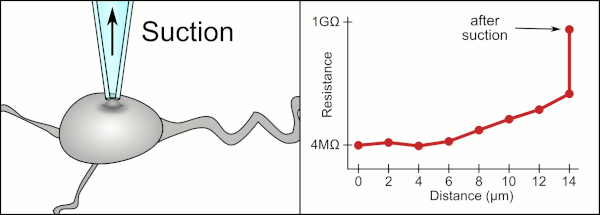
Erwin Neher y Bert Sakmann desarrollaron el método manual tradicional para parchar con pipetas de vidrio y requería un técnico altamente calificado. El técnico colocaría la pipeta de vidrio cerca de una celda y aplicaría la succión adecuada para crear un sello eléctrico entre la pipeta y la membrana celular. Este sello garantiza una grabación de calidad al evitar que se escape corriente entre la punta de la pipeta y la membrana celular. Este sello se realiza cuando la membrana de la célula se une químicamente con la punta de la pipeta de modo que el interior de la pipeta solo esté conectado al citoplasma de la célula. Esta conexión o junta entre membrana y vidrio se denomina "gigaseal". [2]
El técnico tradicionalmente usaba su boca para proporcionar las presiones precisas necesarias para sellarlo a la celda. Además de controlar la presión, el técnico también debe colocar la pipeta exactamente a la distancia correcta de la célula para que la membrana selle con ella. Usando un micromanipulador , la pipeta se mueve hacia la celda hasta que el técnico ve un cambio en la resistencia eléctrica entre el fluido dentro de la pipeta y el fluido circundante (ver animación). Por lo general, esto requiere de 3 a 12 meses de capacitación antes de que un técnico pueda registrar de manera confiable desde las células. Básicamente, el técnico realiza un acto de equilibrio tratando de observar y manipular varios sistemas simultáneamente (movimiento, presión y señales eléctricas). A menos que cada parte del proceso se realice con precisión y en el momento adecuado, el sello no se formará correctamente y el técnico tendrá que reemplazar la pipeta y comenzar de nuevo.
Estos desafíos reducen la cantidad de grabaciones que un técnico puede obtener y aumentan significativamente el costo. La automatización busca reducir el tiempo, la complejidad y el costo de la sujeción manual de parches. Mejorar el rendimiento también será clave para permitir patch-seq de alto rendimiento para combinar simultáneamente electrofisiología , morfología y propiedades transcriptómicas de las neuronas a una escala comparable a otros métodos de secuenciación [3].
La técnica de automatización varía dependiendo del entorno circundante de las células. Para las células in vivo, esto generalmente significa que las células están en el cerebro y rodeadas por otras células. Este entorno también contiene vasos sanguíneos , dendritas , axones y células gliales que dificultan la formación de un gigasello al obstruir la punta de la pipeta de 1 a 2 μm de diámetro. En este caso, el control preciso de la presión y la posición en la punta de la pipeta juega un papel importante a la hora de prevenir la obstrucción y detectar si una célula está cerca de la punta de la pipeta, como se analizó anteriormente.
Las células in vitro se pueden suspender en un líquido, hacer que se adhieran a una placa de cultivo o permanecer como parte de un trozo de tejido que se ha extraído del animal. Por lo general, estos entornos no tienen que compensar el movimiento del tejido debido a los latidos del corazón o la respiración de un animal. En el caso de las células en suspensión, la pipeta se sustituye completamente por un microchip con agujeros que permite crear gigasellos y medir la actividad eléctrica. La obstrucción también es menos problemática para las células o tejidos en placas de cultivo porque las células y la pipeta se pueden ver a través de un microscopio, lo que ayuda al técnico a evitar todo excepto la célula de interés.
Cada uno de estos sistemas automatizados debe realizar varias tareas. Debe colocar la celda al lado de la punta de una pipeta o algún otro dispositivo con un orificio de 1 a 2 μm, controlar la presión en el orificio y controlar el voltaje dentro de la celda.
Kodandaramaiah et al. demostraron un ejemplo de sujeción con parche in vivo. [4] En este caso el control de presión consistía en un conjunto de válvulas electrónicas y reguladores de presión electrónicos para proporcionar tres presiones que previamente eran proporcionadas por un técnico (alta presión 800-1000mbar, baja presión 20-30mbar y un pequeño vacío 15- 150 mbar). Tres válvulas electrónicas cambiaban entre las tres presiones y la presión atmosférica. La alta presión se usó para evitar la obstrucción de la pipeta, la baja presión se usó para buscar células y el vacío se usó para ayudar en el proceso de gigasellado. Todos estos estaban controlados por una computadora, para seleccionar entre las presiones a medida que cambiaba la resistencia en la punta de la pipeta.
En este caso, el control de posición manual fue reemplazado por un micromanipulador piezoeléctrico controlado por computadora que movía la pipeta en pasos discretos de 2 a 3 μm dentro del tejido hasta que hacía contacto con una célula. Este control de precisión es mucho más preciso y repetible que el posicionamiento manual y no requiere un operador.
La computadora también calcula y rastrea el cambio en la resistencia eléctrica cuando la pipeta hace contacto con la celda. Envía una señal de voltaje en forma de onda cuadrada por la pipeta que sale por el extremo de la pipeta o es bloqueada por la membrana celular. Cuando la membrana lo bloquea, la computadora detiene el movimiento de la pipeta y aplica succión para formar el gigaseal. Esta automatización elimina la toma de decisiones que tenía que realizar un técnico y, a diferencia de un técnico, la computadora puede realizar estas tareas de manera incansable y con mayor precisión.
Todos estos pasos se realizan en la misma secuencia lógica que la sujeción manual del parche, pero no requieren una gran capacitación para realizarlos y están completamente controlados por la computadora. Esto reduce el gasto necesario para obtener grabaciones de parche y aumenta la repetibilidad y solidez de la grabación en el cerebro vivo.


Se han desarrollado muchos tipos de sistemas para sujetar células en parches en cultivos en suspensión. Un sistema utiliza una pipeta tradicional y células en un cultivo en suspensión de gotitas para obtener registros con pinza de parche (ver figura). Esto tiene el beneficio adicional de utilizar sistemas tradicionales de fabricación de pipetas que calientan un capilar de vidrio y lo tiran a lo largo para crear la punta cónica que se utiliza en la sujeción del parche.
Los sistemas de automatización más comunes para cultivos en suspensión utilizan microchips con pequeños orificios (1-2 μm) en un sustrato plano en lugar de pipetas para crear el gigaseal y registrar a partir de células individuales. Los chips de parche se desarrollaron a principios de la década de 2000 como resultado de la mejora de las tecnologías de microfabricación desarrolladas por la industria de los semiconductores . Los chips suelen estar hechos de silicio , vidrio , PDMS y polimida . Los sistemas de chip de parche suelen ser más complejos y costosos, pero tienen el beneficio adicional de funcionar en paralelo y con manos libres. [5] [6] [7]
Normalmente, las neuronas no crecen en cultivos en suspensión, pero otros tipos de células sí pueden hacerlo. Algunos pueden transfectarse con genes para crear los canales iónicos de membrana de interés. Esto significa que una célula que normalmente no tiene actividad eléctrica puede desarrollar canales iónicos en su membrana que generarán corrientes iónicas. Debido a que las células están disociadas entre sí en cultivos en suspensión, las corrientes iónicas en una sola célula se pueden medir con precisión. Esto permite a los investigadores estudiar el comportamiento de los canales iónicos en entornos más controlados sin que interfieran las corrientes de otras células, como suele ocurrir en las redes neuronales. Esto es particularmente útil en estudios de detección de fármacos donde el objetivo es una proteína específica. [8] Debido a que manipular células en suspensión es mucho más fácil que manipular células en cultivo o in vivo, las grabaciones de patch-clamp se pueden obtener de esta manera mucho más rápido y de manera más confiable, lo que aumenta la productividad y hace posible la detección de miles de compuestos. [9]
Las neuronas derivadas de células madre cultivadas de forma adherente se pueden suspender y se han utilizado con éxito en dispositivos de abrazadera de parche plano. [10] Los canales iónicos, como los canales de sodio dependientes de voltaje , los canales de potasio dependientes de voltaje y los canales iónicos activados por ligando ionotrópico abiertos por el ligando GABA, se registraron a partir de estas células utilizando una pinza de parche manual y automatizada. [11]
Existen muchos métodos in vitro para el pinzamiento automatizado de células cultivadas o cortes de tejido cerebral.
Uno utiliza un chip de parche, como los mencionados anteriormente, junto con tratamientos de superficie que hacen que las células cultivadas migren a los orificios donde se forma el gigaseal a medida que crecen. [12] Al permitir que las neuronas crezcan en cultivo, forman redes espontáneamente, como las del cerebro, que se parecen más a los tejidos naturales que a las células aisladas en suspensión.
En otro método, las células se extraen de un animal y se cultivan en el chip de parche durante 2 a 4 horas mientras forman gigaseals espontáneamente con chips de parche de poliimida y PDMS [13]. Este sistema no requiere equipo externo para formar gigaseals.
Otra técnica automatiza el posicionamiento de células de sujeción del parche en cultivos. Utiliza una nanopipeta en un escenario preciso accionado piezoeléctricamente para escanear una superficie dentro de una placa de cultivo. Mientras escanea, mantiene una capacitancia eléctrica constante entre la punta de la pipeta y la superficie o las células debajo de ella moviéndola hacia arriba y hacia abajo. (A medida que se acerca a una celda, la capacitancia aumenta, por lo que el actuador aleja la pipeta y viceversa). Esto proporciona un mapeo topográfico preciso de la superficie dentro de la placa de cultivo. Una vez que se han mapeado las celdas, la computadora mueve la pipeta a una celda seleccionada y la baja para formar un gigasello con ella. [14]
Otra técnica simplemente automatiza la tarea de establecer contacto cuidadoso con las células. El operador coloca una pipeta sobre la muestra y luego deja que el software automatizado se haga cargo, bajando la pipeta y buscando detectar un aumento en la resistencia de la pipeta cuando hace contacto con una célula. En este punto finaliza el proceso y un técnico crea el gigaseal manualmente.
La instrumentación de automatización de abrazadera de parche estuvo disponible comercialmente en 2003. Debido al alto costo inicial, esta tecnología de casi 20 años estaba originalmente destinada a servir a las industrias biotecnológica y farmacéutica, pero durante los últimos años su presencia ha ido creciendo en entornos académicos y sin fines de lucro. dada su cada vez más probada confiabilidad técnica y relativa accesibilidad en costo. Un número creciente de universidades y otras instituciones académicas cuentan ahora con laboratorios e instalaciones centrales equipadas con aparatos automatizados de parcheo en conexión y coexistencia con otras tecnologías y métodos asociados o complementarios. [15] La aceptación y el reconocimiento de la electrofisiología automatizada con patch-clamp se refleja en el crecimiento exponencial de la literatura científica publicada con resultados obtenidos con esta nueva y revolucionaria tecnología [16]