PCR en tiempo real
A dicha mezcla se le adiciona una sustancia marcada con un fluoróforo que permita medir la tasa de generación de uno o más productos específicos.
Dicha medición se realiza tras cada ciclo de amplificación, y es por esto que se le denomina PCR en tiempo real (es decir, PCR inmediata, simultánea).
En muchos casos, el molde que se emplea para la PCR cuantitativa no es ADN desde el principio, sino que puede ser ADN complementario (ADNc), de hebra simple, obtenido por retrotranscripción de ácido ribonucleico (ARN); en este caso, la técnica es una RT-PCR cuantitativa o en tiempo real, o RT-Q-PCR.
Debe evitarse la confusión con la técnica denominada «PCR tras transcripción inversa» (RT-PCR, del inglés reverse transcriptase PCR), en la cual existe un paso de retrotranscripción de ARN a ADN pero que no necesariamente cuantifica el producto a tiempo real.
La PCR cuantitativa junto a los chip de ADN son modernas metodologías para el estudio de la expresión génica, aunque otros métodos tradicionales como el Northern blot, si bien no con tanta precisión, también permiten su abordaje.
[2] La variabilidad que se introduce en la cuantificación y que conlleva a errores en la estimación deriva de la integridad del ADN, la eficiencia enzimática y otros muchos factores, por lo que se han desarrollado numerosos sistemas de estandarización.
No obstante, según el criterio del investigador, posteriormente puede ser necesario correr en geles las muestras cuyo resultados previos en el PCR en tiempo real se pueden considerar dudosos o para ratificar resultados en muestras positivas.
En el segundo caso, se expresa el cambio en los niveles de expresión de ARN mensajero (ARNm) interpretado como ADN complementario (ADNc, generado por retrotranscripción del ARNm); esta cuantificación relativa es más fácil de realizar, puesto que no requiere curva de calibrado, y se sustenta en la comparación entre el nivel de expresión del gen a estudiar versus un gen control (también llamado de referencia, interno o normalizador o, en inglés, housekeeping gene).
No obstante, el aspecto crucial es que la estabilidad del gen de referencia sea una realidad.
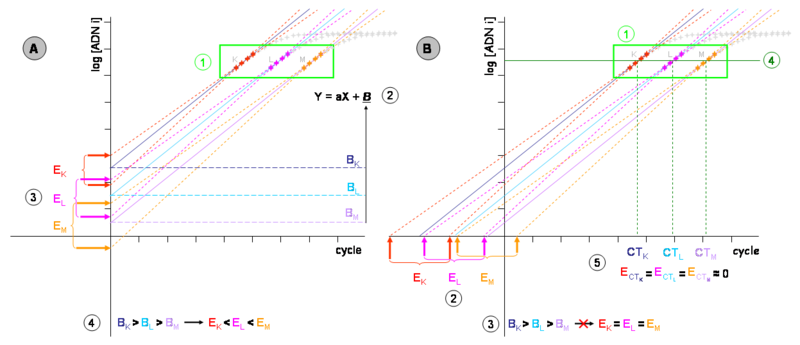
Este umbral conocido Ct (en inglés "cycle threshold") indica el ciclo de la PCR en la cual la fluorescencia sobrepasa un límite arbitrario durante la fase exponencial de la amplificación.
Como se indicaba en el apartado de cuantificación, el empleo del CT como valor matemático permite obtener resultados fiables, pero este hecho puede no ser explotado directamente.
Cada cinética permite determinar un CT para cada uno (es decir, un número en referencia a un ciclo en concreto).
Los rendimientos representados corresponden a «n» reacciones, cuya fiabilidad se debe a la existencia de réplicas independientes en la reacción, personal y reactivos.
La fluorescencia se muestra en unidades arbitrarias y el ruido de fondo ha sido limpiado.
Cabe destacar que la muestra Nn no ha amplificado en absoluto.
En el segundo caso, es de vital importancia seleccionar como gen estándar aquel cuya expresión realmente no varíe cuando se somete al individuo al tratamiento experimental cuyo efecto en la transcripción se desea estudiar (por ejemplo, un estrés ambiental,[17] biótico[18] o tipo de tejido[19]).
Con el fin de evaluar esta estabilidad, en algunos casos se comparan sus valores CT; en otros, sus cantidades relativas empleando la muestra cuyo CT es menor como elemento normalizador.
El cómputo de valor M se basa en definir la variación media, calculada comparando pares dos a dos, de un gen frente a todos los demás genes presentes en el estudio.
El algoritmo se realiza en varios pasos, ya que en cada ronda de comparaciones se suprime el gen que ha obtenido un peor valor; de este modo, es capaz de adjudicar un valor M a cada gen salvo para los más estables, que comparten el mismo.
En cuanto a valores PV, su cómputo se basa en el diseño de un factor de normalización óptimo; al principio, este se realiza con los dos genes más estables pero, secuencialmente, se le van añadiendo otros hasta que dicha adición no mejora significativamente la precisión de aquel.
De este modo, los genes con una variación inter e intragrupo mínima son los considerados más estables, con un valor de estabilidad S mínimo.
Se deben realizar varios tipos de controles: La PCR en tiempo real puede realizarse marcando de manera fluorescente oligonucleótidos que detectan específicamente la aparición del producto deseado.
EL FRET se fundamenta en que la excitación de un cromóforo puede transferirse a otro cercano, generalmente cuando ambos se sitúan en la misma molécula, mediante un mecanismo acoplador dipolo-dipolo.
[23] En el caso de que los cromóforos sean fluorescentes (esto es, fluorocromos), el mecanismo subyacente continúa siendo el mismo: la energía se transfiere, lo que desemboca en la aparición de fluorescencia (cabe destacar que no es la fluorescencia la transferida)[24][25] Las sondas empleadas en PCR a tiempo real existentes son: Si bien el uso más ampliamente difundido de la Q-PCR consiste en la evaluación de la expresión génica de genes concretos de forma relativa (empleando ARNm de la muestra y retrotranscribiéndolo a ADNc, que se mide mediante esta técnica), se han desarrollado otras aplicaciones fuera del entorno puramente académico.
[29] Existen variantes con sondas Scorpion, Molecular Beacon y LAMP para el mismo patógeno que, además, pueden ser aplicadas en campo.
Para ello se emplean cebadores específicos que amplifiquen no ya el propio transgén, sino su promotor, terminador e incluso sus secuencias intermedias, empleadas durante el proceso de ingeniería del vector.
[31][32] Las virosis en humanos pueden ser debidas a infecciones por parte de patógenos concretos o a coinfecciones, y este hecho no solo puede dificultar el diagnóstico mediante las técnicas clásicas, sino que puede redundar en un pronóstico diferente y en la necesidad de emplear una u otra quimioterapia.