Las tecnologías de recombinasa específica del sitio son herramientas de ingeniería genómica que dependen de las enzimas recombinasas para reemplazar secciones específicas de ADN.
A finales de los años 1980, la selección de genes en células madre embrionarias (CME) murinas permitió la transmisión de mutaciones a la línea germinal del ratón y surgió como una opción novedosa para estudiar la base genética de las redes reguladoras tal como existen en el genoma. Aun así, la selección de genes clásica resultó limitada de varias maneras, ya que las funciones de los genes se destruyeron irreversiblemente por el gen marcador que debía introducirse para seleccionar las CME recombinantes. Estos primeros pasos condujeron a animales en los que la mutación estaba presente en todas las células del cuerpo desde el principio, lo que dio lugar a fenotipos complejos y/o a una letalidad temprana. Había una clara necesidad de métodos para restringir estas mutaciones a puntos específicos del desarrollo y a tipos celulares específicos. Este sueño se hizo realidad cuando grupos de los EE. UU. pudieron introducir sistemas de recombinación específica de sitio (SSR) derivados de bacteriófagos y levaduras en células de mamíferos, así como en ratones. [1] [2] [3]
Las estrategias comunes de ingeniería genética requieren una modificación permanente del genoma objetivo. Para ello, es necesario invertir en un diseño muy sofisticado de las rutas que se utilizan para la administración de transgenes. Aunque para fines biotecnológicos la integración aleatoria sigue siendo común, puede dar lugar a una expresión genética impredecible debido a la variabilidad en el número de copias de los transgenes, la falta de control sobre los sitios de integración y las mutaciones asociadas. Los requisitos moleculares en el campo de las células madre son mucho más estrictos. En este campo, la recombinación homóloga (HR) puede, en principio, proporcionar especificidad al proceso de integración, pero en el caso de los eucariotas se ve comprometida por una eficiencia extremadamente baja. Aunque las meganucleasas, las nucleasas efectoras de tipo dedo de zinc y las nucleasas efectoras de tipo activador de la transcripción (ZFN y TALEN) son herramientas reales que respaldan la HR, fue la disponibilidad de recombinasas específicas de sitio (SSR) lo que desencadenó la construcción racional de líneas celulares con propiedades predecibles. Hoy en día, ambas tecnologías, HR y SSR, se pueden combinar en "tecnologías de etiquetado e intercambio" altamente eficientes. [4]
Se han identificado muchos sistemas de recombinación específicos de sitio para realizar estos reordenamientos de ADN con diversos fines, pero casi todos ellos pertenecen a una de dos familias, las recombinasas de tirosina (YR) y las recombinasas de serina (SR), según su mecanismo . Estas dos familias pueden mediar hasta tres tipos de reordenamientos de ADN (integración, escisión/resolución e inversión) a lo largo de diferentes rutas de reacción en función de su origen y arquitectura. [5]
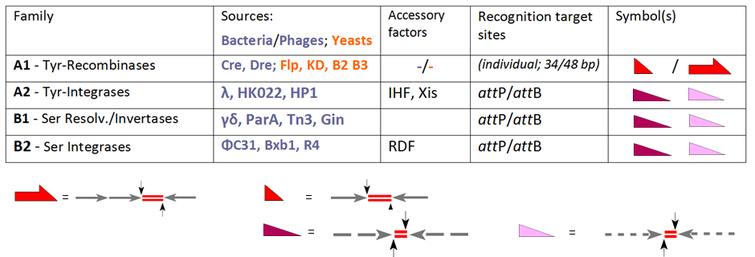
El miembro fundador de la familia YR es la integrasa lambda, codificada por el bacteriófago λ , que permite la integración del ADN del fago en el genoma bacteriano. Una característica común de esta clase es un nucleófilo de tirosina conservado que ataca el fosfato de ADN escindible para formar un enlace 3'-fosfotirosina. Los primeros miembros de la familia SR son las resolvasas/invertasas de ADN estrechamente relacionadas de los transposones bacterianos Tn3 y γδ, que dependen de una serina catalítica responsable de atacar el fosfato escindible para formar un enlace 5'-fosfoserina. Sin embargo, estos hechos indiscutibles se vieron comprometidos por una gran cantidad de confusión en el momento en que otros miembros entraron en escena, por ejemplo, las recombinasas YR Cre y Flp (capaces de integración, escisión/resolución así como inversión), que sin embargo fueron bienvenidas como nuevos miembros de la "familia de las integrasas". Los ejemplos inversos son PhiC31 y SR relacionados, que se introdujeron originalmente como resolvasas/invertasas aunque, en ausencia de factores auxiliares, la integración es su única función. Actualmente, la actividad estándar de cada enzima determina su clasificación, reservando el término general "recombinasa" para los miembros de la familia que, per se, comprenden las tres rutas, INT, RES e INV:
Nuestra tabla amplía la selección de los sistemas SSR convencionales y los agrupa según su rendimiento. Todas estas enzimas recombinan dos sitios diana, que son idénticos (subfamilia A1) o distintos (enzimas derivadas de fagos en A2, B1 y B2). [6] Mientras que para A1 estos sitios tienen designaciones individuales (" FRT " en el caso de la recombinasa Flp, loxP para la recombinasa Cre), los términos " att P" y " att B" (sitios de unión en la parte del fago y la parte bacteriana, respectivamente) son válidos en los otros casos. En el caso de la subfamilia A1 tenemos que lidiar con sitios cortos (normalmente de 34 pb) que consisten en dos brazos (casi) idénticos de 13 pb (flechas) que flanquean un espaciador de 8 pb (la región de cruce, indicada por dobletes de líneas rojas). [7] Nótese que para Flp hay un sitio alternativo de 48 pb disponible con tres brazos, cada uno de los cuales aloja una unidad Flp (un llamado "protómero"). Los sitios att P y att B siguen reglas arquitectónicas similares, pero aquí los brazos muestran solo una identidad parcial (indicada por las líneas discontinuas) y difieren en ambos casos. Estas características explican las diferencias relevantes:
Para simplificar este capítulo, las siguientes implementaciones se centrarán en dos recombinasas (Flp y Cre) y una sola integrasa (PhiC31), ya que su espectro cubre las herramientas que, en la actualidad, se utilizan principalmente para modificaciones dirigidas del genoma. Esto se hará en el marco de la siguiente descripción general.

El modo de integración/resolución e inversión (INT/RES e INV) dependen de la orientación de los sitios diana de la recombinasa (RTS), entre estos pares de att P y att B. La Sección C indica, de manera simplificada, la forma en que se puede alcanzar el intercambio de casetes mediado por recombinasa (RMCE) mediante cruces recíprocos dobles sincrónicos (en lugar de integración, seguida de resolución). [8] [9]
Las recombinasas Tyr son reversibles, mientras que la integrasa Ser es unidireccional. Cabe destacar la forma en que la integración/resolución de Flp reversible (una recombinasa Tyr) está modulada por versiones FRT de 48 pb (en lugar de los 34 pb mínimos) : el brazo adicional de 13 pb sirve como una "vía de aterrizaje" de Flp que contribuye a la formación del complejo sináptico, tanto en el contexto de las funciones Flp-INT como Flp-RMCE (ver las respectivas situaciones de equilibrio). Si bien es casi imposible evitar la reversión (impulsada por la entropía) de la integración en la sección A para Cre y es difícil de lograr para Flp, la RMCE se puede completar si el plásmido donante se proporciona en exceso debido al carácter bimolecular tanto de la reacción directa como de la inversa. Si se colocan ambos sitios FRT de manera inversa, se logrará un equilibrio de ambas orientaciones para el inserto (flecha verde). A diferencia de Flp, la integrasa Ser PhiC31 (representaciones inferiores) conduce a una integración unidireccional, al menos en ausencia de un factor de direccionalidad de la recombinasa (RDF). [10] En relación con Flp-RMCE, que requiere dos mutantes espaciadores FRT diferentes ("heteroespecíficos") , el socio de reacción ( att B) del primer sitio att P que reacciona se golpea arbitrariamente, de modo que no hay control sobre la dirección en la que el casete donante ingresa al objetivo (cf. los productos alternativos). También a diferencia de Flp-RMCE , varios objetivos RMCE distintos no se pueden montar en paralelo, debido a la falta de combinaciones att P/ att B heteroespecíficas (sin interacción cruzada) .
La recombinasa Cre (Cre) es capaz de recombinar secuencias específicas de ADN sin necesidad de cofactores . La enzima reconoce secuencias de ADN de 34 pares de bases llamadas loxP ("locus de cruce en el fago P1"). Dependiendo de la orientación de los sitios objetivo entre sí, Cre integrará/extirpará o invertirá secuencias de ADN. Tras la extirpación (llamada "resolución" en el caso de un sustrato circular) de una región particular de ADN, la expresión normal de genes se ve considerablemente comprometida o termina. [11]
Debido a la pronunciada actividad de resolución de Cre, una de sus aplicaciones iniciales fue la escisión de genes flanqueados por P de lox ("floxed") que conducen a la eliminación génica específica de la célula de dicho gen floxed después de que Cre se expresa en el tejido de interés. Las tecnologías actuales incorporan métodos que permiten el control espacial y temporal de la actividad de Cre. Un método común que facilita el control espacial de la alteración genética implica la selección de un promotor específico de tejido para impulsar la expresión de Cre. La colocación de Cre bajo el control de dicho promotor da como resultado una expresión localizada y específica de tejido. Como ejemplo, Leone et al. han colocado la unidad de transcripción bajo el control de las secuencias reguladoras del gen de la proteína proteolípida de mielina (PLP), lo que conduce a la eliminación inducida de secuencias génicas específicas en oligodendrocitos y células de Schwann . [12] El fragmento de ADN específico reconocido por Cre permanece intacto en las células, que no expresan el gen PLP; Esto a su vez facilita la observación empírica de los efectos localizados de las alteraciones del genoma en la vaina de mielina que rodea las fibras nerviosas en el sistema nervioso central (SNC) y el sistema nervioso periférico (SNP). [13] La expresión selectiva de Cre también se ha logrado en muchos otros tipos de células y tejidos.
Para controlar la actividad temporal de la reacción de escisión, se han desarrollado formas de Cre que aprovechan varios dominios de unión de ligando . Una estrategia exitosa para inducir la actividad temporal específica de Cre implica fusionar la enzima con un dominio de unión de ligando mutado para el receptor de estrógeno humano (ERt). Tras la introducción de tamoxifeno (un antagonista del receptor de estrógeno ), la construcción Cre-ERt puede penetrar el núcleo e inducir una mutación dirigida. ERt se une al tamoxifeno con mayor afinidad que los estrógenos endógenos , lo que permite que Cre-ERt permanezca citoplasmático en animales no tratados con tamoxifeno. El control temporal de la actividad de SSR por tamoxifeno permite que se induzcan cambios genéticos más tarde en la embriogénesis y/o en tejidos adultos. [12] Esto permite a los investigadores evitar la letalidad embrionaria mientras siguen investigando la función de los genes objetivo.
Las extensiones recientes de estos conceptos generales llevaron a generar el "Cre-zoo", es decir, colecciones de cientos de cepas de ratones para las cuales se pueden eliminar genes definidos mediante la expresión dirigida de Cre. [3]

En su hospedador natural (S. cerevisiae), el sistema Flp/ FRT permite la replicación de un "plásmido 2μ" mediante la inversión de un segmento flanqueado por dos sitios FRT idénticos, pero de orientación opuesta (actividad "flippasa"). Esta inversión cambia la orientación relativa de las horquillas de replicación dentro del plásmido, lo que permite el "círculo rodante": la amplificación de la entidad circular 2μ antes de que los intermediarios multiméricos se resuelvan para liberar múltiples productos monoméricos. Mientras que los sitios FRT mínimos de 34 pb favorecen la escisión/resolución en un grado similar a los sitios lox P análogos para Cre, las variantes naturales y más extendidas de FRT de 48 pb permiten un mayor grado de integración, al tiempo que superan ciertas interacciones promiscuas como las descritas para enzimas de fagos como Cre- [5] y PhiC31. [6] Una ventaja adicional es el hecho de que se pueden aplicar reglas simples para generar sitios FRT heteroespecíficos que experimentan cruces con socios iguales pero no con FRT de tipo salvaje . Estos hechos han permitido, desde 1994, el desarrollo y el perfeccionamiento continuo de las estrategias de intercambio de casetes mediadas por recombinasa (RMCE) que permiten el intercambio limpio de un casete objetivo por un casete donante entrante. [6]
Basándose en la tecnología RMCE, se ha desarrollado un recurso particular de cepas de ES precaracterizadas que se prestan a una mayor elaboración en el marco del programa EUCOMM (Mutagénesis Condicional Europea de Ratones), basado en las configuraciones de "FlExing" (escisión/inversión mediada por Flp) basadas en Cre y/o Flp, [6] que involucran las actividades de escisión e inversión. Iniciado en 2005, este proyecto se centró primero en la mutagénesis de saturación para permitir la anotación funcional completa del genoma del ratón (coordinado por el Consorcio Internacional de Ratones Knockout, IKMC) con el objetivo final de tener todos los genes de proteínas mutados a través de captura y selección de genes en células ES murinas. [14] Estos esfuerzos marcan la cima de varias estrategias de "etiquetado e intercambio", que se dedican a etiquetar un sitio genómico distinto de modo que la "etiqueta" pueda servir como una dirección para introducir información genética nueva (o alterar la existente). El paso de etiquetado en sí puede abordar ciertas clases de sitios de integración explotando las preferencias de integración de los retrovirus o incluso integrasas específicas del sitio como PhiC31, las cuales actúan de manera esencialmente unidireccional.
Los procedimientos tradicionales y laboriosos de "etiquetado e intercambio" se basaban en dos pasos sucesivos de recombinación homóloga (HR), el primero ("HR1") para introducir una etiqueta que consistía en un gen marcador de selección. "HR2" se utilizó luego para reemplazar el marcador por el "GOI". En la primera reacción ("knock-out"), el gen se etiquetó con un marcador seleccionable, típicamente mediante la inserción de un casete hygtk ([+/-]) que proporciona resistencia a G418. En el siguiente paso de "knock-in", la secuencia genómica etiquetada se reemplazó por secuencias genómicas homólogas con ciertas mutaciones. Los clones celulares se pudieron aislar entonces por su resistencia al ganciclovir debido a la pérdida del gen HSV-tk, es decir ("selección negativa"). Este procedimiento convencional de etiquetado e intercambio de dos pasos [15] se pudo agilizar después de la llegada de RMCE, que podría hacerse cargo y agregar eficiencia al paso de knock-in.
Sin lugar a dudas, las integrasas Ser son las herramientas de elección actuales para integrar transgenes en un número restringido de sitios aceptores genómicos bien conocidos que, en su mayoría (pero no siempre), imitan el sitio att P del fago, ya que atraen un vector donante que contiene att B. En este momento, el miembro más destacado es PhiC31-INT, con potencial demostrado en el contexto de los genomas humanos y de ratón.
A diferencia de las recombinasas Tyr mencionadas anteriormente, la PhiC31-INT actúa de manera unidireccional, bloqueando firmemente el vector donante en un objetivo anclado genómicamente. Una ventaja obvia de este sistema es que puede depender de sitios donantes att P (aceptores) y att B nativos no modificados. Pueden surgir beneficios adicionales (junto con ciertas complicaciones) del hecho de que los genomas de ratón y humano contienen per se un número limitado de objetivos endógenos (los llamados " pseudositios att P"). La información disponible sugiere que los considerables requisitos de secuencia de ADN permiten que la integrasa reconozca menos sitios que los sistemas de integración retrovirales o incluso basados en transposasas, lo que abre su carrera como un vehículo transportador superior para el transporte y la inserción en varios sitios genómicos bien establecidos, algunos de los cuales con las llamadas propiedades de "puerto seguro". [10]
Aprovechando el hecho de que existen rutas de recombinación específicas ( att P x att B), la RMCE se hace posible sin necesidad de sitios att sintéticos heteroespecíficos . Sin embargo, esta ventaja obvia se produce a expensas de ciertas deficiencias, como la falta de control sobre el tipo o la direccionalidad del casete entrante (donante). [6] Se imponen más restricciones por el hecho de que la irreversibilidad no permite configuraciones estándar de multiplexación-RMCE , incluidas las reacciones "RMCE en serie", es decir, intercambios repetidos de casetes en un locus genómico determinado .
La anotación de los genomas humanos y de ratones ha permitido identificar más de 20 000 genes codificadores de proteínas y más de 3000 genes de ARN no codificadores, que guían el desarrollo del organismo desde la fecundación hasta la vida adulta, pasando por la embriogénesis. Aunque se han observado avances espectaculares, la relevancia de las variantes genéticas raras sigue siendo un tema central de investigación.
Como una de las plataformas más importantes para abordar las funciones de los genes de vertebrados a gran escala, se han establecido recursos genéticos de todo el genoma de células madre embrionarias murinas mutantes. Con este fin, se han fundado cuatro programas internacionales destinados a la mutagénesis por saturación del genoma del ratón en Europa y América del Norte (EUCOMM, KOMP, NorCOMM y TIGM). Coordinados por el Consorcio Internacional de Ratones Knockout (IKSC), estos repositorios de células madre embrionarias están disponibles para el intercambio entre unidades de investigación internacionales. Los recursos actuales comprenden mutaciones en 11 539 genes únicos, 4 414 de ellos condicionales. [14]
Las tecnologías pertinentes han alcanzado ahora un nivel que permite su extensión a otras especies de mamíferos y a las células madre humanas, especialmente aquellas con estatus iPS (pluripotentes inducidas) .