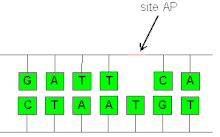
En bioquímica y genética molecular , un sitio AP ( sitio apurínico/apirimidínico ), también conocido como sitio abásico , es una ubicación en el ADN (también en el ARN , pero mucho menos probable) que no tiene una base púrica ni pirimidínica , ya sea de manera espontánea o debido a un daño en el ADN . Se ha estimado que, en condiciones fisiológicas, se pueden generar diariamente en una célula 10 000 sitios apurínicos y 500 apirimidínicos. [1] [2]
Los sitios AP pueden formarse por despurinación espontánea , pero también se producen como intermediarios en la reparación por escisión de bases . [3] En este proceso, una glicosilasa de ADN reconoce una base dañada y escinde el enlace N-glucosídico para liberar la base, dejando un sitio AP. Existe una variedad de glicosilasas que reconocen diferentes tipos de daño, incluidas las bases oxidadas o metiladas, o el uracilo en el ADN. El sitio AP puede luego ser escindido por una endonucleasa AP , dejando terminales 3'-hidroxilo y desoxirribosa-5-fosfato (ver Estructura del ADN ). De manera alternativa, las glicosilasas-liasas bifuncionales pueden escindir el sitio AP, dejando un fosfato 5' adyacente a un aldehído 3' α,β-insaturado. Ambos mecanismos forman una rotura de cadena sencilla, que luego se repara mediante una reparación por escisión de bases de parche corto o de parche largo. [4]
Si no se reparan, los sitios AP pueden provocar mutaciones durante la replicación semiconservativa . Pueden provocar el estancamiento de la horquilla de replicación y se evitan mediante la síntesis por translesión . En E. coli , la adenina se inserta preferentemente frente a los sitios AP, lo que se conoce como la "regla A". La situación es más compleja en eucariotas superiores, donde diferentes nucleótidos muestran una preferencia según el organismo y las condiciones experimentales. [3]
Los sitios AP se forman cuando la desoxirribosa se escinde de su base nitrogenada , rompiendo el enlace glucosídico entre las dos. Esto puede suceder espontáneamente, como resultado de la actividad química, la radiación o debido a la actividad enzimática. Los enlaces glucosídicos en el ADN se pueden romper mediante hidrólisis catalizada por ácido . Las bases de purina se pueden expulsar en condiciones débilmente ácidas, mientras que las pirimidinas requieren una acidez más fuerte para ser escindidas. Las purinas incluso se pueden eliminar a pH neutro , si la temperatura aumenta lo suficiente. La formación del sitio AP también puede ser causada por varios productos químicos modificadores de bases. La alquilación , la desaminación y la oxidación de bases individuales pueden conducir al debilitamiento del enlace glucosídico, por lo que la exposición a agentes que causan esas modificaciones puede fomentar la formación del sitio AP. [2]
La radiación ionizante también puede provocar la formación de sitios AP. Los entornos irradiados contienen radicales que pueden contribuir a la formación de sitios AP de múltiples maneras. Los radicales hidroxilo pueden atacar los enlaces glucosídicos, creando directamente un sitio AP, o hacer que el enlace glucosídico sea menos favorable al unirse a la base o al anillo desoxirribosa. [2]
Las enzimas, en concreto las glicosilasas de ADN, también suelen crear sitios AP, como parte de la vía de reparación por escisión de bases. Se estima que en una célula de mamífero determinada se forman entre 5000 y 10 000 sitios apurínicos al día. Los sitios apirimidínicos se forman a un ritmo aproximadamente 20 veces más lento, con estimaciones de alrededor de 500 eventos de formación al día, por célula. A ritmos tan elevados, es fundamental que las células cuenten con un aparato de reparación robusto para evitar mutaciones.
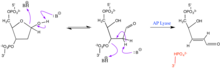
Los sitios AP son extremadamente reactivos. Fluctúan entre un anillo de furanosa y una conformación de aldehído libre y alcohol libre de cadena abierta . La exposición a un nucleófilo puede causar una reacción de β-eliminación, en la que se rompe el enlace fosfoéster 3', lo que provoca una ruptura de cadena simple. Esta reacción puede ser catalizada por la liasa AP . [2] En presencia de un exceso de reactivo, puede ocurrir una eliminación adicional en el lado 5'. El aldehído libre también puede reaccionar con aldehídos nucleófilos que contienen amina. Estas reacciones pueden promover aún más la escisión del enlace fosfoéster. Los aldehídos que contienen grupos O-HN2 pueden servir para estabilizar el sitio abásico al reaccionar con el grupo aldehído. Esta interacción no escinde el enlace fosfoéster.
Los sitios AP en células vivas pueden causar diversas y graves consecuencias, incluida la muerte celular. Las roturas de cadena sencilla que se producen debido a la eliminación β requieren reparación por la ADN Ligasa para evitar la mutación. Cuando la ADN polimerasa encuentra un sitio abásico, la replicación del ADN suele bloquearse, lo que puede conducir a una rotura de cadena sencilla o doble en la hélice del ADN. [4] En E. coli , cuando la enzima logra eludir el sitio abásico, una adenina se incorpora preferentemente a la nueva cadena. [2] [3] Si los sitios AP en el ADN no se reparan, la replicación del ADN no puede proceder normalmente y pueden producirse mutaciones significativas. [4] Si las mutaciones son simplemente polimorfismos de un solo nucleótido , entonces la célula puede no verse afectada. Sin embargo, si se producen mutaciones más graves, la función celular puede verse gravemente afectada, el crecimiento y la división pueden verse afectados, o la célula puede simplemente morir.
Los sitios AP son una característica importante de la vía de reparación por escisión de bases. Las glicosilasas de ADN primero crean sitios abásicos al reconocer y eliminar bases modificadas. Existen muchas variantes de glicosilasas para lidiar con las múltiples formas en que una base puede dañarse. Las circunstancias más comunes son la alquilación de bases, la oxidación y la presencia de un uracilo en la cadena de ADN. [4] Una vez que se ha creado con éxito un sitio AP, una endonucleasa AP cataliza la rotura de un enlace fosfoéster, creando una muesca en la estructura principal de la hélice. [4] La rotura puede ser a 3' o 5' del sitio, según la variante de la enzima. Luego, las enzimas de procesamiento final preparan el sitio para la ligadura de la muesca, que realiza la ADN polimerasa. [4] La base insertada en la muesca está determinada por la base correspondiente en la cadena opuesta. Luego, la ADN ligasa sella la muesca.
La actividad de la endonucleasa AP en la reparación de los sitios AP en la corteza frontal/parietal, el cerebelo , el tronco encefálico , el mesencéfalo y el hipotálamo disminuye con la edad en ratas con una dieta ad libitum . [5] En comparación, en ratas con restricción calórica , la actividad de la endonucleasa AP en estas regiones cerebrales sigue siendo mayor con la edad. [5] Estos hallazgos sugieren que la reparación elevada del sitio AP en animales con restricción calórica puede retrasar el proceso de envejecimiento.