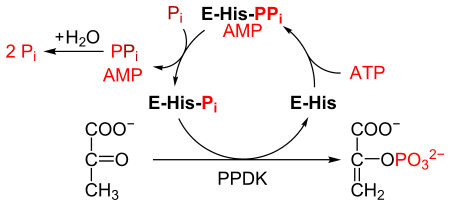
El piruvato, fosfato diquinasa o PPDK ( EC 2.7.9.1) es una enzima de la familia de las transferasas que cataliza la reacción química.
Esta enzima se ha estudiado principalmente en plantas, pero también en algunas bacterias. [1] Es una enzima clave en la gluconeogénesis y la fotosíntesis que es responsable de revertir la reacción realizada por la piruvato quinasa en la glucólisis de Embden-Meyerhof-Parnas. No debe confundirse con piruvato, agua diquinasa .
Pertenece a la familia de las transferasas , en concreto a las que transfieren grupos que contienen fósforo ( fosfotransferasas ) con aceptores pareados ( dicinasas ). Esta enzima participa en el metabolismo del piruvato y en la fijación de carbono .
El nombre sistemático de esta clase de enzimas es ATP:piruvato, fosfato fosfotransferasa. Otros nombres de uso común incluyen piruvato, ortofosfato dicinasa, piruvato-fosfato dicinasa (fosforilante), piruvato-fosfato dicinasa, piruvato-fosfato dicinasa, piruvato-fosfato dicinasa, piruvato-fosfato ligasa, piruvato-fosfato dicinasa, piruvato-fosfato ligasa, piruvato , Pi diquinasa y PPDK.
PPDK cataliza la conversión de piruvato en fosfoenolpiruvato (PEP), consumiendo 1 molécula de ATP y produciendo una molécula de AMP en el proceso. El mecanismo consta de 3 reacciones reversibles: [2]
La reacción es similar a la reacción catalizada por la piruvato quinasa , que también convierte el piruvato en PEP. [3] Sin embargo, la piruvato quinasa cataliza una reacción irreversible y no consume ATP. Por el contrario, PPDK cataliza una reacción reversible y consume 1 molécula de ATP por cada molécula de piruvato convertida.
Actualmente, se desconocen los detalles de cada paso mecanicista [3]
En su forma activa, PPDK es un homotetrámero con subunidades de aproximadamente 95 kDa [4]
Hay dos centros de reacción diferentes, separados por unos 45 angstroms , en los que se unen diferentes sustratos . [5] El sitio de unión de nucleótidos (ATP) está en el extremo N , tiene 240 aminoácidos y una característica captura de ATP. El sitio de unión de piruvato/PEP está en el extremo C , tiene 340 aminoácidos y un pliegue en barril α/β. También hay un dominio central, que contiene His 455, el residuo primario responsable de la catálisis. His455 es el residuo aceptor o donante de fosforilo. [3] La estructura de la enzima sugiere que el brazo His455 experimenta un movimiento giratorio para transportar un grupo fosforilo entre los dos centros de reacción. [6] Durante este giro, el dominio central gira al menos 92 grados y se traduce 0,5 Angstroms. [7]
Los estudios de las estructuras cristalinas de PPDK muestran que el dominio central se encuentra en diferente proximidad a los otros dos dominios dependiendo de la fuente de la enzima. [7] En el maíz , está más cerca del C-terminal, mientras que en Clostridium symbiosum , está más cerca del N-terminal.
La investigación ha demostrado que los mecanismos de unión de PPDK son similares a los de la D-Ala-D-Ala ligasa y la piruvato quinasa . [5] En particular, PPDK es muy similar a la piruvato quinasa, que también cataliza la conversión de piruvato en fosfoenolpiruvato; sin embargo, lo hace sin una enzima fosforilada intermedia. [3] Aunque sus secuencias de aminoácidos son diferentes, los residuos clave para la catálisis se conservan en ambas enzimas. Los experimentos de mutagénesis puntual han demostrado que los residuos catalíticos incluyen Arg 561, Arg 617, Glu 745, Asn 768 y Cys 831 (numeración relativa a la proteína C, simbiosa , PDB : 1KBL, 1KC7 ). [3]
PPDK se utiliza en la vía C4 , para mejorar la eficiencia de la fijación de dióxido de carbono . [8] En ambientes donde hay mucha luz, la tasa de fotosíntesis en las plantas está limitada por la tasa de absorción de dióxido de carbono (CO 2 ). Esto se puede mejorar mediante el uso de una serie de reacciones químicas para transportar CO 2 desde las células del mesófilo (que se encuentran en el exterior de una hoja) hasta las células de la vaina del haz (que se encuentran dentro de las células). PPDK convierte el piruvato en PEP, que reacciona con CO 2 para producir oxaloacetato . Cuando se libera CO 2 en las células de la vaina del haz, se regenera el piruvato y el ciclo continúa. [8]
Aunque la reacción catalizada por PPDK es reversible, la PEP se prefiere como producto en condiciones biológicas. Esto se debe al pH básico en el estroma , donde ocurre la reacción, así como a las altas concentraciones de adenilato quinasa y pirofosfatasa . Debido a que estas dos enzimas catalizan reacciones exergónicas que involucran AMP y difosfato, respectivamente, impulsan la reacción catalizada por PPDK. [9] Debido a que PPDK consume ATP, la vía C4 es desfavorable para las plantas en ambientes con poco acceso a la luz, ya que no pueden producir grandes cantidades de ATP. [8]
La PPDK es muy abundante en las hojas C4 y comprende hasta el 10% de la proteína total . [10] Las investigaciones han demostrado que la enzima es aproximadamente un 96% idéntica en diferentes especies de plantas. Los experimentos de hibridación revelaron que las diferencias genéticas se correlacionan con el grado en que las plantas realizan la vía C4; las secuencias poco comunes existen en plantas que también muestran características C3. [11] PPDK también se encuentra en pequeñas cantidades en plantas C3. La historia evolutiva sugiere que alguna vez tuvo un papel en la glucólisis como la piruvato quinasa similar , y finalmente evolucionó hacia la vía C4. [10]
Además de las plantas, PPDK también se encuentra en la ameoba parásita Entamoeba histolytica ( P37213 ) y en la bacteria Clostridium symbiosum ( P22983 ; así como en otras bacterias). [12] En esos dos organismos, la PPDK funciona de manera similar (y a veces en lugar de) la piruvato quinasa, catalizando la reacción en la dirección de producción de ATP como parte de la glucólisis. Se han propuesto inhibidores de Entamoeba PPDK como amebicidas contra este organismo. [13]

La PPDK vegetal está regulada por la proteína reguladora de piruvato y fosfato diquinasa (PDRP). [4] Cuando los niveles de luz son altos, PDRP desfosforila Thr 456 en PPDK usando AMP , activando así la enzima. [10] PDRP desactiva PPDK fosforilando el mismo residuo de treonina , usando difosfato . PDRP es un regulador único porque cataliza tanto la activación como la desactivación de PPDK, a través de dos mecanismos diferentes. [10]
La investigación sobre PPDK de maíz sugiere que los intrones , las secuencias terminadoras y quizás otras secuencias potenciadoras actúan de manera cooperativa para aumentar el nivel de ARNm funcional y estable . El ADNc de PPDK se expresó sólo ligeramente en el arroz transgénico , en comparación con el ADN intacto que experimentó una expresión significativa. [14]
A principios de 2018, se han resuelto 14 estructuras para esta clase de enzimas, con códigos de acceso PDB 1DIK, 1GGO, 1H6Z, 1JDE, 1KBL, 1KC7, 1VBG, 1VBH, 2DIK, 2FM4, 5JVJ, 5JVL, 5JVN, 5LU4.