Hepadnaviridae [a] es una familia de virus . [2] Los humanos, los simios y las aves sirven como huéspedes naturales. Actualmente hay 18 especies en esta familia, divididas entre 5 géneros. [3] Su miembro más conocido es el virus de la hepatitis B. Las enfermedades asociadas con esta familia incluyen: infecciones del hígado , como hepatitis, carcinomas hepatocelulares (infecciones crónicas) y cirrosis. [3] [4] Es la única familia aceptada en el orden Blubervirales .
Se reconocen los siguientes géneros: [ cita requerida ]
Aunque las enfermedades hepáticas transmisibles entre poblaciones humanas fueron identificadas temprano en la historia de la medicina, la primera hepatitis conocida con un agente etiológico viral fue la Hepatitis A, en la familia picornaviridae . El Virus de la Hepatitis B (VHB) fue identificado como una infección distinta de la Hepatitis A a través de su contaminación de la vacuna contra la fiebre amarilla . La vacuna contenía suero humano como agente estabilizador que estaba infectado con VHB. [5] El VHB fue identificado como un nuevo virus de ADN en la década de 1960, seguido un par de décadas después por el descubrimiento del flavivirus de la hepatitis C. El VHB fue identificado por primera vez en el laboratorio como el "agente de Australia" por Blumberg y colegas en la sangre de un paciente aborigen transfundido. Este trabajo le valió a Blumberg el Premio Nobel de Medicina de 1976. [ cita requerida ]
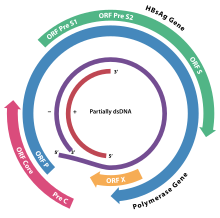
Los hepadnavirus tienen genomas muy pequeños de ADN circular parcialmente bicatenario y parcialmente monocatenario (pdsDNA). El genoma consta de dos hebras, una hebra más larga de sentido negativo y una hebra más corta de sentido positivo de longitud variable. En el virión, estas hebras están dispuestas de tal manera que los dos extremos de la hebra larga se encuentran pero no están unidos covalentemente. La hebra más corta se superpone a esta división y está conectada a la hebra más larga a cada lado de la división a través de un segmento de repetición directa (DR) que empareja las dos hebras. En la replicación, el pdsDNA viral se convierte en el núcleo de la célula huésped en ADN circular cerrado covalentemente (cccDNA) por la polimerasa viral. [ cita requerida ]
La replicación implica un intermediario de ARN , como en los virus pertenecientes al grupo VII de la clasificación de Baltimore . Se codifican cuatro marcos de lectura abiertos (ORF) principales y el virus tiene cuatro genes conocidos que codifican siete proteínas: la proteína de la cápside central, la polimerasa viral , antígenos de superficie (preS1, preS2 y S), la proteína X y HBeAg. Se cree que la proteína X no es estructural. Su función y significado son poco conocidos, pero se sospecha que está asociada con la modulación de la expresión génica del huésped. [ cita requerida ]
Los miembros de la familia Hepadnaviridae codifican su propia polimerasa, en lugar de cooptar la maquinaria del huésped como lo hacen otros virus. Esta enzima es única entre las polimerasas virales en el sentido de que tiene actividad de transcriptasa inversa para convertir el ARN en ADN para replicar el genoma (la única otra familia de virus patógenos para humanos que codifica una polimerasa con esta capacidad es Retroviridae ), actividad de ARNasa (utilizada cuando el genoma de ADN se sintetiza a partir de pgRNA que se empaquetó en viriones para la replicación para destruir la plantilla de ARN y producir el genoma pdsDNA) y actividad de ADN-polimerasa dependiente de ADN (utilizada para crear cccDNA a partir de pdsDNA en el primer paso del ciclo de replicación). [ cita requerida ]
Las proteínas de la envoltura de la hepatitis están compuestas de subunidades creadas a partir de los genes virales preS1, preS2 y S. La proteína de envoltura L (de "grande") contiene las tres subunidades. La proteína M (de "mediana") contiene solo preS2 y S. La proteína S (de "pequeña") contiene solo S. Las porciones del genoma que codifican estas subunidades de la proteína de envoltura comparten tanto el mismo marco como el mismo codón de terminación, lo que genera transcripciones anidadas en un único marco de lectura abierto. El pre-S1 se codifica primero (más cerca del extremo 5'), seguido directamente por el pre-S2 y el S. Cuando se realiza una transcripción desde el comienzo de la región pre-S1, los tres genes se incluyen en la transcripción y se produce la proteína L. Cuando la transcripción comienza después del pro-S1 al comienzo del pre-S2, la proteína final contiene solo las subunidades pre-S2 y S y, por lo tanto, es una proteína M. La proteína de envoltura más pequeña que contiene solo la subunidad S es la que más se produce porque se codifica más cerca del extremo 3' y proviene de la transcripción más corta. Estas proteínas de envoltura pueden ensamblarse independientemente de la cápside viral y del genoma en partículas no infecciosas similares a virus que le dan al virus una apariencia pleomórfica y promueven una fuerte respuesta inmune en los huéspedes. [ cita requerida ]
Los hepadnavirus se replican a través de un ARN intermediario (que luego transcriben nuevamente en ADNc mediante la transcriptasa inversa ). La transcriptasa inversa se une covalentemente a un cebador corto de 3 o 4 nucleótidos. [6] La mayoría de los hepadnavirus solo se replican en huéspedes específicos, lo que dificulta mucho los experimentos que utilizan métodos in vitro.
El virus se une a receptores específicos en las células y la partícula central entra en el citoplasma celular . Luego, se transloca al núcleo, donde la polimerasa viral "repara" el ADN parcialmente bicatenario para formar un genoma de dsADN circular completo (llamado ADN circular cerrado covalentemente o cccADN). Luego, el genoma se somete a transcripción por la ARN polimerasa de la célula huésped y el ARN pregenómico (pgARN) se envía fuera del núcleo. El pgARN se inserta en una cápside viral ensamblada que contiene la polimerasa viral. Dentro de esta cápside, el genoma se convierte de ARN a pdsADN mediante la actividad de la polimerasa como una ADN polimerasa dependiente de ARN y, posteriormente, como una ARNasa para eliminar la transcripción de pgARN. Estos nuevos viriones abandonan la célula para infectar a otras o se desmantelan inmediatamente para que los nuevos genomas virales puedan ingresar al núcleo y magnificar la infección. Los viriones que abandonan la célula salen a través de la gemación. [ cita requerida ]
Los virus de la familia Hepadnaviridae tienen una envoltura, geometría esférica y simetría T=4. El diámetro es de alrededor de 42 nm. Los genomas son circulares, de alrededor de 3,2 kb de longitud. El genoma codifica siete proteínas. [3] [4]
Basándose en la presencia de genomas virales en el ADN de las aves, parece que los hepadnavirus evolucionaron hace más de 82 millones de años . [7] Las aves pueden ser los huéspedes originales de los Hepadnaviridae y los mamíferos pueden infectarse después de un ave (véase cambio de huésped ).
Se han descrito genomas endógenos del virus de la hepatitis B en genomas de cocodrilos , serpientes y tortugas . [8] Esto sugiere que estos virus han infectado a los vertebrados hace más de 200 millones de años . [ cita requerida ]
También se han descrito hepadnavirus en peces y anfibios. [9] Esto sugiere que esta familia ha coevolucionado con los vertebrados. [ cita requerida ]
Los árboles filogenéticos sugieren que los virus de las aves se originaron a partir de los que infectan a los reptiles. Los que afectan a los mamíferos parecen estar más estrechamente relacionados con los que se encuentran en los peces. [10]
Se ha aislado de peces una familia propuesta de virus, los Nackednaviridae . Esta familia tiene una organización genómica similar a la de los miembros de la familia Hepadnaviridae . Estas dos familias se separaron hace más de 400 millones de años , lo que sugiere un origen antiguo para la familia Hepadnaviridae . [10]
Los virus de la familia tienen una estructura isosaédrica sin envoltura con simetría T=3, más pequeños que los viriones típicos de Hepadnaviridae (alrededor del 5% de estos últimos muestran una simetría T=3). El genoma circular y monopartito tiene alrededor de 3 kb, muy parecido al de Hepadnaviridae . Por lo tanto, la proteína de envoltura S no está presente, probablemente el estado ancestral según el análisis de secuencia. A diferencia de los virus Hepadnaviridae que generalmente divergen junto con sus hospedadores, los virus de la familia saltan de hospedador con mayor frecuencia. [10] El "tipo" de esta familia es el nackednavirus de los cíclidos africanos (ACNDV), anteriormente hepadnavirus de los cíclidos africanos (ACHBV), una especie propuesta y aún no aceptada. [9]
Los hepadnavirus, como su nombre "hepa" implica, infectan las células del hígado y causan hepatitis. Esto es cierto no sólo en el caso del virus de la hepatitis B, patógeno humano, sino también en el de los hepadnavirus que infectan a otros organismos. El paso de "adhesión" de la fase dinámica (en la que una proteína viral externa interactúa de manera estable con una proteína de la célula huésped) determina el tropismo celular. En el caso del VHB, el receptor del huésped es el receptor de taurocolato de sodio humano ( NTCP ), un mediador de la captación de ácidos biliares, y el antirreceptor del virus es la abundante proteína de envoltura HB-AgS. [11]