La esfingomielina fosfodiesterasa (EC 3.1.4.12, también conocida como esfingomielinasa neutra , esfingomielinasa o SMasa ; nombre sistemático esfingomielina colinafosfohidrolasa ) es una enzima hidrolasa que participa en las reacciones del metabolismo de los esfingolípidos . La SMasa es un miembro de la superfamilia de enzimas DNasa I y es responsable de descomponer la esfingomielina (SM) en fosfocolina y ceramida . La activación de la SMasa se ha sugerido como una ruta principal para la producción de ceramida en respuesta al estrés celular. [2]
Se han identificado cinco tipos de SMasa, que se clasifican según su dependencia de cationes y su pH óptimo de acción, y son:
De éstas, la SMasa ácida lisosomal y la SMasa neutra dependiente de magnesio se consideran candidatas principales para la producción de ceramida en la respuesta celular al estrés.
La actividad de la esfingomielinasa neutra (N-SMasa) se describió por primera vez en fibroblastos de pacientes con enfermedad de Niemann-Pick , una enfermedad de almacenamiento lisosomal caracterizada por deficiencias en la SMasa ácida. [3] Un estudio posterior descubrió que esta enzima era el producto de un gen distinto, tenía un pH óptimo de 7,4, dependía de iones Mg 2+ para su actividad y estaba particularmente enriquecida en el cerebro. [4] Sin embargo, un estudio más reciente en cerebro bovino sugirió la existencia de múltiples isoformas de N-SMasa con diferentes propiedades bioquímicas y cromatográficas. [5]
Un gran avance se produjo a mediados de la década de 1980 con la clonación de las primeras N-SMasas de Bacillus cereus y Staphylococcus aureus . [6] [7] El uso de las secuencias de estas esfingomielinasas bacterianas en búsquedas de homología condujo finalmente a la identificación de las N-SMasas de levadura ISC1 en la levadura en ciernes Saccharomyces cerevisiae [8] y las enzimas N-SMasas de mamíferos, nSMase1 y nSMase2. [9] [10] La identidad entre las SMasas de mamíferos, levaduras y bacterias es muy baja, siendo aproximadamente del 20% entre nSMase2 y la SMasa de B. cereus. Sin embargo, una alineación de las secuencias (ver figura) indica una serie de residuos conservados en toda la familia, particularmente en la región catalítica de las enzimas. [11] Esto ha llevado a la sugerencia de un mecanismo catalítico común para la familia N-SMase.
En 2006 se clonó y caracterizó una tercera proteína N-SMasa, denominada nSMase3 . [12] La secuencia de nSMase3 es muy similar a la de nSMase1 o nSMase2. Sin embargo, parece haber un alto grado de conservación evolutiva de organismos inferiores a superiores, lo que sugiere que puede comprender una N-SMasa única y distinta. La alta expresión de nSMase3 en el corazón y el músculo esquelético también sugiere posibles funciones en la función cardíaca. [13]

La resolución de la estructura cristalina de la esfingomielinasa neutra de Listeria ivanovii y Bacillus cereus ha permitido una comprensión más completa de su sitio enzimático. El sitio activo de la SMasa de B. cereus comprende los residuos Asn -16, Glu -53, Asp -195, Asn-197 y His -296. De estos, se sabe que los residuos Glu-53, Asp-195 y His-296 son esenciales para la actividad. Las actividades catalíticas relativas de la SMasa cuando los iones metálicos están unidos al sitio activo se han estudiado para los iones metálicos divalentes Co 2+ , Mn 2+ , Mg 2+ , Ca 2+ y Sr 2+ . De estos cinco iones metálicos, Co 2+ , Mn 2+ y Mg 2+ unidos al sitio activo dan como resultado una alta actividad catalítica de la SMasa. El Ca 2+ y el Sr 2+ unidos al sitio activo exhiben una actividad catalítica mucho menor de la SMasa. Cuando un ion Mg 2+ o dos iones Co 2+ se unen al sitio activo, resulta una geometría doblemente hexa- coordinada con dos bipirámides octaédricas para Co 2+ y una bipirámide octaédrica para Mg 2+ . Cuando un ion Ca 2+ se une al sitio activo, resulta una geometría hepta-coordinada. Por lo tanto, se predice que la diferencia en la actividad catalítica para los iones metálicos se debe a diferencias geométricas. De Co 2+ y Mg 2+ , la SMasa tiene mejor reactividad cuando dos iones Co 2+ están unidos a la SMasa; cuando estos iones Co 2+ están unidos, Glu-53 e His-296 se unen cada uno a un catión metálico divalente. Estos cationes están rodeados por moléculas de agua puenteadas y funcionan como ácidos de Lewis . [1]
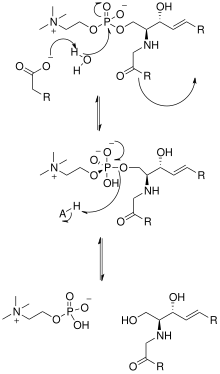
La resolución de la estructura cristalina de la esfingomielinasa neutra de Listeria ivanovii y Bacillus cereus también ha arrojado luz sobre sus mecanismos catalíticos. El sitio activo de SMase contiene residuos de Glu y His que están unidos a uno o dos cationes metálicos divalentes, normalmente Co 2+ , Mg 2+ o Ca 2+ para un rendimiento óptimo. Estos dos cationes ayudan en la catálisis reclutando SM al sitio activo de SMase. El catión divalente unido al residuo de Glu interactúa con el amido-oxígeno y el éster -oxígeno entre C1 y el grupo fosfato de SM; un residuo de Asn y el catión metálico divalente unido al residuo de His se unen a los átomos de oxígeno del grupo fosfato de SM. Esto estabiliza la carga negativa del grupo fosfato. El catión metálico unido al residuo de His y las cadenas laterales de Asp y Asn reducen el valor de pKa de una de las moléculas de agua puenteadas, activando así una molécula de agua. Esta molécula de agua actúa entonces como un nucleófilo y ataca al grupo fosfato de SM, creando un átomo de fósforo pentavalente cuya carga negativa es estabilizada por los cationes metálicos divalentes. El fosfato luego reforma su conformación tetraédrica y da como resultado los productos ceramida y fosfocolina . [1] En 2016 se propuso un modelo basado en la estructura cristalina del estudio de la esfingomielinasa ácida de mamíferos mediante el cual la ASMasa existe en equilibrio entre las formas abiertas y cerradas del dominio de saposina . En ausencia de membranas, la ASMasesap cerrada desacoplada de la ASMasecat predominaría y haría que la enzima fuera inactiva. En presencia de membranas aniónicas, la ASMasesap abierta se vuelve predominante, se acopla a la superficie de la membrana y forma concomitantemente una interfaz con el dominio catalítico activándolo para la hidrólisis de la esfingomielina . [14]