La inmunoprecipitación de ADN metilado (MeDIP o mDIP) es una técnica de purificación a gran escala ( a nivel de cromosomas o genoma ) en biología molecular que se utiliza para enriquecer secuencias de ADN metilado . Consiste en aislar fragmentos de ADN metilado a través de un anticuerpo generado contra la 5-metilcitosina (5mC). Esta técnica fue descrita por primera vez por Weber M. et al. [1] en 2005 y ha ayudado a allanar el camino para esfuerzos viables de evaluación a nivel de metiloma , ya que la fracción purificada de ADN metilado se puede introducir en métodos de detección de ADN de alto rendimiento como microarreglos de ADN de alta resolución ( MeDIP-chip ) o secuenciación de próxima generación (MeDIP-seq). No obstante, la comprensión del metiloma sigue siendo rudimentaria; su estudio se complica por el hecho de que, al igual que otras propiedades epigenéticas , los patrones varían de un tipo de célula a otro.
La metilación del ADN , que se refiere a la metilación reversible de la posición 5 de la citosina por las metiltransferasas , es una modificación epigenética importante en los organismos multicelulares. [2] En los mamíferos, esta modificación ocurre principalmente en los sitios CpG , que a su vez tienden a agruparse en regiones llamadas islas CpG . [3] Hay una pequeña fracción de islas CpG que pueden superponerse o estar muy cerca de las regiones promotoras de los sitios de inicio de la transcripción. La modificación también puede ocurrir en otros sitios, [4] pero la metilación en cualquiera de estos sitios puede reprimir la expresión génica al interferir con la unión de los factores de transcripción o modificar la estructura de la cromatina a un estado represivo. [5]
Los estudios sobre enfermedades han impulsado en gran medida el esfuerzo por comprender el papel de la metilación del ADN. Actualmente, el principal interés de investigación radica en investigar enfermedades como el cáncer para identificar regiones del ADN que han sufrido cambios extensos de metilación. Los genes contenidos en estas regiones son de interés funcional, ya que pueden ofrecer una explicación mecanicista de las causas genéticas subyacentes de una enfermedad. Por ejemplo, inicialmente se demostró que el patrón de metilación anormal de las células cancerosas [6] [7] [8] era un mecanismo a través del cual se silencian los genes similares a los supresores de tumores [9] , aunque más tarde se observó que afectaba a una gama mucho más amplia de tipos de genes [10] [11] [12]
Existen dos enfoques para el análisis de la metilación: tecnologías de tipificación y de elaboración de perfiles. Las tecnologías de tipificación están dirigidas a una pequeña cantidad de loci en muchas muestras e implican el uso de técnicas como PCR , enzimas de restricción y espectrometría de masas . Las tecnologías de elaboración de perfiles como MeDIP están dirigidas a una evaluación a nivel de genoma o metiloma de la metilación; esto incluye el escaneo genómico de puntos de referencia de restricción (RLGS), [13] y métodos basados en conversión de bisulfito , que se basan en el tratamiento del ADN con bisulfito para convertir los residuos de citosina no metilados en uracilo . [14] [15] [16] [17]
Otros métodos de mapeo y perfilado del metiloma han sido efectivos, pero no están exentos de limitaciones que pueden afectar la resolución, el nivel de rendimiento o las variaciones experimentales. Por ejemplo, RLGS está limitado por la cantidad de sitios de restricción en el genoma que pueden ser objetivos para la enzima de restricción; por lo general, se puede evaluar un máximo de ~4100 puntos de referencia. [18] Los métodos basados en secuenciación con bisulfito , a pesar de la posible resolución de un solo nucleótido, tienen un inconveniente: la conversión de citosina no metilada en uracilo puede ser inestable. [19] Además, cuando la conversión con bisulfito se combina con microarreglos de ADN para detectar sitios convertidos con bisulfito, la complejidad reducida de la secuencia de ADN es un problema. Los microarreglos capaces de perfilar de manera integral todo el genoma se vuelven difíciles de diseñar ya que hay menos sondas únicas disponibles. [20]
Las siguientes secciones describen el método de MeDIP combinado con hibridación de matriz de alta resolución o secuenciación de alto rendimiento. Cada método de detección de ADN también describirá brevemente el procesamiento y análisis posteriores al laboratorio. Se requiere un posprocesamiento diferente de los datos sin procesar según la tecnología utilizada para identificar las secuencias metiladas. Esto es análogo a los datos generados utilizando ChIP-chip y ChIP-seq .
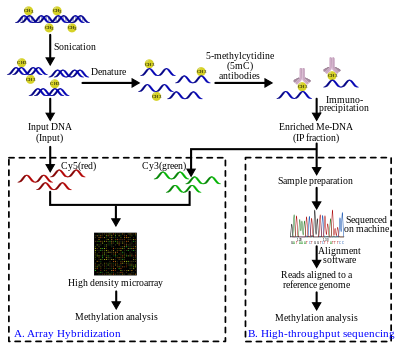
El ADN genómico se extrae ( extracción de ADN ) de las células y se purifica. Luego, el ADN purificado se somete a sonicación para cortarlo en fragmentos aleatorios. Este proceso de sonicación es rápido, simple y evita los sesgos de las enzimas de restricción . Los fragmentos resultantes varían de 300 a 1000 pares de bases (pb) de longitud, aunque normalmente tienen entre 400 y 600 pb. [21] La corta longitud de estos fragmentos es importante para obtener una resolución adecuada, mejorar la eficiencia del paso posterior en la inmunoprecipitación y reducir los efectos o sesgos de la longitud del fragmento. Además, el tamaño del fragmento afecta la unión del anticuerpo 5-metil-citidina (5mC) porque el anticuerpo necesita más de un solo 5mC para una unión eficiente. [22] Para mejorar aún más la afinidad de unión de los anticuerpos, los fragmentos de ADN se desnaturalizan para producir ADN monocatenario. Después de la desnaturalización, el ADN se incuba con anticuerpos monoclonales 5mC. A continuación se aplica la técnica clásica de inmunoprecipitación : se utilizan perlas magnéticas conjugadas con IgG antirratón para unir los anticuerpos anti-5mC y se elimina el ADN no unido en el sobrenadante. Para purificar el ADN, se añade proteinasa K para digerir los anticuerpos y liberar el ADN, que se puede recolectar y preparar para la detección de ADN.
Para más detalles sobre los pasos experimentales, véase [1] [19] [23] [24] .
Una fracción del ADN de entrada obtenido después del paso de sonicación anterior se marca con cianina -5 (Cy5; rojo) desoxi-citosina-trifosfato mientras que el ADN metilado, enriquecido después del paso de inmunoprecipitación, se marca con cianina -3 (Cy3; verde). Las muestras de ADN marcadas se cohibridan en una micromatriz genómica de alta densidad de 2 canales para sondear la presencia y las cantidades relativas. El propósito de esta comparación es identificar secuencias que muestran diferencias significativas en los niveles de hibridación, confirmando así que la secuencia de interés está enriquecida. La identificación basada en matrices de secuencias MeDIP se limita al diseño de la matriz. Como resultado, la resolución se restringe a las sondas en el diseño de la matriz. Hay pasos estándar adicionales necesarios en el procesamiento de señales para corregir problemas de hibridación como el ruido, como es el caso con la mayoría de las tecnologías de matrices.
Consulte [23] [24] [25] para más detalles.
El enfoque MeDIP-seq, es decir, el acoplamiento de MeDIP con tecnologías de secuenciación de lectura corta de próxima generación como la pirosecuenciación 454 o Illumina (Solexa), fue descrito por primera vez por Down et al. en 2008. [20] La secuenciación de alto rendimiento de los fragmentos de ADN metilados produce una gran cantidad de lecturas cortas (36-50 pb [26] o 400 pb, [27] dependiendo de la tecnología). Las lecturas cortas se alinean con un genoma de referencia utilizando un software de alineación como Mapping and Assembly with Quality (Maq), que utiliza un enfoque bayesiano , junto con cualidades de base y mapeo para modelar las probabilidades de error para las alineaciones. [28] Las lecturas luego se pueden extender para representar los fragmentos de ~400 a 700 pb del paso de sonicación. La cobertura de estas lecturas extendidas se puede utilizar para estimar el nivel de metilación de la región. También se puede utilizar un navegador de genoma como Ensembl para visualizar los datos.
La validación del método para evaluar la calidad y la precisión de los datos se puede realizar con PCR cuantitativa . Esto se hace comparando una secuencia de la muestra de MeDIP con una secuencia de control no metilada. Luego, las muestras se procesan en un gel y se comparan las intensidades de las bandas. [19] La intensidad relativa sirve como guía para encontrar el enriquecimiento. Los resultados también se pueden comparar con los resultados del chip MeDIP para ayudar a determinar la cobertura necesaria.

Las estimaciones del nivel de metilación del ADN pueden verse confundidas por las diferentes densidades de sitios CpG metilados en todo el genoma al observar los datos generados por MeDIP. Esto puede ser problemático para analizar regiones pobres en CpG (menor densidad). Una razón para este problema de densidad es su efecto sobre la eficiencia de la inmunoprecipitación. En su estudio, Down et al. [20] desarrollaron una herramienta para estimar los niveles absolutos de metilación a partir de los datos generados por MeDIP modelando la densidad de sitios CpG metilados. Esta herramienta se llama herramienta bayesiana para el análisis de metilación (Batman) . El estudio informa la cobertura de ~90% de todos los sitios CpG en promotores, regiones codificantes de genes, islas y elementos reguladores donde se pueden estimar los niveles de metilación; esto es casi 20 veces mejor cobertura que cualquier método anterior.
Los estudios que utilizan MeDIP-seq o MeDIP-chip son enfoques que abarcan todo el genoma y que tienen como objetivo común obtener el mapeo funcional del metiloma. Una vez que se identifican las regiones de metilación del ADN, se pueden aplicar varios análisis bioinformáticos para responder a ciertas preguntas biológicas. Un paso obvio es investigar los genes contenidos en estas regiones e investigar el significado funcional de su represión. Por ejemplo, el silenciamiento de los genes supresores de tumores en el cáncer se puede atribuir a la metilación del ADN. [29] Al identificar los eventos mutacionales que conducen a la hipermetilación y la posterior represión de los genes supresores de tumores conocidos, se pueden caracterizar de manera más específica los factores que contribuyen a la causa de la enfermedad. Alternativamente, se pueden identificar genes que se sabe que están metilados normalmente pero que, como resultado de algún evento de mutación, ya no están silenciados.
También se puede intentar investigar e identificar si algún regulador epigenético se ha visto afectado, como la ADN metiltransferasa (DNMT); [21] en estos casos, el enriquecimiento puede ser más limitado.
Se ha demostrado que el análisis de conjuntos de genes (por ejemplo, utilizando herramientas como DAVID y GoSeq) está severamente sesgado cuando se aplica a datos de metilación de alto rendimiento (por ejemplo, MeDIP-seq y MeDIP-ChIP); se ha sugerido que esto se puede corregir utilizando permutaciones de etiquetas de muestra o utilizando un modelo estadístico para controlar las diferencias en los números de sondas CpG / sitios CpG que se dirigen a cada gen. [30]
Las limitaciones que se deben tener en cuenta al utilizar MeDIP son factores experimentales típicos. Esto incluye la calidad y la reactividad cruzada de los anticuerpos 5mC utilizados en el procedimiento. Además, los métodos de detección de ADN (es decir, la hibridación en matriz y la secuenciación de alto rendimiento) suelen implicar limitaciones bien establecidas. En particular, para los procedimientos basados en matrices, como se mencionó anteriormente, las secuencias que se analizan están limitadas al diseño de matriz específico utilizado.
La mayoría de las limitaciones típicas de la secuenciación de alto rendimiento y de próxima generación se aplican. El problema de la precisión de la alineación con las regiones repetitivas en el genoma dará como resultado un análisis menos preciso de la metilación en esas regiones. Además, como se mencionó anteriormente, las lecturas cortas (por ejemplo, 36-50 pb de un analizador de genoma Illumina) representan una parte de un fragmento cortado cuando se alinean con el genoma; por lo tanto, el sitio de metilación exacto puede caer en cualquier lugar dentro de una ventana que es una función del tamaño del fragmento. [19] En este sentido, la secuenciación con bisulfito tiene una resolución mucho mayor (hasta un solo sitio CpG; nivel de nucleótido único). Sin embargo, este nivel de resolución puede no ser necesario para la mayoría de las aplicaciones, ya que se ha demostrado que el estado de metilación de los sitios CpG dentro de < 1000 pb está significativamente correlacionado. [20]