La hibridación in situ con fluorescencia ( FISH ) es una técnica citogenética molecular que utiliza sondas fluorescentes que se unen solo a partes particulares de una secuencia de ácido nucleico con un alto grado de complementariedad de secuencia . Fue desarrollada por investigadores biomédicos a principios de la década de 1980 [1] para detectar y localizar la presencia o ausencia de secuencias de ADN específicas en los cromosomas . La microscopía de fluorescencia se puede utilizar para averiguar dónde está unida la sonda fluorescente a los cromosomas. La FISH se utiliza a menudo para encontrar características específicas en el ADN para su uso en asesoramiento genético , medicina e identificación de especies. [2] La FISH también se puede utilizar para detectar y localizar objetivos de ARN específicos ( ARNm , lncRNA y miRNA ) [ cita requerida ] en células, células tumorales circulantes y muestras de tejido. En este contexto, puede ayudar a definir los patrones espacio-temporales de la expresión génica dentro de las células y los tejidos.

En biología, una sonda es una sola cadena de ADN o ARN que es complementaria a una secuencia de nucleótidos de interés.
Las sondas de ARN se pueden diseñar para cualquier gen o cualquier secuencia dentro de un gen para la visualización de ARNm , [3] [4] [5] lncRNA [6] [7] [8] y miRNA en tejidos y células. FISH se utiliza examinando el ciclo de reproducción celular, específicamente la interfase de los núcleos para detectar cualquier anomalía cromosómica. [9] FISH permite el análisis de una gran serie de casos de archivo mucho más fácil para identificar el cromosoma señalado mediante la creación de una sonda con una base cromosómica artificial que atraerá cromosomas similares. [9] Las señales de hibridación para cada sonda cuando se detecta una anomalía nucleica. [9] Cada sonda para la detección de ARNm y lncRNA se compone de ~20-50 pares de oligonucleótidos, cada par cubre un espacio de 40-50 pb. Los detalles dependen de la técnica FISH específica utilizada. Para la detección de miRNA, las sondas utilizan química patentada para la detección específica de miRNA y cubren toda la secuencia de miRNA.
Las sondas se derivan a menudo de fragmentos de ADN que se aislaron, purificaron y amplificaron para su uso en el Proyecto Genoma Humano . El tamaño del genoma humano es tan grande, en comparación con la longitud que se podría secuenciar directamente, que fue necesario dividir el genoma en fragmentos. (En el análisis final, estos fragmentos se ordenaron mediante la digestión de una copia de cada fragmento en fragmentos aún más pequeños utilizando endonucleasas específicas de secuencia, midiendo el tamaño de cada fragmento pequeño utilizando cromatografía de exclusión por tamaño y utilizando esa información para determinar dónde se superponían los fragmentos grandes). Para preservar los fragmentos con sus secuencias de ADN individuales, los fragmentos se agregaron a un sistema de poblaciones de bacterias que se replican continuamente. Las poblaciones clonales de bacterias, cada población que mantiene un solo cromosoma artificial, se almacenan en varios laboratorios en todo el mundo. Los cromosomas artificiales ( BAC ) se pueden cultivar, extraer y etiquetar en cualquier laboratorio que contenga una biblioteca. Las bibliotecas genómicas a menudo reciben el nombre de la institución en la que se desarrollaron. Un ejemplo es la biblioteca RPCI-11, que lleva el nombre del Roswell Park Comprehensive Cancer Center (antes conocido como Roswell Park Cancer Institute) en Buffalo, Nueva York . Estos fragmentos tienen alrededor de 100 mil pares de bases y son la base de la mayoría de las sondas FISH.
El propósito de utilizar RNA FISH es detectar transcripciones de ARNm objetivo en células, secciones de tejido o incluso preparaciones completas. [10] El proceso se realiza en tres procedimientos principales: preparación de tejido (prehibridación), hibridación y lavado (poshibridación).
La preparación del tejido comienza con la recolección de las secciones de tejido adecuadas para realizar la hibridación in situ con ARN. En primer lugar, se fijan las células, las células tumorales circulantes (CTC), las fijadas con formalina e incluidas en parafina (FFPE) o las secciones de tejido congeladas. Algunos fijadores de uso común son el formaldehído al 4 % o el paraformaldehído (PFA) en solución salina tamponada con fosfato (PBS). [10] La hibridación in situ con ARN también se ha realizado con éxito en células no fijadas. [11] Después de la fijación, las muestras se permeabilizan para permitir la penetración de los reactivos de hibridación. El uso de detergentes a una concentración del 0,1 % se utiliza comúnmente para mejorar la permeabilidad del tejido, como el Tween-20 o el Triton X-100. [12]
Para que el proceso de hibridación tenga un resultado in situ satisfactorio, es fundamental que se cumplan todas las condiciones óptimas, como la temperatura, el pH, la concentración de sal y el tiempo de la reacción de hibridación. Después de comprobar todas las condiciones necesarias, se pueden iniciar los pasos de hibridación añadiendo primero una sonda específica para el objetivo, compuesta por 20 pares de oligonucleótidos, que se hibrida con el ARN objetivo. Los sistemas de amplificación de señales independientes pero compatibles permiten el ensayo multiplex (hasta dos objetivos por ensayo). La amplificación de la señal se consigue mediante una serie de pasos de hibridación secuenciales. [13]
Después de los pasos de hibridación, se realizan los pasos de lavado. Estos pasos tienen como objetivo eliminar los híbridos no específicos y deshacerse de las moléculas de sonda no unidas de las muestras para reducir cualquier señalización de fondo. El uso de lavados con etanol se utiliza típicamente en esta etapa para reducir la autofluorescencia en tejidos o células. [14] Al final del ensayo, las muestras de tejido se visualizan bajo un microscopio de fluorescencia, como el microscopio de fluorescencia confocal y el microscopio Keyence. [12]
.jpg/440px-FISH_(Fluorescent_In_Situ_Hybridization).jpg)
En primer lugar, se construye una sonda. La sonda debe ser lo suficientemente grande como para hibridar específicamente con su diana, pero no tan grande como para impedir el proceso de hibridación. La sonda se marca directamente con fluoróforos , con dianas para anticuerpos o con biotina . El marcado se puede realizar de varias formas, como la traducción de mellas o la reacción en cadena de la polimerasa utilizando nucleótidos marcados .
A continuación, se produce una preparación cromosómica en interfase o metafase . Los cromosomas se adhieren firmemente a un sustrato , generalmente vidrio. Las secuencias repetitivas de ADN se deben bloquear añadiendo fragmentos cortos de ADN a la muestra. A continuación, se aplica la sonda al ADN cromosómico y se incuba durante aproximadamente 12 horas mientras se hibrida. Varios pasos de lavado eliminan todas las sondas no hibridadas o parcialmente hibridadas. A continuación, los resultados se visualizan y cuantifican utilizando un microscopio que sea capaz de excitar el colorante y registrar imágenes.
Si la señal fluorescente es débil, puede ser necesaria la amplificación de la señal para superar el umbral de detección del microscopio . La intensidad de la señal fluorescente depende de muchos factores, como la eficiencia de marcado de la sonda, el tipo de sonda y el tipo de colorante. Los anticuerpos marcados con fluorescencia o la estreptavidina se unen a la molécula del colorante. Estos componentes secundarios se seleccionan de modo que tengan una señal potente.
La hibridación in situ fluorescente es una técnica muy general. Las diferencias entre las distintas técnicas de hibridación in situ fluorescente suelen deberse a variaciones en la secuencia y el marcado de las sondas, y en cómo se utilizan en combinación. Las sondas se dividen en dos categorías genéricas: celulares y acelulares. En la hibridación in situ fluorescente se refiere a la colocación celular de la sonda.
El tamaño de la sonda es importante porque las sondas más cortas se hibridan de forma menos específica que las sondas más largas, de modo que a menudo se utilizan cadenas de ADN o ARN lo suficientemente largas (a menudo de 10 a 25 nucleótidos) que sean complementarias a una secuencia diana dada para localizar una diana. La superposición define la resolución de las características detectables. Por ejemplo, si el objetivo de un experimento es detectar el punto de ruptura de una translocación , entonces la superposición de las sondas (el grado en el que una secuencia de ADN está contenida en las sondas adyacentes) define la ventana mínima en la que se puede detectar el punto de ruptura.
La mezcla de secuencias de sonda determina el tipo de característica que la sonda puede detectar. Las sondas que se hibridan a lo largo de un cromosoma entero se utilizan para contar el número de un cromosoma determinado, mostrar translocaciones o identificar fragmentos extracromosómicos de cromatina . Esto a menudo se denomina "pintura de cromosoma completo". Si se utilizan todas las sondas posibles, cada cromosoma (el genoma completo) se marcaría con fluorescencia, lo que no sería particularmente útil para determinar características de secuencias individuales. Sin embargo, es posible crear una mezcla de sondas más pequeñas que sean específicas de una región particular (locus) de ADN; estas mezclas se utilizan para detectar mutaciones por deleción . Cuando se combinan con un color específico, se utiliza una mezcla de sondas específicas de locus para detectar translocaciones muy específicas. A menudo se utilizan mezclas de sondas especiales específicas de locus para contar cromosomas, uniéndose a las regiones centroméricas de los cromosomas, que son lo suficientemente distintivas para identificar cada cromosoma (con la excepción de los cromosomas 13 , 14 , 21 y 22 ).
Existen otras técnicas que utilizan mezclas de sondas de diferentes colores. Se puede detectar una gama de colores en mezclas de tintes fluorescentes, de modo que cada cromosoma humano se puede identificar por un color característico utilizando mezclas de sondas de cromosomas completos y una variedad de proporciones de colores. Aunque hay más cromosomas que colores de tintes fluorescentes fácilmente distinguibles, se pueden utilizar proporciones de mezclas de sondas para crear colores secundarios . De manera similar a la hibridación genómica comparativa , la mezcla de sondas para los colores secundarios se crea mezclando la proporción correcta de dos conjuntos de sondas de diferentes colores para el mismo cromosoma. Esta técnica a veces se denomina M-FISH.
La misma física que permite la variedad de colores en la técnica M-FISH se puede utilizar para la detección de translocaciones. Es decir, los colores adyacentes parecen superponerse; se observa un color secundario. Algunos ensayos están diseñados para que el color secundario esté presente o ausente en los casos de interés. Un ejemplo es la detección de translocaciones BCR/ABL , donde el color secundario indica enfermedad. Esta variación se denomina a menudo FISH de doble fusión o D-FISH. La situación opuesta (en la que la ausencia del color secundario es patológica) se ilustra con un ensayo utilizado para investigar translocaciones en las que solo se conoce o es constante uno de los puntos de ruptura. Se realizan sondas específicas de locus para un lado del punto de ruptura y el otro cromosoma intacto. En las células normales, se observa el color secundario, pero solo se observan los colores primarios cuando se produce la translocación. Esta técnica a veces se denomina "FISH de separación".
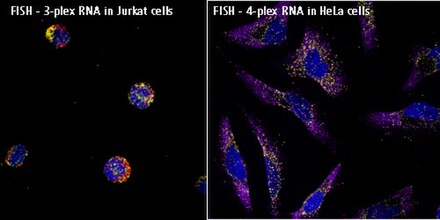
La hibridación in situ de ARN monomolécula, también conocida como hibridación in situ de ARN Stellaris® [15] o smFISH [16] , es un método para detectar y cuantificar ARNm y otras moléculas largas de ARN en una capa fina de muestra de tejido. Los objetivos se pueden visualizar de forma fiable mediante la aplicación de múltiples sondas de oligonucleótidos cortos marcados individualmente . [17] La unión de hasta 48 oligonucleótidos marcados con fluorescencia a una sola molécula de ARNm proporciona suficiente fluorescencia para detectar y localizar con precisión cada ARNm objetivo en una imagen de microscopía fluorescente de campo amplio . Las sondas que no se unen a la secuencia deseada no logran suficiente fluorescencia localizada para distinguirse del fondo . [18]
Los ensayos FISH de ARN de una sola molécula se pueden realizar en forma simple o múltiple , y se pueden utilizar como un experimento de seguimiento de la PCR cuantitativa , o se pueden obtener imágenes simultáneamente con un ensayo de anticuerpos fluorescentes . La tecnología tiene aplicaciones potenciales en el diagnóstico del cáncer , [19] la neurociencia , el análisis de la expresión genética , [20] y los diagnósticos complementarios .
En una técnica alternativa a las preparaciones en interfase o metafase, la hibridación in situ con fibra (FISH), los cromosomas en interfase se unen a un portaobjetos de tal manera que se estiran en línea recta, en lugar de estar fuertemente enrollados, como en la FISH convencional, o adoptar una conformación de territorio cromosómico , como en la FISH en interfase. Esto se logra aplicando un esfuerzo mecánico a lo largo del portaobjetos, ya sea a las células que se han fijado al portaobjetos y luego se han lisado , o a una solución de ADN purificado. Una técnica conocida como peinado cromosómico se utiliza cada vez más para este propósito. La conformación extendida de los cromosomas permite una resolución drásticamente mayor, incluso de hasta unas pocas kilobases . La preparación de muestras de FISH en fibra, aunque conceptualmente simple, es un arte bastante especializado, y solo los laboratorios especializados utilizan la técnica de forma rutinaria. [21]
La técnica Q-FISH combina la técnica FISH con PNA y software informático para cuantificar la intensidad de la fluorescencia. Esta técnica se utiliza de forma rutinaria en la investigación de la longitud de los telómeros .
Flow-FISH utiliza citometría de flujo para realizar FISH automáticamente utilizando mediciones de fluorescencia por célula.
La hibridación in situ asistida por microfluidos (MA-FISH) utiliza un flujo microfluídico para aumentar la eficiencia de la hibridación del ADN, lo que disminuye el costoso consumo de sondas FISH y reduce el tiempo de hibridación. La MA-FISH se aplica para detectar el gen HER2 en tejidos de cáncer de mama. [22]
La microautorradiografía FISH es una técnica para combinar sustratos radiomarcados con FISH convencional para detectar grupos filogenéticos y actividades metabólicas simultáneamente. [23]
La tecnología Hybrid Fusion FISH (HF-FISH) utiliza una combinación de fluoróforos de excitación/emisión aditiva primaria para generar espectros adicionales a través de un proceso de etiquetado conocido como transmisión óptica dinámica (DOT). Tres fluoróforos primarios pueden generar un total de 7 espectros de emisión fácilmente detectables como resultado del etiquetado combinatorio mediante DOT. La tecnología Hybrid Fusion FISH permite aplicaciones FISH altamente multiplexadas que están dirigidas a paneles de oncología clínica. La tecnología ofrece una puntuación más rápida con conjuntos de sondas eficientes que se pueden detectar fácilmente con microscopios fluorescentes tradicionales.
La hibridación in situ con fluorescencia resistente a errores multiplexada [24] es una versión altamente multiplexada de smFISH. Utiliza el etiquetado combinatorio, seguido de la obtención de imágenes y luego la codificación resistente a errores [25] para capturar una gran cantidad de moléculas de ARN y la localización espacial dentro de la célula. La captura de una gran cantidad de moléculas de ARN permite la elucidación de redes reguladoras de genes, la predicción de la función de genes no anotados y la identificación de patrones de distribución de moléculas de ARN, que se correlacionan con sus proteínas asociadas.
Starfish es un conjunto de herramientas de software desarrollado en 2019 por un consorcio de científicos para analizar datos de nueve variaciones diferentes de FISH, ya que todas las variaciones producen el mismo conjunto de datos: valores de expresión genética asignados a las coordenadas x e y en una célula. El software, creado para todos los científicos, no solo para los bioinformáticos, lee un conjunto de imágenes, elimina el ruido e identifica moléculas de ARN. Este enfoque se ha propuesto definir un esquema de análisis estándar de conjuntos de datos FISH de manera similar al análisis transcriptómico de células individuales . [26]
A menudo, los padres de niños con una discapacidad del desarrollo desean saber más sobre las condiciones de su hijo antes de elegir tener otro hijo. Estas preocupaciones se pueden abordar mediante el análisis del ADN de los padres y del niño. En los casos en que no se comprende la discapacidad del desarrollo del niño, la causa de la misma se puede determinar potencialmente utilizando FISH y técnicas citogenéticas . Los ejemplos de enfermedades que se diagnostican mediante FISH incluyen el síndrome de Prader-Willi , el síndrome de Angelman , el síndrome de deleción 22q13 , la leucemia mieloide crónica , la leucemia linfoblástica aguda , el cri-du-chat , el síndrome velocardiofacial y el síndrome de Down . La FISH en células espermáticas está indicada para hombres con un cariotipo somático o meiótico anormal, así como para aquellos con oligozoospermia , ya que aproximadamente el 50% de los hombres oligozoospérmicos tienen una mayor tasa de anomalías cromosómicas en los espermatozoides. [27] El análisis de los cromosomas 21, X e Y es suficiente para identificar a los individuos oligozoospérmicos en riesgo. [27]
En medicina, la hibridación in situ con fluorescencia se puede utilizar para formular un diagnóstico , evaluar el pronóstico o evaluar la remisión de una enfermedad, como el cáncer . De este modo, el tratamiento puede personalizarse específicamente. Un examen tradicional que implica el análisis de cromosomas en metafase a menudo no puede identificar características que distingan una enfermedad de otra, debido a características cromosómicas sutiles; la hibridación in situ con fluorescencia puede dilucidar estas diferencias. La hibridación in situ con fluorescencia también se puede utilizar para detectar células enfermas con mayor facilidad que los métodos citogenéticos estándar , que requieren la división de células y requieren una preparación y un análisis manual que requieren mucho tiempo y trabajo de un tecnólogo. La hibridación in situ con fluorescencia, por otro lado, no requiere células vivas y se puede cuantificar automáticamente; una computadora cuenta los puntos fluorescentes presentes. Sin embargo, se requiere un tecnólogo capacitado para distinguir las diferencias sutiles en los patrones de bandas de los cromosomas en metafase doblados y retorcidos. La hibridación in situ con fluorescencia se puede incorporar en un dispositivo microfluídico de laboratorio en un chip . Esta tecnología aún se encuentra en una etapa de desarrollo, pero, al igual que otros métodos de laboratorio en un chip, puede conducir a técnicas de diagnóstico más portátiles. [28] [29]

La hibridación in situ con fluorescencia (FISH) se ha estudiado ampliamente como técnica de diagnóstico para la identificación de patógenos en el campo de la microbiología médica. [30] Aunque se ha demostrado que es una técnica útil y aplicable, todavía no se aplica ampliamente en los laboratorios de diagnóstico. El corto tiempo para el diagnóstico (menos de 2 horas) ha sido una ventaja importante en comparación con la diferenciación bioquímica, pero esta ventaja se ve desafiada por la MALDI-TOF-MS, que permite la identificación de una gama más amplia de patógenos en comparación con las técnicas de diferenciación bioquímica. El uso de FISH con fines de diagnóstico ha encontrado su propósito cuando se necesita la identificación inmediata de especies, específicamente para la investigación de hemocultivos para los que la FISH es una técnica barata y fácil para el diagnóstico rápido preliminar. [30]
La hibridación in situ con hibridación fluorescente in situ (FISH) también se puede utilizar para comparar los genomas de dos especies biológicas y deducir relaciones evolutivas . Una técnica de hibridación similar se denomina zoo blot . Las sondas FISH bacterianas suelen ser cebadores para la región 16s del ARNr .
La hibridación in situ con fluorescencia (FISH) se utiliza ampliamente en el campo de la ecología microbiana para identificar microorganismos . Las biopelículas , por ejemplo, están compuestas por organizaciones bacterianas complejas (a menudo) de múltiples especies. La preparación de sondas de ADN para una especie y la realización de la hibridación in situ con esta sonda permite visualizar la distribución de esta especie específica dentro de la biopelícula. La preparación de sondas (en dos colores diferentes) para dos especies permite a los investigadores visualizar/estudiar la co-localización de estas dos especies en la biopelícula y puede ser útil para determinar la arquitectura fina de la biopelícula.
La hibridación genómica comparativa puede describirse como un método que utiliza FISH de manera paralela a la comparación de la fuerza de hibridación para recordar cualquier interrupción importante en el proceso de duplicación de las secuencias de ADN en el genoma del núcleo. [31]
El cariotipo virtual es otra alternativa clínicamente disponible y rentable a los paneles FISH, que utilizan miles o millones de sondas en una única matriz para detectar cambios en el número de copias, en todo el genoma, con una resolución sin precedentes. Actualmente, este tipo de análisis solo detecta ganancias y pérdidas de material cromosómico y no detecta reordenamientos equilibrados, como translocaciones e inversiones, que son aberraciones características que se observan en muchos tipos de leucemia y linfoma.
El cariotipo espectral es una imagen de cromosomas coloreados. El cariotipo espectral implica la hibridación in situ con múltiples formas de muchos tipos de sondas, con el resultado de ver cada cromosoma etiquetado a través de su etapa de metafase. Este tipo de cariotipo se utiliza específicamente cuando se buscan disposiciones cromosómicas.

La hibridación in situ con fluorescencia (FISH) se puede utilizar para estudiar la evolución de los cromosomas . Las especies relacionadas tienen cromosomas similares. Esta homología se puede detectar mediante la secuenciación de genes o genomas, pero también mediante la hibridación in situ con fluorescencia. Por ejemplo, los cromosomas humanos y de chimpancé son muy similares y la hibridación in situ con fluorescencia puede demostrar que dos cromosomas de chimpancé se fusionaron para dar lugar a un cromosoma humano. De manera similar, las especies que están más distantemente relacionadas tienen cromosomas similares, pero a medida que aumenta la distancia, los cromosomas tienden a romperse y fusionarse y, por lo tanto, dan lugar a cromosomas en mosaico. Esto se puede demostrar de forma impresionante mediante la hibridación in situ con fluorescencia (véase la figura). [32]