La espectroscopia de correlación de fluorescencia ( FCS ) es un análisis estadístico, a través de la correlación temporal, de las fluctuaciones estacionarias de la intensidad de la fluorescencia . Su fundamento teórico se originó a partir de la hipótesis de regresión de L. Onsager . El análisis proporciona parámetros cinéticos de los procesos físicos subyacentes a las fluctuaciones. Una de las aplicaciones interesantes de esto es un análisis de las fluctuaciones de concentración de partículas fluorescentes (moléculas) en solución. En esta aplicación, se observa la fluorescencia emitida desde un espacio muy pequeño en solución que contiene una pequeña cantidad de partículas fluorescentes (moléculas). La intensidad de la fluorescencia fluctúa debido al movimiento browniano de las partículas. En otras palabras, el número de partículas en el subespacio definido por el sistema óptico cambia aleatoriamente alrededor del número promedio. El análisis proporciona el número promedio de partículas fluorescentes y el tiempo de difusión promedio, cuando la partícula pasa a través del espacio. Finalmente, se determinan tanto la concentración como el tamaño de la partícula (molécula). Ambos parámetros son importantes en la investigación bioquímica, la biofísica y la química.
El FCS es una herramienta analítica muy sensible porque permite observar una pequeña cantidad de moléculas (concentraciones nanomolares a picomolares) en un volumen pequeño (~1 μm 3 ). [1] A diferencia de otros métodos (como el análisis HPLC ), el FCS no tiene un proceso de separación física; en cambio, logra su resolución espacial a través de su óptica. Además, el FCS permite la observación de moléculas marcadas con fluorescencia en la vía bioquímica en células vivas intactas. [2] Esto abre una nueva área, la "bioquímica in situ o in vivo": el seguimiento de la vía bioquímica en células y órganos intactos. [3]
Comúnmente, FCS se emplea en el contexto de la microscopía óptica , en particular la microscopía confocal o la microscopía de excitación de dos fotones . En estas técnicas, la luz se enfoca en una muestra y las fluctuaciones de intensidad de fluorescencia medidas (debido a la difusión , reacciones físicas o químicas, agregación, etc.) se analizan utilizando la autocorrelación temporal. Debido a que la propiedad medida está esencialmente relacionada con la magnitud y/o la cantidad de fluctuaciones, existe un régimen de medición óptimo en el nivel cuando las especies individuales entran o salen del volumen de observación (o se encienden y apagan en el volumen). Cuando se miden demasiadas entidades al mismo tiempo, las fluctuaciones generales son pequeñas en comparación con la señal total y pueden no ser resolubles; en la otra dirección, si los eventos de fluctuación individuales son demasiado dispersos en el tiempo, una medición puede tomar demasiado tiempo. FCS es en cierto modo la contraparte fluorescente de la dispersión de luz dinámica , que utiliza dispersión de luz coherente, en lugar de fluorescencia (incoherente).
Cuando se conoce un modelo apropiado, se puede utilizar FCS para obtener información cuantitativa como:
Debido a que los marcadores fluorescentes vienen en una variedad de colores y pueden unirse específicamente a una molécula particular (por ejemplo, proteínas, polímeros, complejos metálicos, etc.), es posible estudiar el comportamiento de moléculas individuales (en rápida sucesión en soluciones compuestas). Con el desarrollo de detectores sensibles como los fotodiodos de avalancha, la detección de la señal de fluorescencia proveniente de moléculas individuales en muestras altamente diluidas se ha vuelto práctica. Con esto surgió la posibilidad de realizar experimentos de FCS en una amplia variedad de muestras, que van desde la ciencia de los materiales hasta la biología. El advenimiento de células diseñadas con proteínas etiquetadas genéticamente (como la proteína fluorescente verde ) ha hecho que el FCS sea una herramienta común para estudiar la dinámica molecular en células vivas. [4]
Las técnicas de correlación de señales se aplicaron por primera vez de forma experimental a la fluorescencia en 1972 por Magde, Elson y Webb [5] , a quienes se les suele reconocer como los inventores del FCS. La técnica se desarrolló aún más en un grupo de artículos de estos y otros autores poco después, estableciendo las bases teóricas y los tipos de aplicaciones. [6] [7] [8] Alrededor de 1990, con la capacidad de detectar un número suficientemente pequeño de partículas de fluorescencia, surgieron dos problemas: una distribución no gaussiana de la intensidad de la fluorescencia y el volumen de medición confocal tridimensional de un sistema de microscopía láser. [9] El primero condujo a un análisis de distribuciones y momentos de las señales fluorescentes para extraer información molecular, [10] [11] que finalmente se convirtió en una colección de métodos conocidos como análisis de brillo. Véase Thompson (1991) [12] para una revisión de ese período.
A partir de 1993, [13] una serie de mejoras en las técnicas de medición (especialmente mediante el uso de microscopía confocal y luego microscopía de dos fotones) para definir mejor el volumen de medición y rechazar el fondo, mejoraron en gran medida la relación señal-ruido y permitieron la sensibilidad de una sola molécula. [14] [15] Desde entonces, ha habido un renovado interés en FCS y, a partir de agosto de 2007, se han encontrado más de 3000 artículos que utilizan FCS en Web of Science. Consulte Krichevsky y Bonnet [16] para una revisión. Además, ha habido una oleada de actividad para extender FCS de varias maneras, por ejemplo, al escaneo láser y la microscopía confocal de disco giratorio (a partir de una medición estacionaria de un solo punto), en el uso de correlación cruzada (FCCS) entre dos canales fluorescentes en lugar de autocorrelación, y en el uso de transferencia de energía de resonancia de Förster (FRET) en lugar de fluorescencia.

La configuración típica de FCS consiste en una línea láser (longitudes de onda que varían típicamente de 405 a 633 nm ( cw ) y de 690 a 1100 nm (pulsada)), que se refleja en un objetivo de microscopio mediante un espejo dicroico. El haz láser se enfoca en la muestra, que contiene partículas fluorescentes (moléculas) en una dilución tan alta que solo unas pocas están dentro del punto focal (generalmente 1 a 100 moléculas en un fL). Cuando las partículas cruzan el volumen focal, fluorescen. Esta luz es recolectada por el mismo objetivo y, debido a que está desplazada al rojo con respecto a la luz de excitación, pasa el espejo dicroico y llega a un detector, típicamente un tubo fotomultiplicador , un detector de fotodiodo de avalancha o un detector de fotón único de nanocables superconductores . La señal electrónica resultante se puede almacenar directamente como un trazo de intensidad versus tiempo para analizarlo en un momento posterior, o calcularse para generar la autocorrelación directamente (lo que requiere tarjetas de adquisición especiales). La curva FCS por sí sola sólo representa un espectro temporal. De ahí se deben extraer conclusiones sobre los fenómenos físicos con modelos adecuados. Los parámetros de interés se encuentran después de ajustar la curva de autocorrelación a las formas funcionales modeladas. [17]
El volumen de medición es una convolución de las geometrías de iluminación (excitación) y detección, que resultan de los elementos ópticos involucrados. El volumen resultante se describe matemáticamente mediante la función de dispersión de puntos (o PSF), que es esencialmente la imagen de una fuente puntual. La PSF se describe a menudo como un elipsoide (con límites poco nítidos) de unos pocos cientos de nanómetros de diámetro de foco y casi un micrómetro a lo largo del eje óptico. La forma varía significativamente (y tiene un gran impacto en las curvas FCS resultantes) dependiendo de la calidad de los elementos ópticos (es crucial evitar el astigmatismo y verificar la forma real de la PSF en el instrumento). En el caso de la microscopía confocal, y para pequeños orificios (alrededor de una unidad Airy), la PSF se aproxima bien mediante gaussianas:
donde es la intensidad pico, r y z son la posición radial y axial, y y son los radios radial y axial, y . Esta forma gaussiana se supone al derivar la forma funcional de la autocorrelación.
Por lo general , es de 200 a 300 nm y es de 2 a 6 veces más grande. [18] Una forma común de calibrar los parámetros de volumen de medición es realizar FCS en una especie con coeficiente de difusión y concentración conocidos (ver a continuación). Los coeficientes de difusión para fluoróforos comunes en agua se dan en una sección posterior.
La aproximación gaussiana funciona en distintos grados dependiendo de los detalles ópticos, y a veces se pueden aplicar correcciones para compensar los errores en la aproximación. [19]

La función de autocorrelación (temporal) es la correlación de una serie temporal consigo misma desplazada en el tiempo , en función de :
donde es la desviación de la intensidad media. La normalización (denominador) aquí es la más utilizada para FCS, porque entonces la correlación en , G (0), está relacionada con el número promedio de partículas en el volumen de medición.
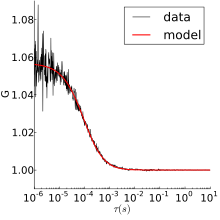
Como ejemplo, en la figura de la derecha se muestran los datos FCS sin procesar y su autocorrelación para la difusión libre de rodamina 6G. El gráfico de la parte superior muestra la intensidad de fluorescencia en función del tiempo. La intensidad fluctúa a medida que la rodamina 6G entra y sale del volumen focal. En el gráfico inferior se muestra la autocorrelación de los mismos datos. Se puede obtener información sobre la velocidad de difusión y la concentración utilizando uno de los modelos que se describen a continuación.
Para un perfil de iluminación gaussiano , la función de autocorrelación viene dada por la fórmula maestra general [20]
donde el vector denota el desplazamiento estocástico en el espacio de un fluoróforo después del tiempo . La expresión es válida si el número promedio de fluoróforos en el volumen focal es bajo y si se pueden ignorar los estados oscuros, etc., del fluoróforo. En particular, no se hizo ninguna suposición sobre el tipo de movimiento difusivo bajo investigación. La fórmula permite una interpretación de como (i) una probabilidad de retorno para parámetros de haz pequeños y (ii) la función generadora de momentos de si se varían.
Para extraer cantidades de interés, los datos de autocorrelación se pueden ajustar, generalmente mediante un algoritmo de mínimos cuadrados no lineal . La forma funcional del ajuste depende del tipo de dinámica (y de la geometría óptica en cuestión).
Las partículas fluorescentes utilizadas en el FCS son pequeñas y, por lo tanto, experimentan movimientos térmicos en solución. El experimento FCS más simple es, por lo tanto, la difusión tridimensional normal, para la cual la autocorrelación es:
donde es la relación entre los radios axial y radial del volumen de medición, y es el tiempo de residencia característico. Esta forma se derivó asumiendo un volumen de medición gaussiano. Normalmente, el ajuste tendría tres parámetros libres: G(0), y , a partir de los cuales se pueden obtener el coeficiente de difusión y la concentración de fluoróforo.
Con la normalización utilizada en la sección anterior, G (0) da el número medio de difusores en el volumen <N>, o equivalentemente, conociendo el tamaño del volumen de observación, la concentración media:
donde el volumen efectivo se obtiene integrando la forma gaussiana del volumen de medición y se expresa mediante:
Si las partículas que se difunden se ven obstaculizadas por obstáculos o empujadas por una fuerza (motores moleculares, flujo, etc.), la dinámica a menudo no se describe lo suficientemente bien mediante el modelo de difusión normal, donde el desplazamiento cuadrático medio (MSD) crece linealmente con el tiempo. En cambio, la difusión puede describirse mejor como difusión anómala , donde la dependencia temporal del MSD no es lineal, como en la ley de potencia:
donde es un coeficiente de difusión anómalo. "Difusión anómala" se refiere comúnmente solo a este modelo muy genérico, y no a las muchas otras posibilidades que podrían describirse como anómalas. Además, una ley de potencia es, en sentido estricto, la forma esperada solo para un rango estrecho de sistemas definidos rigurosamente, por ejemplo cuando la distribución de obstáculos es fractal . No obstante, una ley de potencia puede ser una aproximación útil para un rango más amplio de sistemas.
La función de autocorrelación FCS para la difusión anómala es:
donde el exponente anómalo es el mismo que el anterior y se convierte en un parámetro libre en el ajuste.
Utilizando FCS, se ha demostrado que el exponente anómalo es una indicación del grado de aglomeración molecular (es menor que uno y menor para mayores grados de aglomeración). [21]
Si hay partículas que se difunden con diferentes tamaños (coeficientes de difusión), es común ajustarlas a una función que es la suma de las formas de los componentes individuales:
donde la suma es sobre el número de tamaños diferentes de partículas, indexado por i, y da la ponderación, que está relacionada con el rendimiento cuántico y la concentración de cada tipo. Esto introduce nuevos parámetros, lo que hace que el ajuste sea más difícil ya que se debe buscar un espacio de mayor dimensión. El ajuste de mínimos cuadrados no lineal generalmente se vuelve inestable incluso con un pequeño número de s. Un esquema de ajuste más robusto, especialmente útil para muestras polidispersas, es el método de máxima entropía. [22]
Con difusión junto con un flujo uniforme con velocidad en dirección lateral, la autocorrelación es: [23]
¿Dónde está el tiempo de residencia promedio si solo hay flujo (sin difusión)?
Una amplia gama de posibles experimentos de FCS implican reacciones químicas que fluctúan continuamente desde el equilibrio debido a los movimientos térmicos (y luego se "relajan"). A diferencia de la difusión, que también es un proceso de relajación, las fluctuaciones causan cambios entre estados de diferentes energías. Un sistema muy simple que muestra relajación química sería un sitio de enlace estacionario en el volumen de medición, donde las partículas solo producen señal cuando están enlazadas (por ejemplo, por FRET, o si el tiempo de difusión es mucho más rápido que el intervalo de muestreo). En este caso, la autocorrelación es:
dónde
es el tiempo de relajación y depende de la cinética de la reacción (velocidades de activación y desactivación), y:
está relacionada con la constante de equilibrio K.
La mayoría de los sistemas con relajación química también muestran difusión medible, y la función de autocorrelación dependerá de los detalles del sistema. Si la difusión y la reacción química están desacopladas, la autocorrelación combinada es el producto de las autocorrelaciones química y difusiva.
Las autocorrelaciones anteriores suponen que las fluctuaciones no se deben a cambios en las propiedades fluorescentes de las partículas. Sin embargo, para la mayoría de los fluoróforos (bio)orgánicos (por ejemplo, la proteína fluorescente verde , la rodamina, los colorantes Cy3 y Alexa Fluor ), una fracción de las partículas iluminadas se excitan hasta un estado triplete (u otros estados de descomposición no radiativos) y luego no emiten fotones durante un tiempo de relajación característico . Normalmente es del orden de microsegundos, que suele ser menor que la dinámica de interés (por ejemplo, ) pero lo suficientemente grande como para ser medido. Se agrega un término multiplicativo a la autocorrelación para tener en cuenta el estado triplete. Para la difusión normal:
donde es la fracción de partículas que han entrado en el estado triplete y es el tiempo de relajación del estado triplete correspondiente. Si la dinámica de interés es mucho más lenta que la relajación del estado triplete, el componente de tiempo corto de la autocorrelación puede simplemente truncarse y el término triplete es innecesario.
La especie fluorescente utilizada en FCS es típicamente una biomolécula de interés que ha sido marcada con un fluoróforo (usando inmunohistoquímica , por ejemplo), o es un fluoróforo desnudo que se utiliza para investigar algún entorno de interés (por ejemplo, el citoesqueleto de una célula). La siguiente tabla muestra los coeficientes de difusión de algunos fluoróforos comunes en agua a temperatura ambiente y sus longitudes de onda de excitación.
El término FCS casi siempre se refiere a la medición de autocorrelación temporal de un solo punto y un solo canal, aunque el término "espectroscopia de correlación de fluorescencia" fuera de su contexto científico histórico no implica tal restricción. El término FCS ha sido ampliado en varias variaciones por diferentes investigadores, y cada extensión ha generado un nombre diferente (normalmente un acrónimo).
Mientras que la FCS es una medición puntual que proporciona el tiempo de difusión en un volumen de observación determinado, la svFCS es una técnica en la que se varía el punto de observación para medir los tiempos de difusión en diferentes tamaños de punto. La relación entre el tiempo de difusión y el área del punto es lineal y se puede representar gráficamente para descifrar la principal contribución del confinamiento. La curva resultante se denomina ley de difusión. Esta técnica se utiliza en biología para estudiar la organización de la membrana plasmática en células vivas.
donde es la intersección con el eje y. En caso de difusión browniana, . En caso de confinamiento debido a dominios aislados, mientras que en caso de dominios aislados, .
Estudios de svFCS en células vivas y artículos de simulación [32] [33] [34] [35] [36]
Espectroscopia de correlación de fluorescencia controlada por volumen de muestreo (SVC-FCS): [37]
FCS de escaneo z [38]
FCS con nanoaperturas: rompiendo la barrera de la difracción [39]
STED-FCS: [40]
El FCS se utiliza a veces para estudiar interacciones moleculares utilizando diferencias en los tiempos de difusión (por ejemplo, el producto de una reacción de asociación será más grande y, por lo tanto, tendrá tiempos de difusión mayores que los reactivos individualmente); sin embargo, el FCS es relativamente insensible a la masa molecular, como se puede ver en la siguiente ecuación que relaciona la masa molecular con el tiempo de difusión de partículas globulares (por ejemplo, proteínas):
donde es la viscosidad de la muestra y es la masa molecular de la especie fluorescente. En la práctica, los tiempos de difusión deben ser suficientemente diferentes (un factor de al menos 1,6), lo que significa que las masas moleculares deben diferir en un factor de 4. [41] La espectroscopia de correlación cruzada de fluorescencia de doble color (FCCS) mide las interacciones mediante la correlación cruzada de dos o más canales fluorescentes (un canal para cada reactivo), lo que distingue las interacciones con mayor sensibilidad que la FCS, en particular cuando el cambio de masa en la reacción es pequeño.
Este conjunto de métodos incluye número y brillo (N&B), [42] histograma de conteo de fotones (PCH), [43] análisis de distribución de intensidad de fluorescencia (FIDA), [44] y análisis cumulante. [45] y análisis de distribución de intensidad espacial. [46] También se informa la combinación de múltiples métodos. [47] La espectroscopia de correlación cruzada de fluorescencia supera la débil dependencia de la tasa de difusión en la masa molecular al observar la coincidencia multicolor. ¿Qué pasa con las homointeracciones? La solución está en el análisis de brillo. Estos métodos utilizan la heterogeneidad en la distribución de intensidad de la fluorescencia para medir el brillo molecular de diferentes especies en una muestra. Dado que los dímeros contendrán el doble de etiquetas fluorescentes que los monómeros, su brillo molecular será aproximadamente el doble que el de los monómeros. Como resultado, el brillo relativo es una medida sensible de oligomerización. El brillo molecular promedio ( ) está relacionado con la varianza ( ) y la intensidad promedio ( ) de la siguiente manera: [48]
Aquí y son la intensidad fraccionaria y el brillo molecular, respectivamente, de las especies .
Otro enfoque basado en FCS para estudiar las interacciones moleculares utiliza la transferencia de energía por resonancia de fluorescencia (FRET) en lugar de la fluorescencia, y se denomina FRET-FCS. [49] Con FRET, hay dos tipos de sondas, al igual que con FCCS; sin embargo, solo hay un canal y la luz solo se detecta cuando las dos sondas están muy cerca, lo suficientemente cerca como para garantizar una interacción. La señal FRET es más débil que con la fluorescencia, pero tiene la ventaja de que solo hay señal durante una reacción (aparte de la autofluorescencia ).
En la espectroscopia de correlación de fluorescencia de barrido (sFCS), el volumen de medición se mueve a lo largo de la muestra de una manera definida. La introducción del barrido está motivada por su capacidad de aliviar o eliminar varios problemas distintos que suelen encontrarse en la FCS estándar y, por lo tanto, de ampliar el rango de aplicabilidad de los métodos de correlación de fluorescencia en sistemas biológicos. [50]
Algunas variaciones de FCS solo son aplicables a microscopios láser de barrido en serie. La espectroscopia de correlación de imágenes y sus variaciones se implementaron en un microscopio confocal de barrido o un microscopio de barrido de dos fotones, pero se transfieren a otros microscopios, como un microscopio confocal de disco giratorio. Raster ICS (RICS), [51] y FCS sensible a la posición (PSFCS) [52] incorporan el retraso de tiempo entre partes del escaneo de la imagen en el análisis. Además, los escaneos de baja dimensión (por ejemplo, un anillo circular) [53] , solo posibles en un sistema de escaneo, pueden acceder a escalas de tiempo entre mediciones de un solo punto y de imagen completa. La trayectoria de escaneo también se ha diseñado para seguir partículas de forma adaptativa. [54]
Cualquiera de los métodos de espectroscopia de correlación de imágenes también se puede realizar en un microscopio confocal de disco giratorio, que en la práctica puede obtener velocidades de obtención de imágenes más rápidas en comparación con un microscopio confocal de barrido láser. Este enfoque se ha aplicado recientemente a la difusión en un entorno complejo que varía espacialmente, produciendo un mapa de resolución de píxeles de un coeficiente de difusión. [55] El mapeo espacial de la difusión con FCS se ha extendido posteriormente al sistema TIRF. [56] El mapeo espacial de la dinámica utilizando técnicas de correlación se había aplicado antes, pero solo en puntos dispersos [57] o con una resolución gruesa. [58]
Cuando el movimiento es lento (en biología, por ejemplo, la difusión en una membrana), obtener estadísticas adecuadas a partir de un experimento de FCS de un solo punto puede llevar un tiempo prohibitivamente largo. Se pueden obtener más datos realizando el experimento en múltiples puntos espaciales en paralelo, utilizando un microscopio confocal de barrido láser. Este enfoque se ha denominado espectroscopia de correlación de imágenes (ICS). [59] Luego, las mediciones se pueden promediar juntas.
Otra variación de ICS realiza una autocorrelación espacial en imágenes, lo que brinda información sobre la concentración de partículas. [60] Luego, la correlación se promedia en el tiempo. Si bien el ruido blanco de la cámara no se autocorrela en el tiempo, sí lo hace en el espacio; esto crea una amplitud de ruido blanco en la función de autocorrelación espacial que debe tenerse en cuenta al ajustar la amplitud de autocorrelación para encontrar la concentración de moléculas fluorescentes.
Una extensión natural de las versiones de correlación temporal y espacial es la ICS espacio-temporal (STICS). [58] En la STICS no hay un promedio explícito en el espacio o el tiempo (solo el promedio inherente a la correlación). En sistemas con movimiento no isotrópico (por ejemplo, flujo dirigido, difusión asimétrica), la STICS puede extraer la información direccional. Una variación que está estrechamente relacionada con la STICS (por la transformada de Fourier) es la espectroscopia de correlación de imágenes en el espacio k (kICS). [61]
También existen versiones de correlación cruzada de ICS, que pueden proporcionar la concentración, distribución y dinámica de moléculas fluorescentes colocalizadas. [59] Las moléculas se consideran colocalizadas cuando las contribuciones de fluorescencia individuales son indistinguibles debido a funciones superpuestas de intensidades de fluorescencia de punto-dispersión.
Fuente: [62]
PICS es una potente herramienta de análisis que resuelve correlaciones en escalas de tiempo de milisegundos y longitudes nanométricas. Adaptada de métodos de espectroscopia de correlación de imágenes espacio-temporales, [58] explota la alta precisión posicional del seguimiento de partículas individuales. Mientras que los métodos de seguimiento convencionales fallan si se cruzan múltiples trayectorias de partículas, este método funciona en principio para densidades de moléculas arbitrariamente grandes y parámetros dinámicos (por ejemplo, coeficientes de difusión, velocidades) siempre que se puedan identificar moléculas individuales. Es computacionalmente barato y robusto y permite identificar y cuantificar movimientos (por ejemplo, difusión, transporte activo, difusión confinada) dentro de un conjunto de partículas, sin ningún conocimiento a priori sobre la dinámica.
Hay una extensión de espectroscopia de correlación cruzada de imágenes de partículas (PICCS) disponible para procesos biológicos que involucran múltiples socios de interacción, como se puede observar mediante microscopía de dos colores. [63]
La obtención de imágenes de fluctuación óptica de superresolución (SOFI) es una técnica de superresolución que logra resoluciones espaciales por debajo del límite de difracción mediante análisis de posprocesamiento con ecuaciones de correlación, similares a FCS. Si bien los informes originales de SOFI utilizaban fluctuaciones de fluoróforos estacionarios y parpadeantes, FCS se ha combinado con SOFI, donde las fluctuaciones se producen a partir de sondas difusoras para producir mapas espaciales de superresolución de coeficientes de difusión. [64] Esto se ha aplicado para comprender la difusión y las propiedades espaciales de materiales porosos y confinados. Esto incluye agarosa [64] e hidrogeles PNIPAM sensibles a la temperatura, [65] cristales líquidos, [64] y polímeros separados en fases y condensados de ARN/proteína. [66]
La fluorescencia de reflexión interna total (TIRF) es un método de microscopía que solo es sensible a una capa delgada cerca de la superficie de un cubreobjetos, lo que minimiza en gran medida la fluorescencia de fondo. La FCS se ha extendido a ese tipo de microscopio y se denomina TIR-FCS. [67] Debido a que la intensidad de la fluorescencia en la TIRF disminuye exponencialmente con la distancia desde el cubreobjetos (en lugar de hacerlo de forma gaussiana con un confocal), la función de autocorrelación es diferente.
La microscopía de fluorescencia de lámina de luz o microscopía de imágenes de plano selectivo (SPIM) utiliza iluminación que se realiza perpendicularmente a la dirección de observación, mediante una lámina delgada de luz (láser). En determinadas condiciones, este principio de iluminación se puede combinar con la espectroscopia de correlación de fluorescencia, para permitir la obtención de imágenes con resolución espacial de la movilidad y las interacciones de partículas fluorescentes, como las proteínas marcadas con GFP, dentro de muestras biológicas vivas. [68]
Hay dos alternativas principales de no correlación al FCS que se utilizan ampliamente para estudiar la dinámica de las especies fluorescentes.
En FRAP , una región se expone brevemente a una luz intensa, fotoblanqueando irremediablemente los fluoróforos, y se obtiene una imagen de la recuperación de la fluorescencia debido a la difusión de los fluoróforos cercanos (no blanqueados). Una ventaja principal de FRAP sobre FCS es la facilidad de interpretación de experimentos cualitativos comunes en biología celular. Las diferencias entre líneas celulares, o regiones de una célula, o antes y después de la aplicación de un fármaco, a menudo se pueden caracterizar mediante una simple inspección de películas. Los experimentos FCS requieren un nivel de procesamiento y son más sensibles a influencias potencialmente confusas como: difusión rotacional, vibraciones, fotoblanqueo, dependencia de la iluminación y el color de fluorescencia, estadísticas inadecuadas, etc. Es mucho más fácil cambiar el volumen de medición en FRAP, lo que permite un mayor control. En la práctica, los volúmenes suelen ser mayores que en FCS. Si bien los experimentos FRAP suelen ser más cualitativos, algunos investigadores están estudiando FRAP cuantitativamente e incluyendo la dinámica de unión. [69] Una desventaja de FRAP en biología celular es la perturbación de radicales libres de la célula causada por el fotoblanqueo. También es menos versátil, ya que no puede medir la concentración ni la difusión rotacional ni la colocalización. El FRAP requiere una concentración significativamente mayor de fluoróforos que el FCS.
En el seguimiento de partículas, se miden las trayectorias de un conjunto de partículas, normalmente aplicando algoritmos de seguimiento de partículas a películas.[1] El seguimiento de partículas tiene la ventaja de que toda la información dinámica se mantiene en la medición, a diferencia del FCS, donde la correlación promedia la dinámica en una única curva suave. La ventaja es evidente en los sistemas que muestran difusión compleja, donde el cálculo directo del desplazamiento cuadrático medio permite una comparación directa con la difusión normal o de ley de potencia. Para aplicar el seguimiento de partículas, las partículas tienen que ser distinguibles y, por lo tanto, estar en una concentración menor que la requerida por el FCS. Además, el seguimiento de partículas es más sensible al ruido, que a veces puede afectar los resultados de manera impredecible.
Los recientes avances en nanofotónica ultravioleta han llevado al desarrollo del estudio de moléculas individuales en proteínas sin etiqueta excitándolas con luz ultravioleta profunda y estudiando los procesos dinámicos. [70] [71] [72]
La FCS de excitación de dos o tres fotones obtiene varias ventajas tanto en la resolución espacial como en la minimización del fotodaño/fotoblanqueo en muestras orgánicas y/o biológicas. [73] [74] [75] [76] [77]