El motivo de reconocimiento de ARN, RNP-1, es un supuesto dominio de unión a ARN de aproximadamente 90 aminoácidos que se sabe que se unen a ARN monocatenarios. Se encontró en muchas proteínas eucariotas . [1] [2] [3]
El grupo más grande de proteínas de unión a ARN monocatenario es la familia del motivo de reconocimiento de ARN eucariótico (RRM) que contiene una secuencia consenso RNP-1 de ocho aminoácidos. [4] [5]
Las proteínas RRM tienen una variedad de preferencias y funciones de unión al ARN, e incluyen ribonucleoproteínas nucleares heterogéneas ( hnRNP ), proteínas implicadas en la regulación del empalme alternativo (SR, U2AF2 , Sxl), componentes proteicos de pequeñas ribonucleoproteínas nucleares (U1 y U2 snRNP ) y proteínas que regulan la estabilidad y traducción del ARN ( PABP , La, Hu). [2] [3] [5] El RRM en el factor auxiliar de snRNP del factor de empalme heterodimérico U2 parece tener dos dominios similares a RRM con características especializadas para el reconocimiento de proteínas. [6] El motivo también aparece en algunas proteínas de unión a ADN monocatenarias.
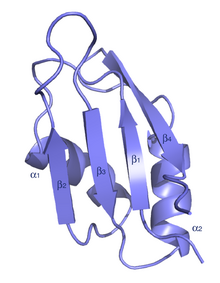
El RRM típico consta de cuatro cadenas beta antiparalelas y dos hélices alfa dispuestas en un pliegue beta-alfa-beta-beta-alfa-beta con cadenas laterales que se apilan con bases de ARN. En algunos casos, hay una tercera hélice presente durante la unión del ARN. [7] El RRM se analiza en varias publicaciones. [8] [9] [10]
A2BP1 ; ACF ; BOLL ; BRUNOL4 ; BRUNOL5; BRUNOL6; CCBL2; CGI-96; CIRBP ; CNOT4 ; CPEB2; CPEB3; CPEB4; CPSF7 ; CSTF2 ; CSTF2T ; CUGBP1 ; CUGBP2 ; D10S102; DAZ1 ; DAZ2 ; DAZ3 ; DAZ4; DAZAP1 ; DAZL ; ADNJC17; No molestar1; EIF3S4; EIF3S9; FEI4B ; EIF4H ; ELAVL1 ; ELAVL2 ; ELAVL3 ; ELAVL4 ; ENOX1; ENOX2 ; EWSR1 ; FUS ; FUSIP1 ; G3BP; G3BP1 ; G3BP2 ; GRSF1; HNRNPL ; HNRPA0; HNRPA1 ; HNRPA2B1 ; HNRPA3 ; HNRPAB ; HNRPC ; HNRPCL1; HNRPD ; HNRPDL ; HNRPF ; HNRPH1 ; HNRPH2 ; HNRPH3 ; HNRPL; HNRPLL; HNRPM; HNRPR; HRNBP1; HSU53209; HTATSF1 ; IGF2BP1 ; IGF2BP2 ; IGF2BP3 ; LARP7; MKI67IP ; MSI1 ; MSI2 ; MSSP-2; MTHFSD; MIEF2 ; NCBP2; NCL ; NOL8 ; NO NO ; P14; PABPC1 ; PABPC1L; PABPC3 ; PABPC4 ; PABPC5 ; PABPN1 ; POLDIP3 ; PPARGC1; PPARGC1A ; PPARGC1B ; PPIE ; PPIL4 ; PPRC1 ; PSPC1; PTBP1 ; PTBP2 ; PUF60 ; RALY ; RALILO ; RAVER1 ; RAVER2; RBM10 ; RBM11 ; RBM12; RBM12B; RBM14 ; RBM15 ; RBM15B; RBM16; RBM17 ; RBM18; RBM19 ; RBM22 ; RBM23 ; RBM24; RBM25 ; RBM26 ; RBM27; RBM28 ; RBM3 ; RBM32B; RBM33 ; RBM34 ; RBM35A; RBM35B; RBM38; RBM39 ; RBM4 ; RBM41; RBM42; RBM44; RBM45; RBM46; RBM47 ; RBM4B; RBM5 ; RBM7 ; RBM8A ; RBM9 ; RBMS1 ; RBMS2; RBMS3 ; RBMX ; RBMX2; RBMXL2; RBMY1A1 ; RBMY1B; RBMY1E; RBMY1F; RBMY2FP; RBPMS ; RBPMS2; RDBP ; RNPC3; RNPC4; RNPS1 ; VARILLA1; SAFB ; SAFB2 ; SART3 ; SETD1A ; SF3B14 ; SF3B4 ; SFPQ ; SFRS1 ; SFRS10 ; SFRS11 ; SFRS12 ; SFRS15; SFRS2 ; SFRS2B; SFRS3 ; SFRS4 ; SFRS5 ; SFRS6 ; SFRS7 ; SFRS9 ; SLIP; SLTM; SNRP70 ; SNRPA ; SNRPB2 ; GASTAR ; SR140 ; SRRP35; SSB ; SINCRIPCIÓN ; TAF15 ; TARBP ; THOC4 ; TIA1 ; TIAL1 ; TNRC4; TNRC6C; TRA2A ; TRSPAP1; TUT1; U1SNRNPBP; U2AF1 ; U2AF2 ; UHMK1 ; ZCRB1; ZNF638 ; ZRSR1 ; ZRSR2 ;