En el campo de la biología celular , el análisis de células individuales y el análisis subcelular [1] es el estudio de la genómica , la transcriptómica , la proteómica , la metabolómica y las interacciones célula-célula a nivel de células individuales. [2] [3] [4] El concepto de análisis de células individuales se originó en la década de 1970. Antes del descubrimiento de la heterogeneidad, el análisis de células individuales se refería principalmente al análisis o manipulación de una célula individual en una población masiva de células en una condición particular utilizando un microscopio óptico o electrónico. [5] Hasta la fecha, debido a la heterogeneidad observada en poblaciones de células eucariotas y procariotas, el análisis de una sola célula permite descubrir mecanismos que no se ven al estudiar una población masiva de células. [6] Tecnologías como la clasificación de células activadas por fluorescencia (FACS) permiten el aislamiento preciso de células individuales seleccionadas de muestras complejas, mientras que las tecnologías de partición de células individuales de alto rendimiento, [7] [8] [9] permiten el análisis molecular simultáneo de cientos o miles de células individuales sin clasificar; Esto es particularmente útil para el análisis de la variación del transcriptoma en células genotípicamente idénticas, lo que permite la definición de subtipos celulares que de otro modo serían indetectables. El desarrollo de nuevas tecnologías está aumentando nuestra capacidad para analizar el genoma y el transcriptoma de células individuales, [10] así como para cuantificar su proteoma y metaboloma . [11] [12] [13] Las técnicas de espectrometría de masas se han convertido en herramientas analíticas importantes para el análisis proteómico y metabolómico de células individuales. [14] [15] Los avances recientes han permitido cuantificar miles de proteínas en cientos de células individuales, [16] y, por lo tanto, hacen posibles nuevos tipos de análisis. [17] [18] La secuenciación in situ y la hibridación in situ con fluorescencia (FISH) no requieren que las células estén aisladas y se utilizan cada vez más para el análisis de tejidos. [19]
Muchas técnicas de análisis de células individuales requieren el aislamiento de células individuales. Los métodos que se utilizan actualmente para el aislamiento de células individuales incluyen: clasificación digital dielectroforética, digestión enzimática, FACS , trampas hidrodinámicas, microdisección por captura láser , selección manual, microfluídica , impresión por inyección de tinta o IJP, micromanipulación , dilución en serie y pinzas Raman.
La recolección manual de células individuales es un método en el que las células en una suspensión se observan bajo un microscopio y se seleccionan individualmente utilizando una micropipeta . [20] [21] Las pinzas Raman son una técnica en la que la espectroscopia Raman se combina con pinzas ópticas , que utilizan un rayo láser para atrapar y manipular las células. [22]
El método de clasificación digital dielectroforética utiliza una matriz de electrodos controlada por semiconductores en un chip microfluídico para atrapar células individuales en jaulas dielectroforéticas (DEP). La identificación de las células se garantiza mediante la combinación de marcadores fluorescentes con la observación de imágenes. La entrega precisa se garantiza mediante el movimiento controlado por semiconductores de las jaulas DEP en la celda de flujo.
La impresión por inyección de tinta [23] combina la microfluídica con MEMS en un chip CMOS para proporcionar un control individual sobre una gran cantidad de boquillas de impresión, utilizando la misma tecnología que la impresión por inyección de tinta doméstica. La IJP permite ajustar la fuerza de corte a la expulsión de la muestra, lo que mejora enormemente la capacidad de supervivencia de las células. Este enfoque, cuando se combina con la inspección óptica y el reconocimiento de imágenes impulsado por IA, no solo garantiza la dispensación de células individuales en la placa de pocillos u otro medio, sino que también puede calificar la muestra de células para la calidad de la muestra, rechazando células defectuosas, desechos y fragmentos.
El desarrollo de biochips microfluídicos basados en la hidrodinámica ha ido en aumento a lo largo de los años. En esta técnica, las células o partículas se atrapan en una región particular para su análisis de células individuales (SCA), normalmente sin la aplicación de campos de fuerza externos como ópticos, eléctricos, magnéticos o acústicos. Existe la necesidad de explorar los conocimientos del SCA en el estado natural de la célula y el desarrollo de estas técnicas es sumamente esencial para ese estudio. Los investigadores han destacado el vasto campo potencial que se necesita explorar para desarrollar dispositivos de biochip que se adapten a las demandas del mercado y de los investigadores. La microfluídica hidrodinámica facilita el desarrollo de aplicaciones pasivas de laboratorio en chip. Una revisión reciente da cuenta de los avances recientes en este campo, junto con sus mecanismos, métodos y aplicaciones. [24]
El método de clasificación digital dielectroforética utiliza una matriz de electrodos controlada por semiconductores en un chip microfluídico para atrapar células individuales en jaulas dielectroforéticas (DEP). La identificación de las células se garantiza mediante la combinación de marcadores fluorescentes con la observación de imágenes. La entrega precisa se garantiza mediante el movimiento controlado por semiconductores de las jaulas DEP en la celda de flujo.
Las trampas hidrodinámicas permiten aislar una célula individual en una "trampa" en un momento determinado mediante transporte microfluídico pasivo. La cantidad de células aisladas se puede manipular en función de la cantidad de trampas del sistema.
La técnica de microdisección por captura láser utiliza un láser para diseccionar y separar células individuales o secciones de muestras de tejido de interés. Los métodos implican la observación de una célula bajo un microscopio, de modo que se pueda identificar y etiquetar una sección para su análisis, de modo que el láser pueda cortar la célula. Luego, se puede extraer la célula para su análisis.
La recolección manual de células individuales es un método en el que las células de una suspensión se observan bajo un microscopio y se seleccionan individualmente utilizando una micropipeta .
La microfluídica permite aislar células individuales para realizar análisis posteriores. Los principios siguientes describen los distintos procesos microfluídicos para la separación de células individuales: aislamiento basado en gotas en aceite, valvulería neumática de membrana y trampas hidrodinámicas para células. La microfluídica basada en gotas en aceite utiliza canales llenos de aceite para contener gotas acuosas separadas. Esto permite contener y aislar la célula individual del interior de los canales basados en aceite. Las válvulas neumáticas de membrana utilizan la manipulación de la presión del aire para aislar células individuales mediante la deflexión de la membrana. La manipulación de la fuente de presión permite abrir o cerrar canales en una red microfluídica. Normalmente, el sistema requiere un operador y tiene un rendimiento limitado.
La técnica de pinzas Raman combina el uso de la espectroscopia Raman y pinzas ópticas , que utilizan un rayo láser para atrapar y manipular las células.
El desarrollo de biochips microfluídicos basados en la hidrodinámica ha ido en aumento a lo largo de los años. En esta técnica, las células quedan atrapadas en una región particular para su análisis unicelular (SCA). Esto suele ocurrir sin la aplicación de campos de fuerza externos, como ópticos, eléctricos, magnéticos o acústicos. Existe la necesidad de explorar los conocimientos del SCA en el estado natural de la célula, y el desarrollo de estas técnicas es sumamente esencial para ese estudio. Los investigadores han destacado la necesidad de desarrollar dispositivos de biochip que se adapten a las demandas del mercado y de los investigadores. La microfluídica hidrodinámica facilita el desarrollo de aplicaciones pasivas de laboratorio en chip.
La genómica unicelular depende en gran medida del aumento de las copias de ADN que se encuentran en la célula para que haya suficiente para secuenciar. Esto ha llevado al desarrollo de estrategias para la amplificación del genoma completo (WGA). Actualmente, las estrategias WGA se pueden agrupar en tres categorías:
En muchos estudios comparativos se informa que el adaptador de enlace PCR WGA tiene el mejor rendimiento para el análisis de mutaciones de células individuales diploides, gracias a su efecto de pérdida alélica muy bajo, [25] [26] [27] y para el perfil de variación del número de copias debido a su bajo ruido, tanto con aCGH como con secuenciación de paso bajo NGS. [28] [29] Este método solo es aplicable a células humanas, tanto fijadas como no fijadas.
Una técnica de WGA ampliamente adoptada se llama reacción en cadena de la polimerasa con oligonucleótidos degenerados (DOP-PCR). Este método utiliza el método de amplificación de ADN bien establecido PCR para intentar amplificar todo el genoma utilizando un gran conjunto de cebadores . Aunque simple, se ha demostrado que este método tiene una cobertura genómica muy baja. Una mejora en DOP-PCR es la amplificación por desplazamiento múltiple (MDA), que utiliza cebadores aleatorios y una enzima de alta fidelidad , generalmente la ADN polimerasa Φ29 , para lograr la amplificación de fragmentos más grandes y una mayor cobertura del genoma que DOP-PCR. A pesar de estas mejoras, MDA todavía tiene un sesgo dependiente de la secuencia (ciertas partes del genoma se amplifican más que otras debido a su secuencia). El método que se ha demostrado que evita en gran medida el sesgo observado en DOP-PCR y MDA es Multiple Annealing and Looping–Based Amplification Cycles (MALBAC). El sesgo en este sistema se reduce copiando solo la cadena de ADN original en lugar de hacer copias de copias. La principal desventaja de utilizar MALBA es que tiene una precisión reducida en comparación con DOP-PCR y MDA debido a la enzima utilizada para copiar el ADN. [11] Una vez amplificado mediante cualquiera de las técnicas anteriores, el ADN se puede secuenciar mediante Sanger o secuenciación de próxima generación (NGS).
El estudio del genoma a nivel de células individuales tiene dos aplicaciones principales. Una de ellas es el seguimiento de los cambios que se producen en las poblaciones bacterianas, donde a menudo se observan diferencias fenotípicas. Estas diferencias no se detectan mediante la secuenciación en masa de una población, pero se pueden observar en la secuenciación de células individuales. [30] La segunda aplicación principal es el estudio de la evolución genética del cáncer. Dado que las células cancerosas mutan constantemente, resulta de gran interés observar cómo evolucionan los cánceres a nivel genético. Estos patrones de mutaciones somáticas y aberraciones del número de copias se pueden observar mediante la secuenciación de células individuales. [2]
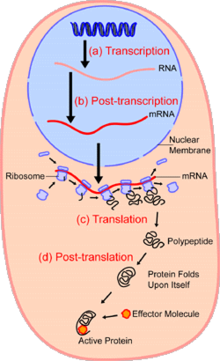
La transcriptómica de células individuales utiliza técnicas de secuenciación similares a la genómica de células individuales o la detección directa mediante hibridación in situ con fluorescencia . El primer paso para cuantificar el transcriptoma es convertir el ARN en ADNc mediante la transcriptasa inversa , de modo que el contenido de la célula pueda secuenciarse mediante métodos de NGS, como se hacía en genómica. Una vez convertido, no hay suficiente ADNc para secuenciar, por lo que se aplican las mismas técnicas de amplificación de ADN analizadas en la genómica de células individuales al ADNc para hacer posible la secuenciación. [2] Alternativamente, se utilizan compuestos fluorescentes unidos a sondas de hibridación de ARN para identificar secuencias específicas y la aplicación secuencial de diferentes sondas de ARN creará un transcriptoma completo. [31] [32]
El objetivo de la transcriptómica de células individuales es determinar qué genes se expresan en cada célula. El transcriptoma se utiliza a menudo para cuantificar la expresión génica en lugar del proteoma debido a la dificultad que actualmente se asocia con la amplificación de los niveles de proteínas. [2]
Existen tres razones principales por las que se ha estudiado la expresión génica utilizando esta técnica: para estudiar la dinámica génica, el empalme del ARN y la tipificación celular. La dinámica génica se estudia generalmente para determinar qué cambios en la expresión génica afectan a las diferentes características celulares. Por ejemplo, este tipo de análisis transcriptómico se ha utilizado a menudo para estudiar el desarrollo embrionario. Los estudios de empalme del ARN se centran en comprender la regulación de las diferentes isoformas de transcripción . La transcriptómica de células individuales también se ha utilizado para la tipificación celular, donde los genes expresados en una célula se utilizan para identificar tipos de células. El objetivo principal de la tipificación celular es encontrar una forma de determinar la identidad de las células que no tienen marcadores genéticos conocidos . [2]
La expresión del ARN puede servir como indicador de la abundancia de proteínas. Sin embargo, la abundancia de proteínas está determinada por la compleja interacción entre la expresión del ARN y los procesos postranscripcionales. Si bien es un desafío técnico, la traducción se puede monitorear mediante el análisis de los ribosomas en células individuales. [33]
Existen tres enfoques principales para la proteómica de células individuales: métodos basados en anticuerpos, métodos basados en proteínas fluorescentes y métodos basados en espectroscopia de masas. [34] [18]
Los métodos basados en anticuerpos utilizan anticuerpos diseñados para unirse a las proteínas de interés, lo que permite identificar la abundancia relativa de múltiples objetivos individuales mediante una de varias técnicas diferentes.
Imágenes: Los anticuerpos se pueden unir a moléculas fluorescentes como puntos cuánticos o etiquetarse con fluoróforos orgánicos para su detección mediante microscopía de fluorescencia . Dado que a cada anticuerpo se le unen puntos cuánticos de diferentes colores o fluoróforos únicos, es posible identificar múltiples proteínas diferentes en una sola célula. Los puntos cuánticos se pueden lavar de los anticuerpos sin dañar la muestra, lo que hace posible realizar múltiples rondas de cuantificación de proteínas utilizando este método en la misma muestra. [35] Para los métodos basados en fluoróforos orgánicos, las etiquetas fluorescentes se unen mediante un enlace reversible como un híbrido de ADN (que se puede fundir/disociar en condiciones de baja salinidad) [36] o inactivar químicamente [37] , lo que permite múltiples ciclos de análisis, con 3-5 objetivos cuantificados por ciclo. Estos enfoques se han utilizado para cuantificar la abundancia de proteínas en muestras de biopsia de pacientes (por ejemplo, cáncer) para mapear la expresión variable de proteínas en tejidos y/o tumores [37] y para medir los cambios en la expresión de proteínas y la señalización celular en respuesta al tratamiento del cáncer. [36]
Citometría de masas : los isótopos de metales raros, que normalmente no se encuentran en las células o los tejidos, se pueden unir a los anticuerpos individuales y detectarse mediante espectrometría de masas para la identificación simultánea y sensible de proteínas. [38] Estas técnicas se pueden multiplexar en gran medida para la cuantificación simultánea de muchos objetivos (paneles de hasta 38 marcadores) en células individuales. [39]
Cuantificación de anticuerpos-ADN: otro método basado en anticuerpos convierte los niveles de proteína en niveles de ADN. [34] La conversión a ADN permite amplificar los niveles de proteína y utilizar la NGS para cuantificar las proteínas. En uno de estos enfoques, se seleccionan dos anticuerpos para cada proteína que se necesita cuantificar. Luego, los dos anticuerpos se modifican para que tengan ADN monocatenario conectado a ellos que sea complementario. Cuando los dos anticuerpos se unen a una proteína, las cadenas complementarias se aparearán y producirán un segmento de ADN bicatenario que luego se puede amplificar mediante PCR. Cada par de anticuerpos diseñados para una proteína se etiqueta con una secuencia de ADN diferente. Luego, el ADN amplificado a partir de la PCR se puede secuenciar y los niveles de proteína se pueden cuantificar. [40]
En la proteómica basada en espectroscopia de masas hay tres pasos principales necesarios para la identificación de péptidos: preparación de la muestra, separación de péptidos e identificación de péptidos. Varios grupos se han centrado en ovocitos o células en una etapa de escisión muy temprana, ya que estas células son inusualmente grandes y proporcionan suficiente material para el análisis. [41] [42] [43] [44] Otro enfoque, la proteómica de células individuales por espectrometría de masas (SCoPE-MS), ha cuantificado miles de proteínas en células de mamíferos con tamaños celulares típicos (diámetro de 10-15 μm) combinando células portadoras y códigos de barras de células individuales. [45] [46] La segunda generación, SCoPE2, [47] [48] aumentó el rendimiento mediante la preparación de muestras automatizada y miniaturizada; [49] También mejoró la confiabilidad cuantitativa y la cobertura del proteoma mediante la optimización basada en datos de LC-MS/MS [50] y la identificación de péptidos. [51] La sensibilidad y la consistencia de estos métodos se han mejorado aún más mediante la priorización, [52] y la preparación masiva de muestras en paralelo en gotas de tamaño nanométrico. [53] Otra dirección para el análisis de proteínas de una sola célula se basa en un marco escalable de adquisición independiente de datos multiplexados (plexDIA) que permite ahorrar tiempo mediante el análisis paralelo de iones de péptidos y muestras de proteínas, logrando así ganancias multiplicativas en el rendimiento. [54] [55] [56]
La separación de proteínas de diferentes tamaños se puede lograr mediante electroforesis capilar (CE) o cromatografía líquida (LC) (el uso de cromatografía líquida con espectroscopia de masas también se conoce como LC-MS). [42] [43] [44] [45] Este paso da orden a los péptidos antes de la cuantificación mediante espectroscopia de masas en tándem (MS/MS). La principal diferencia entre los métodos de cuantificación es que algunos usan etiquetas en los péptidos, como etiquetas de masa en tándem (TMT) o etiquetas de dimetilo que se usan para identificar de qué célula proviene una determinada proteína (las proteínas que provienen de cada célula tienen una etiqueta diferente), mientras que otros no usan etiquetas (cuantifican las células individualmente). Luego, los datos de espectroscopia de masas se analizan ejecutando los datos a través de bases de datos que convierten la información sobre los péptidos identificados en cuantificación de los niveles de proteína. [42] [43] [44] [45] [57] Estos métodos son muy similares a los utilizados para cuantificar el proteoma de células a granel , con modificaciones para acomodar el volumen de muestra muy pequeño. [58]
Se puede utilizar una gran variedad de técnicas de ionización para analizar células individuales. La elección del método de ionización es crucial para la detección del analito. Puede ser decisivo qué tipo de compuestos son ionizables y en qué estado aparecen, por ejemplo, la carga y la posible fragmentación de los iones. [59] En los párrafos siguientes se mencionan algunos ejemplos de ionización.
Una de las posibles formas de medir el contenido de células individuales es la nano-DESI (ionización por electrospray de desorción por nanopulverización). A diferencia de la ionización por electrospray de desorción , que es una técnica de desorción, la nano-DESI es una técnica de extracción de líquidos que permite el muestreo de superficies pequeñas, por lo que es adecuada para el análisis de células individuales. En la nano-DESI, se colocan dos capilares de sílice fundida en forma de V, cerrando un ángulo de aproximadamente 85 grados. Los dos capilares están en contacto, por lo que se puede formar un puente líquido entre ellos y permitir el muestreo de superficies tan pequeñas como una sola célula. El capilar primario suministra el disolvente a la superficie de la muestra donde se produce la extracción y el capilar secundario dirige el disolvente con las moléculas extraídas a la entrada de MS. La espectrometría de masas (MS) nano-DESI permite la elaboración de perfiles moleculares sensibles y la cuantificación de especies endógenas tan pequeñas como unos pocos cientos de fmol-s en células individuales de una manera de mayor rendimiento. Lanekoff et al. Se identificaron 14 aminoácidos, 6 metabolitos y varias moléculas lipídicas a partir de células de una sola mejilla utilizando nano-DESI MS. [60]
En la ionización por electrospray por ablación láser (LAESI), se utiliza un láser para extirpar la superficie de la muestra y las moléculas emitidas se ionizan en la fase gaseosa mediante gotitas cargadas de electrospray. De manera similar a la DESI, la ionización ocurre en condiciones ambientales . Anderton et al . utilizaron esta técnica de ionización acoplada a un espectrómetro de masas por transformada de Fourier para analizar 200 células individuales de Allium cepa (cebolla roja) en alta resolución espacial. [61]
La espectrometría de masas de iones secundarios (SIMS) es una técnica similar a la DESI, pero mientras que la DESI es una técnica de ionización ambiental, la SIMS se realiza en vacío . La superficie de la muestra sólida es bombardeada por un haz altamente enfocado de iones primarios . A medida que golpean la superficie, las moléculas se emiten desde la superficie y se ionizan. La elección de los iones primarios determina el tamaño del haz y también el grado de ionización y fragmentación. [62] Pareek et al. realizaron metabolómica para rastrear cómo se sintetizan las purinas dentro de los purinosomas y utilizaron el etiquetado de isótopos y la obtención de imágenes SIMS para observar directamente los puntos críticos de actividad metabólica dentro de las células HeLa congeladas. [63]
En la desorción e ionización láser asistida por matriz (MALDI), la muestra se incorpora a una matriz química que es capaz de absorber energía de un láser. De manera similar a SIMS, la ionización ocurre en el vacío. La irradiación láser elimina el material de la matriz de la superficie y da como resultado partículas de matriz en fase gaseosa cargadas; las moléculas de analito se ionizan a partir de esta matriz química cargada. Liu et al. utilizaron MALDI-MS para detectar ocho fosfolípidos de células A549 individuales. [64] Las imágenes MALDI MS se pueden utilizar para la metabolómica espacial y el análisis de células individuales. [65] [66]
El objetivo de estudiar el proteoma es comprender mejor la actividad de las células a nivel de células individuales. Dado que las proteínas son responsables de determinar cómo actúa la célula, comprender el proteoma de una sola célula brinda la mejor comprensión de cómo funciona una célula y cómo cambia la expresión génica en una célula debido a diferentes estímulos ambientales. Aunque la transcriptómica tiene el mismo propósito que la proteómica, no es tan precisa para determinar la expresión génica en las células, ya que no tiene en cuenta la regulación postranscripcional . [12] La transcriptómica sigue siendo importante ya que estudiar la diferencia entre los niveles de ARN y los niveles de proteína podría brindar información sobre qué genes están regulados postranscripcionalmente.
Existen cuatro métodos principales que se utilizan para cuantificar el metaboloma de células individuales: detección basada en fluorescencia, biosensores de fluorescencia, biosensores FRET y espectroscopia de masas. Los primeros tres métodos enumerados utilizan microscopía de fluorescencia para detectar moléculas en una célula. Por lo general, estos ensayos utilizan pequeñas etiquetas fluorescentes adheridas a las moléculas de interés, sin embargo, se ha demostrado que esto es demasiado invasivo para la metabolómica de células individuales y altera la actividad de los metabolitos. La solución actual a este problema es utilizar proteínas fluorescentes que actuarán como detectores de metabolitos, emitiendo fluorescencia siempre que se unan a un metabolito de interés. [67]
La espectroscopia de masas se está convirtiendo en el método más utilizado para la metabolómica de células individuales. Sus ventajas son que no es necesario desarrollar proteínas fluorescentes para todas las moléculas de interés y es capaz de detectar metabolitos en el rango de femtomoles . [15] De manera similar a los métodos analizados en proteómica, también ha habido éxito en la combinación de la espectroscopia de masas con técnicas de separación como la electroforesis capilar para cuantificar metabolitos. Este método también es capaz de detectar metabolitos presentes en concentraciones de femtomoles. [67] Se ha demostrado que otro método que utiliza micromuestreo capilar combinado con espectrometría de masas con separación por movilidad iónica mejora la cobertura molecular y la separación iónica para la metabolómica de células individuales. [21] [68] Los investigadores están tratando de desarrollar una técnica que pueda satisfacer lo que las técnicas actuales carecen: alto rendimiento, mayor sensibilidad para metabolitos que tienen una menor abundancia o que tienen bajas eficiencias de ionización, buena replicabilidad y que permiten la cuantificación de metabolitos. [69]
El objetivo de la metabolómica de células individuales es obtener una mejor comprensión a nivel molecular de los principales temas biológicos, como el cáncer, las células madre, el envejecimiento y el desarrollo de la resistencia a los fármacos. En general, el objetivo de la metabolómica es principalmente comprender cómo las células se enfrentan a las tensiones ambientales a nivel molecular y proporcionar una comprensión más dinámica de las funciones celulares. [67]
Los ensayos transcriptómicos de células individuales han permitido reconstruir trayectorias de desarrollo. La ramificación de estas trayectorias describe la diferenciación celular. Se han desarrollado varios métodos para reconstruir trayectorias de desarrollo ramificadas a partir de datos transcriptómicos de células individuales. [70] [71 ] [72] [73] [74] Utilizan varios conceptos matemáticos avanzados, desde el transporte óptimo [72] hasta los gráficos principales. [73] Algunas bibliotecas de software para la reconstrucción y visualización de trayectorias de diferenciación de linaje están disponibles gratuitamente en línea. [75]
Las interacciones célula-célula se caracterizan por interacciones estables y transitorias.