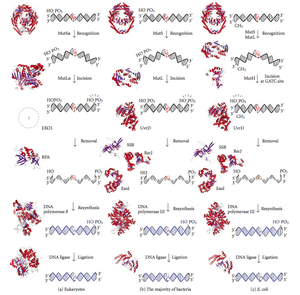

La reparación de desajustes del ADN ( MMR ) es un sistema para reconocer y reparar inserciones, eliminaciones e incorporaciones erróneas de bases que pueden surgir durante la replicación y recombinación del ADN , así como para reparar algunas formas de daño al ADN . [1] [2]
La reparación de errores de apareamiento es específica de cada cadena. Durante la síntesis de ADN, la cadena recién sintetizada (hija) generalmente incluye errores. Para comenzar la reparación, la maquinaria de reparación de errores de apareamiento distingue la cadena recién sintetizada de la cadena molde (parental). En las bacterias gramnegativas, la hemimetilación transitoria distingue las cadenas (la parental está metilada y la hija no). Sin embargo, en otros procariotas y eucariotas, el mecanismo exacto no está claro. Se sospecha que, en eucariotas, el ADN de la cadena rezagada recién sintetizada contiene mellas transitoriamente (antes de ser sellado por la ADN ligasa) y proporciona una señal que dirige los sistemas de corrección de errores de apareamiento a la cadena apropiada. Esto implica que estas mellas deben estar presentes en la cadena líder, y recientemente se han encontrado pruebas de esto. [3] Trabajos recientes [4] han demostrado que las muescas son sitios para la carga dependiente de RFC de la abrazadera deslizante de replicación, antígeno nuclear de células proliferantes (PCNA), de una manera específica en cuanto a orientación, de modo que una cara de la proteína con forma de rosquilla se yuxtapone hacia el extremo 3'-OH en la muesca. El PCNA cargado luego dirige la acción de la endonucleasa MutLalpha [5] a la cadena hija en presencia de un desajuste y MutSalpha o MutSbeta.
Cualquier mutación que altere la estructura superhelicoidal del ADN conlleva el potencial de comprometer la estabilidad genética de una célula. El hecho de que los sistemas de detección y reparación de daños sean tan complejos como la propia maquinaria de replicación pone de relieve la importancia que la evolución ha concedido a la fidelidad del ADN.
Los ejemplos de bases no coincidentes incluyen un emparejamiento G/T o A/C (ver reparación del ADN ). Los desajustes se deben comúnmente a la tautomerización de bases durante la replicación del ADN. El daño se repara mediante el reconocimiento de la deformidad causada por el desajuste, la determinación de la cadena molde y la que no es molde, y la excisión de la base incorporada incorrectamente y su reemplazo por el nucleótido correcto . El proceso de eliminación implica más que solo el nucleótido no coincidente en sí. Se pueden eliminar unos pocos o hasta miles de pares de bases de la cadena de ADN recién sintetizada.
La reparación de errores de emparejamiento es un proceso altamente conservado desde los procariotas hasta los eucariotas . La primera evidencia de la reparación de errores de emparejamiento se obtuvo de S. pneumoniae (los genes hexA y hexB ). Trabajos posteriores en E. coli han identificado una serie de genes que, cuando se inactivan mediante mutaciones , causan cepas hipermutables. Los productos genéticos se denominan, por lo tanto, proteínas "Mut" y son los principales componentes activos del sistema de reparación de errores de emparejamiento. Tres de estas proteínas son esenciales para detectar el error de emparejamiento y dirigir la maquinaria de reparación hacia él: MutS , MutH y MutL (MutS es un homólogo de HexA y MutL de HexB).
MutS forma un dímero (MutS 2 ) que reconoce la base desapareada en la hebra hija y se une al ADN mutado. MutH se une a los sitios hemimetilados a lo largo del ADN hijo, pero su acción es latente, siendo activada solo al contacto con un dímero MutL (MutL 2 ), que se une al complejo MutS-ADN y actúa como mediador entre MutS 2 y MutH, activando este último. El ADN se extiende en bucle para buscar el sitio de metilación d(GATC) más cercano al desapareamiento, que podría estar hasta a 1 kb de distancia. Tras la activación por el complejo MutS-ADN, MutH corta la hebra hija cerca del sitio hemimetilado. MutL recluta la helicasa UvrD (ADN Helicasa II) para separar las dos hebras con una polaridad específica de 3' a 5'. Luego, todo el complejo MutSHL se desliza a lo largo del ADN en la dirección del desapareamiento, liberando la hebra para ser escindida a medida que avanza. Una exonucleasa sigue el complejo y digiere la cola de ss-ADN. La exonucleasa reclutada depende de qué lado del desajuste MutH corta la hebra: 5' o 3'. Si la muesca hecha por MutH está en el extremo 5' del desajuste, se utiliza RecJ o ExoVII (ambas exonucleasas 5' a 3'). Sin embargo, si la muesca está en el extremo 3' del desajuste, se utiliza ExoI (una enzima 3' a 5').
Todo el proceso termina más allá del sitio de desajuste, es decir, tanto el sitio en sí como los nucleótidos circundantes se eliminan por completo. El espacio entre las hebras individuales creado por la exonucleasa puede luego ser reparado por la ADN polimerasa III (con la ayuda de la proteína de unión a hebras individuales), que utiliza la otra hebra como plantilla, y finalmente sellado por la ADN ligasa. Luego, la ADN metilasa metila rápidamente la hebra hija.
Cuando se une, el dímero MutS 2 dobla la hélice de ADN y protege aproximadamente 20 pares de bases. Tiene una actividad ATPasa débil y la unión de ATP conduce a la formación de estructuras terciarias en la superficie de la molécula. La estructura cristalina de MutS revela que es excepcionalmente asimétrica y, si bien su conformación activa es un dímero, solo una de las dos mitades interactúa con el sitio de desajuste.
En eucariotas, los homólogos de MutS forman dos heterodímeros principales: Msh2 /Msh6 (MutSα) y Msh2 /Msh3 (MutSβ). La vía MutSα está involucrada principalmente en la sustitución de bases y la reparación de errores de apareamiento de bucles pequeños. La vía MutSβ también está involucrada en la reparación de bucles pequeños, además de la reparación de bucles grandes (bucles de ~10 nucleótidos). Sin embargo, MutSβ no repara las sustituciones de bases.
MutL también tiene una actividad ATPasa débil (utiliza ATP para fines de movimiento). Forma un complejo con MutS y MutH, aumentando la huella de MutS en el ADN.
Sin embargo, la procesividad (la distancia que la enzima puede recorrer a lo largo del ADN antes de disociarse) de UvrD es de solo ~40–50 pb. Debido a que la distancia entre la muesca creada por MutH y el desajuste puede promediar ~600 pb, si no hay otro UvrD cargado, la sección desenrollada queda libre para volver a anidarse con su cadena complementaria, forzando al proceso a comenzar de nuevo. Sin embargo, cuando es asistida por MutL, la tasa de carga de UvrD aumenta considerablemente. Si bien la procesividad (y la utilización de ATP) de las moléculas individuales de UvrD sigue siendo la misma, el efecto total sobre el ADN aumenta considerablemente; el ADN no tiene posibilidad de volver a anidarse, ya que cada UvrD desenrolla 40-50 pb de ADN, se disocia y luego es reemplazado inmediatamente por otro UvrD, repitiendo el proceso. Esto expone grandes secciones de ADN a la digestión con exonucleasa , lo que permite la escisión rápida (y el reemplazo posterior) del ADN incorrecto.
Los eucariotas tienen cinco homólogos de MutL designados como MLH1, MLH2, MLH3, PMS1 y PMS2. Forman heterodímeros que imitan a MutL en E. coli . Los homólogos humanos de MutL procariota forman tres complejos denominados MutLα, MutLβ y MutLγ. El complejo MutLα está formado por las subunidades MLH1 y PMS2, el heterodímero MutLβ está formado por MLH1 y PMS1, mientras que MutLγ está formado por MLH1 y MLH3. MutLα actúa como una endonucleasa que introduce roturas de cadena en la cadena hija tras la activación por el desajuste y otras proteínas necesarias, MutSα y PCNA. Estas interrupciones de cadena sirven como puntos de entrada para una actividad de exonucleasa que elimina el ADN desajuste. Los papeles que desempeñan MutLβ y MutLγ en la reparación de los desajustes son menos comprendidos.
MutH es una endonucleasa muy débil que se activa una vez unida a MutL (que a su vez está unida a MutS). Corta el ADN no metilado y la hebra no metilada del ADN hemimetilado, pero no corta el ADN completamente metilado. Los experimentos han demostrado que la reparación de los desajustes es aleatoria si ninguna de las hebras está metilada. [ cita requerida ] Estos comportamientos llevaron a la propuesta de que MutH determina qué hebra contiene el desajuste. MutH no tiene homólogo eucariota. Su función de endonucleasa es asumida por los homólogos de MutL, que tienen cierta actividad exonucleasa 5'-3' especializada. El sesgo de hebra para eliminar los desajustes de la hebra hija recién sintetizada en eucariotas puede ser proporcionado por los extremos 3' libres de los fragmentos de Okazaki en la nueva hebra creada durante la replicación.
El PCNA y la pinza deslizante β se asocian con MutSα/β y MutL, respectivamente. Aunque los informes iniciales sugirieron que el complejo PCNA-MutSα puede mejorar el reconocimiento de desajustes, [6] se ha demostrado recientemente [7] que no hay un cambio aparente en la afinidad de MutSα por un desajuste en presencia o ausencia de PCNA. Además, los mutantes de MutSα que no pueden interactuar con PCNA in vitro muestran la capacidad de llevar a cabo el reconocimiento de desajustes y la escisión de desajustes a niveles cercanos al tipo salvaje. Dichos mutantes son defectuosos en la reacción de reparación dirigida por una rotura de la cadena 5', lo que sugiere por primera vez que MutSα funciona en un paso posterior a la escisión de la reacción.
Las mutaciones en los homólogos humanos de las proteínas Mut afectan la estabilidad genómica, lo que puede dar lugar a una inestabilidad de microsatélites (MSI), implicada en algunos cánceres humanos. En concreto, los cánceres colorrectales hereditarios no polipósicos ( HNPCC o síndrome de Lynch) se atribuyen a variantes de línea germinal dañinas en los genes que codifican los homólogos MutS y MutL MSH2 y MLH1 respectivamente, que se clasifican así como genes supresores de tumores. Un subtipo de HNPCC, el síndrome de Muir-Torre (MTS), está asociado a tumores de la piel. Si ambas copias heredadas (alelos) de un gen MMR tienen variantes genéticas dañinas, esto da lugar a una enfermedad muy rara y grave: el síndrome de cáncer por reparación de desajustes (o deficiencia constitucional de reparación de desajustes, CMMR-D), que se manifiesta como múltiples apariciones de tumores a una edad temprana, a menudo tumores de colon y cerebrales . [8]
Los cánceres esporádicos con una deficiencia de reparación del ADN rara vez tienen una mutación en un gen de reparación del ADN, pero en cambio tienden a tener alteraciones epigenéticas como la metilación del promotor que inhibe la expresión del gen de reparación del ADN. [9] Alrededor del 13% de los cánceres colorrectales son deficientes en la reparación de desajustes del ADN, comúnmente debido a la pérdida de MLH1 (9,8%), o algunas veces MSH2, MSH6 o PMS2 (todos ≤1,5%). [10] Para la mayoría de los cánceres colorrectales esporádicos deficientes en MLH1, la deficiencia se debió a la metilación del promotor de MLH1. [10] Otros tipos de cáncer tienen frecuencias más altas de pérdida de MLH1 (ver la tabla a continuación), que nuevamente son en gran medida el resultado de la metilación del promotor del gen MLH1 . Un mecanismo epigenético diferente subyacente a las deficiencias de MMR podría involucrar la sobreexpresión de un microARN, por ejemplo, los niveles de miR-155 se correlacionan inversamente con la expresión de MLH1 o MSH2 en el cáncer colorrectal. [11]
Un defecto de campo (cancerización de campo) es un área del epitelio que ha sido preacondicionada por cambios epigenéticos o genéticos, predisponiéndola al desarrollo de cáncer. Como señala Rubin "...existe evidencia de que más del 80% de las mutaciones somáticas encontradas en tumores colorrectales humanos de fenotipo mutador ocurren antes del inicio de la expansión clonal terminal". [20] [21] De manera similar, Vogelstein et al. [22] señalan que más de la mitad de las mutaciones somáticas identificadas en tumores ocurrieron en una fase preneoplásica (en un defecto de campo), durante el crecimiento de células aparentemente normales.
Las deficiencias de MLH1 eran comunes en los defectos de campo (tejidos histológicamente normales) que rodeaban a los tumores; consulte la Tabla anterior. El MLH1 epigenéticamente silenciado o mutado probablemente no conferiría una ventaja selectiva a una célula madre, sin embargo, causaría mayores tasas de mutación, y uno o más de los genes mutados pueden proporcionar a la célula una ventaja selectiva. El gen MLH1 deficiente podría entonces ser transportado como un gen pasajero selectivamente casi neutral (polizón) cuando la célula madre mutada genere un clon expandido. La presencia continua de un clon con un MLH1 epigenéticamente reprimido continuaría generando más mutaciones, algunas de las cuales podrían producir un tumor.
En un estudio que examinó a los pacientes que respondían a anti-PD1, se observó inicialmente que las mutaciones de reparación de desajustes y MMR se asociaban con la eficacia del bloqueo de puntos de control inmunológico. [23] La asociación entre la positividad de MSI y la respuesta positiva a anti-PD1 se validó posteriormente en un ensayo clínico prospectivo y fue aprobada por la FDA. [24]
En los seres humanos, siete proteínas de reparación de desajustes de ADN (MMR) ( MLH1 , MLH3 , MSH2 , MSH3 , MSH6 , PMS1 y PMS2 ) trabajan de manera coordinada en pasos secuenciales para iniciar la reparación de desajustes de ADN. [25] Además, existen subvías MMR dependientes de Exo1 e independientes de Exo1. [26]
Otros productos genéticos involucrados en la reparación de desajustes (posterior a la iniciación por los genes MMR) en humanos incluyen la ADN polimerasa delta , PCNA , RPA , HMGB1 , RFC y la ADN ligasa I , además de factores modificadores de histonas y cromatina . [27] [28]
En determinadas circunstancias, la vía MMR puede reclutar una ADN polimerasa eta propensa a errores ( POLH ). Esto sucede en los linfocitos B durante la hipermutación somática , donde la POLH se utiliza para introducir variación genética en los genes de anticuerpos. [29] Sin embargo, esta vía MMR propensa a errores puede activarse en otros tipos de células humanas tras la exposición a genotoxinas [30] y, de hecho, es ampliamente activa en varios cánceres humanos, causando mutaciones que llevan una firma de la actividad de POLH. [31]
Reconocer y reparar los desajustes y las indeles es importante para las células porque, si no lo hacen, se produce inestabilidad de microsatélites (MSI) y una tasa elevada de mutación espontánea (fenotipo mutador). En comparación con otros tipos de cáncer, el cáncer deficiente en MMR (MSI) tiene una frecuencia de mutaciones muy alta, similar al melanoma y al cáncer de pulmón, [32] tipos de cáncer causados por una gran exposición a la radiación UV y a sustancias químicas mutagénicas.
Además de una carga de mutación muy alta, las deficiencias de MMR dan lugar a una distribución inusual de mutaciones somáticas en todo el genoma humano: esto sugiere que MMR protege preferentemente las regiones eucromáticas ricas en genes y de replicación temprana. [33] Por el contrario, las regiones heterocromáticas del genoma, pobres en genes y de replicación tardía, presentan altas tasas de mutación en muchos tumores humanos. [34]
La modificación de histona H3K36me3 , una marca epigenética de la cromatina activa, tiene la capacidad de reclutar el complejo MSH2-MSH6 (hMutSα). [35] Consistentemente, las regiones del genoma humano con altos niveles de H3K36me3 acumulan menos mutaciones debido a la actividad de MMR. [31]
La falta de MMR a menudo ocurre en coordinación con la pérdida de otros genes de reparación del ADN. [9] Por ejemplo, los genes MMR MLH1 y MLH3, así como otros 11 genes de reparación del ADN (como MGMT y muchos genes de la vía NER ) se regularon a la baja de manera significativa en astrocitomas de grado inferior y de grado superior , en contraste con el tejido cerebral normal. [36] Además, la expresión de MLH1 y MGMT se correlacionó estrechamente en 135 muestras de cáncer gástrico y la pérdida de MLH1 y MGMT pareció acelerarse sincrónicamente durante la progresión del tumor. [37]
La expresión deficiente de múltiples genes de reparación del ADN se encuentra a menudo en los cánceres, [9] y puede contribuir a las miles de mutaciones que suelen encontrarse en los cánceres (ver Frecuencias de mutación en cánceres ).
Una idea popular, que no ha logrado obtener un apoyo experimental significativo, es la idea de que la mutación, a diferencia del daño del ADN, es la causa principal del envejecimiento. Los ratones defectuosos en el homólogo mutL Pms2 tienen una frecuencia de mutación aproximadamente 100 veces mayor en todos los tejidos, pero no parecen envejecer más rápidamente. [38] Estos ratones muestran un desarrollo y una vida mayormente normales, excepto por la aparición temprana de carcinogénesis e infertilidad masculina.
{{cite book}}: |journal=ignorado ( ayuda )