La visualización bacteriana (o visualización de bacterias o visualización de superficie bacteriana ) es una técnica de ingeniería de proteínas utilizada para la evolución de proteínas in vitro . Las bibliotecas de polipéptidos que se muestran en la superficie de las bacterias se pueden examinar mediante citometría de flujo o procedimientos de selección iterativos (biopanning). Esta técnica de ingeniería de proteínas nos permite vincular la función de una proteína con el gen que la codifica. La presentación bacteriana se puede utilizar para encontrar proteínas diana con propiedades deseadas y se puede utilizar para producir ligandos de afinidad que sean específicos de cada célula. Este sistema se puede utilizar en muchas aplicaciones, incluida la creación de nuevas vacunas, la identificación de sustratos enzimáticos y la búsqueda de la afinidad de un ligando por su proteína diana.
La visualización bacteriana a menudo se combina con técnicas de clasificación de células activadas magnéticamente (MACS) o de clasificación de células activadas por fluorescencia (FACS). Los métodos competitivos para la evolución de proteínas in vitro son la presentación en fagos , la presentación en ribosomas , la presentación en levaduras y la presentación en ARNm . La visualización de bacteriófagos es el tipo de sistema de visualización más común utilizado [1], aunque la visualización de bacterias se está volviendo cada vez más popular a medida que se superan los desafíos técnicos. La visualización bacteriana combinada con FACS también tiene la ventaja de que es una técnica en tiempo real.
Los sistemas de visualización celular se utilizaron por primera vez en 1985, cuando los péptidos se fusionaron genéticamente con proteínas expuestas en el bacteriófago M13 . La visualización de bacteriófagos es un sistema de visualización celular de uso común, aunque conlleva limitaciones en el tamaño de las proteínas que se pueden visualizar. Luego se introdujo la visualización bacteriana en 1986, lo que permitió la visualización en superficie de proteínas más grandes. Los sistemas de visualización bacteriana fueron introducidos por primera vez por Freudl et al. y Charbit et al. en 1986, cuando utilizaron las proteínas de superficie bacteriana OmpA y LamB para mostrar péptidos. Freudl et al. fusionaron péptidos con conectores con el gen ompA , lo que provocó que los péptidos se expresaran en las proteínas OmpA. Demostraron que las proteínas ahora estaban sujetas a escisión por la proteinasa K. Por lo tanto, los péptidos distintos de OmpA insertados eran un objetivo de la proteinasa K. La inserción de péptidos extraños no afectó el crecimiento de las células bacterianas. Charbit et al. En primer lugar, se definieron las áreas de la proteína LamB que eran "permisivas" para la inserción de péptidos extraños ( es decir, que no conducían a una pérdida completa de funcionalidad de la proteína). Luego, exploraron la versatilidad de los sitios permisivos (límite de tamaño, naturaleza del epítopo,...) que estaban todos ubicados en bucles expuestos en la superficie de la porina de la membrana externa trimérica, con el objetivo de desarrollar vacunas bacterianas vivas multivalentes (12-15 ). Esta fue la primera evidencia del uso de técnicas de visualización de superficies bacterianas para expresar proteínas en la superficie de las células, sin alterar la función de la célula. [2]
Los péptidos son muy útiles como sustancias terapéuticas y de diagnóstico. Su uso es cada vez más popular y los sistemas de visualización ofrecen una forma útil de diseñar péptidos y optimizar sus capacidades de unión. Las células expresan proteínas de superficie que pueden participar en una gran cantidad de respuestas, incluido el reconocimiento de otras células, la interacción con otras células y la señalización celular . Muchos tipos de bacterias tienen proteínas de la superficie celular, como la proteína intimina enteropatógena de E. coli , que participa en la unión a las células huésped, o la proteína OmpA de las células de E. coli , que es importante para mantener la estructura de la membrana externa . [3] Muchas proteínas de superficie participan en la unión de las células bacterianas y la invasión de la célula huésped. Mediante el uso de visualización bacteriana, se pueden identificar proteínas diana en la célula huésped. Estas proteínas de superficie primero deben translocarse a través de las membranas celulares bacterianas desde el citoplasma hasta la superficie celular. Las bacterias Gram negativas tienen un espacio periplásmico adicional , del que carecen las bacterias Gram positivas , por lo que tienen una tarea más difícil de translocar proteínas. La visualización de proteínas heterólogas en la superficie de la célula bacteriana normalmente requiere la fusión de la proteína con una proteína de superficie, llamada andamio.
Se utilizan andamios para mostrar la proteína heteróloga en la superficie de la célula bacteriana. Se han utilizado varios andamios, como proteínas de la membrana externa, proteínas de fimbrias/flagelos y CPX (OmpX permutado circularmente). [4] El andamio CPX permite la fusión de péptidos en ambos extremos del andamio.
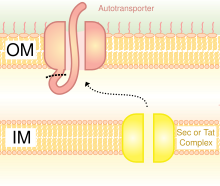
Las OMP son andamios comunes para la visualización de bacterias. Las proteínas también se pueden exhibir en la superficie de las células bacterianas mediante el uso de autotransportadores. Los autotransportadores forman parte del sistema de secreción tipo V. Suelen tener tres dominios: secuencia líder en el N-terminal; dominio central de pasajeros; dominio autotransportador en el C-terminal. La proteína heteróloga se inserta en el dominio pasajero. [5] Otro método de fusión de proteínas heterólogas es la fusión con fimbrias / flagelos , que son protuberancias filamentosas en la superficie celular. Hay muchas fimbrias principalmente en bacterias Gram negativas, por lo que mostrar proteínas en las fimbrias es ventajoso sobre otras proteínas de superficie que son menos numerosas. Una desventaja de utilizar fimbrias es que existe un límite de tamaño de inserción relativamente pequeño de 10 a 30 aminoácidos . [6]

Una vez que la proteína heteróloga se ha fusionado con la proteína de la superficie celular bacteriana, se expone a una enzima , una célula (que expresa una proteína diana) o un anticuerpo (generalmente marcado con fluorescencia ), según la aplicación del experimento. Luego, la muestra pasa a través de un haz de luz durante FACS, en una corriente de líquido muy estrecha para que solo pueda pasar una célula a la vez, y se detecta la fluorescencia emitida. Se puede obtener información sobre el tamaño de la célula mediante la dispersión de la luz y si se ha producido la unión de la proteína heteróloga con la proteína/célula diana, se emitirá más fluorescencia.
La visualización de superficie bacteriana se puede utilizar para una variedad de aplicaciones. Estos incluyen la detección basada en afinidad, el mapeo de epítopos de anticuerpos , la identificación de sustratos peptídicos, la identificación de péptidos de unión a células y la generación de vacunas. [7]
El cribado se utiliza para encontrar las afinidades aparentes de proteínas heterólogas expuestas en la superficie de la célula bacteriana por proteínas diana. Este método generalmente se combina con FACS, y la adición de una proteína diana competidora no fluorescente es beneficiosa para obtener afinidades de unión más precisas. Agregar un competidor reduce la posibilidad de que las proteínas objetivo se vuelvan a unir, lo que haría que la afinidad de unión sea menos precisa.
Los péptidos cíclicos se pueden exhibir con éxito en la superficie de las células bacterianas. [8] Mediante la aleatorización del ADN, se pueden analizar millones de péptidos cíclicos mostrados en la superficie celular frente a una proteína diana utilizando FACS de alto rendimiento. [9]
El mapeo de epítopos de anticuerpos se utiliza para encontrar la especificidad de un anticuerpo. El epítopo (sitio de unión de anticuerpos a antígenos) se expresa en la superficie de la célula bacteriana mediante la expresión de una región del gen que codifica el antígeno. La citometría de flujo con anticuerpos marcados con fluorescencia se utiliza para detectar la cantidad de anticuerpo que se une al epítopo. [10]
Esto se puede aplicar para encontrar los mejores sustratos para las enzimas proteolíticas . El sustrato se muestra en la superficie de la célula bacteriana entre un ligando de afinidad y el andamio, y la cinética de la proteólisis del sustrato se mide usando FACS.
La presentación bacteriana se puede utilizar para encontrar péptidos que se unan a células específicas, por ejemplo, células de cáncer de mama o células madre . Las proteínas mostradas están marcadas con fluorescencia con GFP , por lo que las interacciones de unión entre los péptidos y las células diana pueden verse mediante citometría de flujo. Se requieren muestras de control para medir los niveles de fluorescencia en ausencia de péptidos mostrados. También se requieren muestras que no contengan péptidos mostrados, pero que contengan células de mamíferos y células bacterianas (incluido el andamio).
La administración de vacunas es una aplicación muy común de visualización de superficies bacterianas. Hay dos tipos de vacunas bacterianas vivas que se pueden elaborar:
El uso de la presentación de antígenos en la superficie bacteriana es una alternativa valiosa al diseño de vacunas convencionales por varias razones, una de ellas es que las proteínas expresadas en la superficie de las células bacterianas pueden actuar favorablemente como adyuvante . Las vacunas convencionales requieren la adición de adyuvantes. Otra ventaja de generar vacunas utilizando sistemas de presentación bacteriana es que se puede incorporar toda la célula bacteriana en la vacuna viva [11]. A diferencia de los sistemas de presentación en bacteriófagos que generalmente se usan en el desarrollo de vacunas para encontrar epítopos desconocidos, los sistemas de presentación en bacterias se usan para expresar epítopos conocidos. y las células actúan como un sistema de administración de vacunas. [12]
En condiciones similares, la selección de péptidos mostrados en bacterias para modelar la proteína estreptavidina resultó peor. [13]
{{cite book}}: |journal=ignorado ( ayuda )12. Charbit A, Boulain JC, Ryter A, Hofnung M. Sondeo de la topología de una proteína de membrana bacteriana mediante la inserción genética de un epítopo extraño; expresión en la superficie celular. EMBO J. 1986 de noviembre;5(11):3029-37.
13. Charbit A, Sobczak E, Michel ML, Molla A, Tiollais P, Hofnung M. Presentación de dos epítopos de la región preS2 del virus de la hepatitis B en bacterias recombinantes vivas. J Inmunol. 1 de septiembre de 1987; 139(5):1658-64.
14. Charbit A, Molla A, Saurin W, Hofnung M.Versatilidad de un vector para expresar polipéptidos extraños en la superficie de bacterias gramnegativas. Gene. 15 de octubre de 1988; 70 (1): 181-9.
15. Newton SM, Klebba PE, Michel V, Hofnung M, Charbit A. Topología de la proteína de membrana LamB mediante etiquetado de epítopos y comparación con el modelo de rayos X. J Bacteriol. Junio de 1996; 178(12):3447-56.