En biología evolutiva y genética de poblaciones , el umbral de error (o tasa crítica de mutación ) es un límite en la cantidad de pares de bases que una molécula autorreplicante puede tener antes de que la mutación destruya la información en las generaciones posteriores de la molécula. El umbral de error es crucial para comprender la "paradoja de Eigen".
El umbral de error es un concepto que se utiliza en los orígenes de la vida ( abiogénesis ), en particular en las primeras etapas de la vida, antes de la aparición del ADN . Se postula que las primeras moléculas autorreplicantes podrían haber sido pequeñas moléculas de ARN similares a las ribozimas . Estas moléculas consisten en cadenas de pares de bases o "dígitos", y su orden es un código que dirige cómo interactúa la molécula con su entorno. Toda replicación está sujeta a errores de mutación. Durante el proceso de replicación, cada dígito tiene una cierta probabilidad de ser reemplazado por algún otro dígito, lo que cambia la forma en que la molécula interactúa con su entorno y puede aumentar o disminuir su aptitud, o capacidad para reproducirse, en ese entorno.
En su artículo de 1971 (Eigen 1971), Manfred Eigen observó que este proceso de mutación impone un límite al número de dígitos que puede tener una molécula. Si una molécula supera este tamaño crítico, el efecto de las mutaciones se vuelve abrumador y un proceso de mutación descontrolado destruirá la información en las generaciones posteriores de la molécula. El umbral de error también está controlado por el "paisaje de aptitud" de las moléculas. El paisaje de aptitud se caracteriza por los dos conceptos de altura (=aptitud) y distancia (=número de mutaciones). Las moléculas similares están "cerca" unas de otras, y las moléculas que están más en forma que otras y tienen más probabilidades de reproducirse, están "más arriba" en el paisaje.
Si una secuencia particular y sus vecinas tienen una alta aptitud, formarán una cuasiespecie y podrán soportar longitudes de secuencia más largas que una secuencia apta con pocas vecinas aptas, o un vecindario de secuencias menos apto. Además, Wilke (Wilke 2005) observó que el concepto de umbral de error no se aplica en partes del paisaje donde hay mutaciones letales, en las que la mutación inducida produce una aptitud cero e impide que la molécula se reproduzca.
La paradoja de Eigen es uno de los enigmas más insolubles en el estudio de los orígenes de la vida. Se cree que el concepto de umbral de error descrito anteriormente limita el tamaño de las moléculas autorreplicantes a quizás unos pocos cientos de dígitos, pero casi toda la vida en la Tierra requiere moléculas mucho más largas para codificar su información genética. Este problema se soluciona en las células vivas mediante enzimas que reparan las mutaciones, lo que permite que las moléculas codificadoras alcancen tamaños del orden de millones de pares de bases. Estas grandes moléculas deben, por supuesto, codificar las mismas enzimas que las reparan, y en esto radica la paradoja de Eigen, planteada por primera vez por Manfred Eigen en su artículo de 1971 (Eigen 1971). [1] En pocas palabras, la paradoja de Eigen equivale a lo siguiente:
Se trata de una paradoja del tipo del huevo y la gallina , con una solución aún más difícil. ¿Qué fue primero, el genoma grande o las enzimas correctoras de errores? Se han propuesto varias soluciones a esta paradoja:
Consideremos una molécula de 3 dígitos [A,B,C] donde A, B y C pueden tomar los valores 0 y 1. Hay ocho secuencias de este tipo ([000], [001], [010], [011], [100], [101], [110] y [111]). Digamos que la molécula [000] es la más adecuada; en cada replicación produce un promedio de copias, donde . Esta molécula se llama "secuencia maestra". Las otras siete secuencias son menos adecuadas; cada una produce solo 1 copia por replicación. La replicación de cada uno de los tres dígitos se realiza con una tasa de mutación de μ. En otras palabras, en cada replicación de un dígito de una secuencia, existe una probabilidad de que sea erróneo; 0 será reemplazado por 1 o viceversa. Ignoremos las mutaciones dobles y la muerte de moléculas (la población crecerá infinitamente) y dividamos las ocho moléculas en tres clases dependiendo de su distancia de Hamming respecto de la secuencia maestra:
Obsérvese que el número de secuencias para la distancia d es simplemente el coeficiente binomial para L=3, y que cada secuencia puede visualizarse como el vértice de un cubo de dimensión L=3, donde cada borde del cubo especifica una ruta de mutación en la que el cambio de la distancia de Hamming es cero o ±1. Se puede ver que, por ejemplo, un tercio de las mutaciones de las moléculas [001] producirán moléculas [000], mientras que los otros dos tercios producirán las moléculas de clase 2 [011] y [101]. Ahora podemos escribir la expresión para las poblaciones hijas de la clase i en términos de las poblaciones progenitoras .
donde la matriz ' w ' que incorpora la selección natural y la mutación, según el modelo de cuasiespecies , viene dada por:
donde es la probabilidad de que una molécula entera se replique con éxito. Los vectores propios de la matriz w producirán los números de población de equilibrio para cada clase. Por ejemplo, si la tasa de mutación μ es cero, tendremos Q=1, y las concentraciones de equilibrio serán . La secuencia maestra, al ser la más apta, será la única que sobrevivirá. Si tenemos una fidelidad de replicación de Q=0,95 y una ventaja genética de a=1,05, entonces las concentraciones de equilibrio serán aproximadamente . Se puede ver que la secuencia maestra no es tan dominante; sin embargo, las secuencias con baja distancia de Hamming son mayoría. Si tenemos una fidelidad de replicación de Q que se acerca a 0, entonces las concentraciones de equilibrio serán aproximadamente . Esta es una población con igual número de cada una de las 8 secuencias. (Si tuviéramos una población perfectamente igual de todas las secuencias, tendríamos poblaciones de [1,3,3,1]/8).
Si ahora vamos al caso donde el número de pares de bases es grande, digamos L=100, obtenemos un comportamiento que se asemeja a una transición de fase . El gráfico de abajo a la izquierda muestra una serie de concentraciones de equilibrio divididas por el coeficiente binomial . (Esta multiplicación mostrará la población para una secuencia individual a esa distancia y producirá una línea plana para una distribución igual). La ventaja selectiva de la secuencia maestra se establece en a=1,05. El eje horizontal es la distancia de Hamming d . Las diversas curvas son para varias tasas de mutación totales . Se ve que para valores bajos de la tasa de mutación total, la población consiste en una cuasiespecie reunida en la vecindad de la secuencia maestra. Por encima de una tasa de mutación total de aproximadamente 1-Q=0,05, la distribución se extiende rápidamente para poblar todas las secuencias por igual. El gráfico de abajo a la derecha muestra la población fraccionaria de la secuencia maestra en función de la tasa de mutación total. Nuevamente se observa que por debajo de una tasa de mutación crítica de aproximadamente 1-Q=0,05, la secuencia maestra contiene la mayor parte de la población, mientras que por encima de esta tasa, contiene solo alrededor del 50% de la población total.
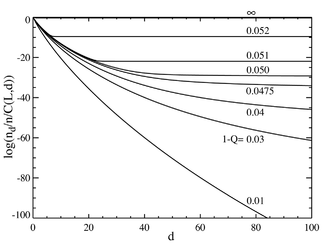

Se puede observar que hay una transición abrupta en un valor de 1-Q apenas mayor que 0,05. Para tasas de mutación superiores a este valor, la población de la secuencia maestra cae prácticamente a cero. Por encima de este valor, domina.
En el límite, cuando L se acerca al infinito, el sistema tiene de hecho una transición de fase en un valor crítico de Q: . Se podría pensar en la tasa de mutación general (1-Q) como una especie de "temperatura", que "funde" la fidelidad de las secuencias moleculares por encima de la "temperatura" crítica de . Para que se produzca una replicación fiel, la información debe estar "congelada" en el genoma.