La piruvato deshidrogenasa es una enzima que cataliza la reacción del piruvato y una lipoamida para dar lugar a la dihidrolipoamida acetilada y dióxido de carbono . La conversión requiere la coenzima pirofosfato de tiamina .
La piruvato deshidrogenasa se encuentra generalmente como un componente, denominado E1, del complejo de piruvato deshidrogenasa (PDC). La PDC consta de otras enzimas, denominadas E2 y E3. En conjunto, E1-E3 transforman el piruvato , NAD + y coenzima A en acetil-CoA , CO2 y NADH. La conversión es crucial porque el acetil-CoA puede luego usarse en el ciclo del ácido cítrico para llevar a cabo la respiración celular . [2] Para distinguir entre esta enzima y la PDC, se la denomina sistemáticamente piruvato deshidrogenasa (acetiltransferasa) .

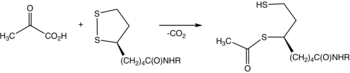
El pirofosfato de tiamina (TPP) se convierte en un iluro por desprotonación. El iluro ataca el grupo cetona del piruvato. El aducto resultante se descarboxila . El 1,3-dipolo resultante acetila reductivamente la lipoamida-E2. [2]
En términos de detalles, los datos bioquímicos y estructurales de E1 revelaron un mecanismo de activación de la coenzima TPP mediante la formación de un enlace de hidrógeno conservado con el residuo de glutamato (Glu59 en E1 humana) y mediante la imposición de una conformación V que lleva al átomo N4' de la aminopirimidina a un enlace de hidrógeno intramolecular con el átomo C2 de tiazolio. Esta combinación única de contactos y conformaciones de TPP conduce a la formación del carbanión C2 reactivo, finalmente. Después de que el cofactor TPP descarboxila el piruvato, la porción acetilada se convierte en un derivado hidroxietilado unido covalentemente a TPP. [1]
La E1 es una proteína multimérica. Las E1 de los mamíferos, incluida la E1 humana, son tetraméricas y están compuestas por dos subunidades α y dos β. [1] Algunas E1 bacterianas, incluida la E1 de Escherichia coli , están compuestas por dos subunidades similares, cada una de las cuales es tan grande como la suma de las masas moleculares de las subunidades α y β. [3]


La E1 tiene dos sitios catalíticos, cada uno de los cuales proporciona pirofosfato de tiamina ( TPP ) e iones de magnesio como cofactores. La subunidad α se une al ion de magnesio y al fragmento de pirofosfato, mientras que la subunidad β se une al fragmento de pirimidina del TPP , formando juntos un sitio catalítico en la interfaz de las subunidades. [1]
El sitio activo de la piruvato deshidrogenasa (imagen creada a partir de PDB : 1NI4 ) retiene el TPP a través de la ligadura de un metal a un ion magnesio (esfera violeta) y a través de la unión de hidrógeno a los aminoácidos. Si bien se pueden encontrar más de 20 aminoácidos en el sitio activo, los aminoácidos Tyr 89, Arg 90, Gly 136, Val 138, Asp 167, Gly 168, Ala 169, Asn 196 e His 263 participan en la unión de hidrógeno para retener el TPP y el piruvato (no se muestra aquí) en el sitio activo. Los aminoácidos se muestran como alambres y el TPP está en forma de bolas y barras. El sitio activo también ayuda en la transferencia del acilo en el TPP a una lipoamida que espera en E2. [1]
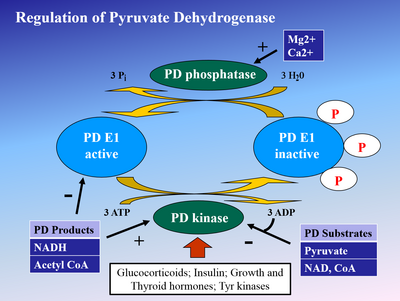
La fosforilación de E1 por la piruvato deshidrogenasa quinasa (PDK) inactiva E1 y posteriormente todo el complejo. La PDK es inhibida por el ácido dicloroacético y el piruvato , lo que da como resultado una mayor cantidad de PDH activa no fosforilada. [4] La fosforilación es revertida por la piruvato deshidrogenasa fosfatasa , que es estimulada por insulina , PEP y AMP , pero inhibida competitivamente por ATP , NADH y acetil-CoA .
La piruvato deshidrogenasa es atacada por un autoantígeno conocido como anticuerpos antimitocondriales (AMA), que produce una destrucción progresiva de los pequeños conductos biliares del hígado, lo que lleva a una cirrosis biliar primaria . Estos anticuerpos parecen reconocer la proteína oxidada que ha resultado de respuestas inmunitarias inflamatorias. Algunas de estas respuestas inflamatorias podrían estar relacionadas con la sensibilidad al gluten , ya que más del 50% de los pacientes con insuficiencia hepática aguda en un estudio mostraron un autoanticuerpo no mitocondrial contra la transglutaminasa tisular . [5] Otros autoantígenos mitocondriales incluyen la oxoglutarato deshidrogenasa y el complejo de alfa-cetoácido deshidrogenasa de cadena ramificada , que son antígenos reconocidos por anticuerpos antimitocondriales .
El aumento de la actividad de la piruvato deshidrogenasa (PDH) puede causar senescencia celular inducida por oncogenes , además de promover el envejecimiento. [6] Se ha demostrado una disminución de la actividad de la PDH mitocondrial con la edad en el corazón, así como en ciertas regiones del cerebro (el cuerpo estriado y el tronco encefálico ). [6]
La deficiencia de piruvato deshidrogenasa (PDH) es una enfermedad metabólica degenerativa congénita que resulta de una mutación del complejo de piruvato deshidrogenasa (PDC) ubicado en el cromosoma X. Si bien se han identificado defectos en las 3 enzimas del complejo, la subunidad E1-α es la principal culpable. El mal funcionamiento del ciclo del ácido cítrico debido a la deficiencia de PDH priva al cuerpo de energía y conduce a una acumulación anormal de lactato. La deficiencia de PDH es una causa común de acidosis láctica en recién nacidos y a menudo se presenta con letargo grave, mala alimentación, taquipnea y se han producido casos de muerte. [7]
Las proteínas humanas que poseen actividad de piruvato deshidrogenasa incluyen:
En las bacterias , existe una forma de piruvato deshidrogenasa (también llamada piruvato oxidasa, EC 1.2.2.2) que vincula la oxidación del piruvato en acetato y dióxido de carbono con la reducción del ferrocitocromo. En E. coli, esta enzima está codificada por el gen pox B y la proteína tiene un cofactor flavina. [8] Esta enzima aumenta la eficiencia del crecimiento de E. coli en condiciones aeróbicas. [9]
{{cite journal}}: CS1 maint: nombres numéricos: lista de autores ( enlace ){{cite book}}: |journal=ignorado ( ayuda )