La pirosecuenciación es un método de secuenciación del ADN (determinación del orden de los nucleótidos en el ADN) basado en el principio de "secuenciación por síntesis", en el que la secuenciación se realiza detectando el nucleótido incorporado por una ADN polimerasa . La pirosecuenciación se basa en la detección de luz basada en una reacción en cadena cuando se libera pirofosfato . De ahí el nombre de pirosecuenciación.
El principio de pirosecuenciación fue descrito por primera vez en 1993 [1] por Bertil Pettersson, Mathias Uhlen y Pål Nyren combinando el método de secuenciación en fase sólida [2] utilizando perlas magnéticas recubiertas de estreptavidina con ADN polimerasa recombinante que carece de actividad exonucleasa de 3' a 5' ( corrección de pruebas) y detección de luminiscencia utilizando la enzima luciferasa de luciérnaga . [3] Se añade una mezcla de tres enzimas ( ADN polimerasa , ATP sulfurilasa y luciferasa de luciérnaga ) y un nucleótido ( dNTP ) al ADN monocatenario que se va a secuenciar y la incorporación del nucleótido se sigue midiendo la luz emitida. La intensidad de la luz determina si se han incorporado 0, 1 o más nucleótidos, mostrando así cuántos nucleótidos complementarios están presentes en la cadena plantilla. La mezcla de nucleótidos se elimina antes de añadir la siguiente mezcla de nucleótidos. Este proceso se repite con cada uno de los cuatro nucleótidos hasta que se determina la secuencia de ADN del molde monocatenario.
Un segundo método basado en soluciones para pirosecuenciación fue descrito en 1998 [4] por Mostafa Ronaghi , Mathias Uhlen y Pål Nyren . En este método alternativo, se introduce una enzima apirasa adicional para eliminar los nucleótidos que no están incorporados por la ADN polimerasa. Esto permitió agregar la mezcla de enzimas que incluía la ADN polimerasa , la luciferasa y la apirasa al inicio y mantenerla durante todo el procedimiento, proporcionando así una configuración simple adecuada para la automatización. Al año siguiente, la empresa Pyrosequencing introdujo en el mercado un instrumento automatizado basado en este principio.
Una tercera variante microfluídica del método de pirosecuenciación fue descrita en 2005 [5] por Jonathan Rothberg y sus colaboradores de la empresa 454 Life Sciences . Este enfoque alternativo para la pirosecuenciación se basó en el principio original de unir el ADN que se va a secuenciar a un soporte sólido y demostraron que la secuenciación se podía realizar de una manera muy paralela utilizando una micromatriz microfabricada . Esto permitió la secuenciación de ADN de alto rendimiento y se introdujo en el mercado un instrumento automatizado. Este se convirtió en el primer instrumento de secuenciación de próxima generación que inició una nueva era en la investigación genómica , con una rápida caída de los precios de la secuenciación del ADN, lo que permitió secuenciar el genoma completo a precios asequibles.
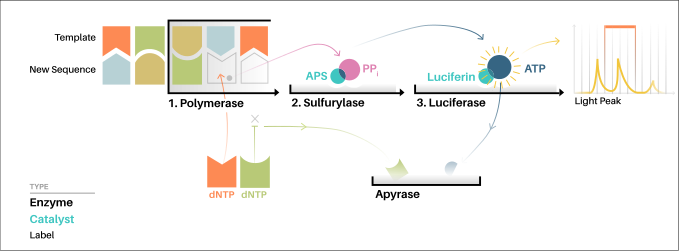
La "secuenciación por síntesis" implica tomar una sola hebra del ADN a secuenciar y luego sintetizar enzimáticamente su hebra complementaria. El método de pirosecuenciación se basa en detectar la actividad de la ADN polimerasa (una enzima sintetizadora de ADN) con otra enzima quimioluminiscente . Básicamente, el método permite secuenciar una sola cadena de ADN sintetizando la cadena complementaria a lo largo de ella, un par de bases a la vez, y detectando qué base se agregó realmente en cada paso. El ADN molde está inmóvil y se añaden y eliminan secuencialmente soluciones de nucleótidos A, C, G y T de la reacción. La luz se produce sólo cuando la solución de nucleótidos complementa la primera base desapareada de la plantilla. La secuencia de soluciones que producen señales quimioluminiscentes permite determinar la secuencia de la plantilla. [6]
Para la versión de pirosecuenciación basada en solución, la plantilla de ADN monocatenario ( ADNss ) se hibrida con un cebador de secuenciación y se incuba con las enzimas ADN polimerasa , ATP sulfurilasa , luciferasa y apirasa , y con los sustratos adenosina 5´fosfosulfato (APS). y luciferina .
El proceso se puede representar mediante las siguientes ecuaciones:
dónde:
Actualmente, una limitación del método es que las longitudes de las lecturas individuales de la secuencia de ADN son del orden de 300 a 500 nucleótidos, más cortas que los 800 a 1000 que se pueden obtener con métodos de terminación de cadena (por ejemplo, secuenciación de Sanger). Esto puede dificultar el proceso de ensamblaje del genoma , particularmente para secuencias que contienen una gran cantidad de ADN repetitivo . La falta de actividad de corrección de pruebas limita la precisión de este método.
La empresa Pyrosequencing AB en Uppsala, Suecia, se fundó con capital riesgo proporcionado por HealthCap con el fin de comercializar maquinaria y reactivos para secuenciar tramos cortos de ADN mediante la técnica de pirosecuenciación. Pyrosequencing AB cotizó en la Bolsa de Valores de Estocolmo en 1999. Pasó a llamarse Biotage en 2003. [7] La línea de negocio de pirosecuenciación fue adquirida por Qiagen en 2008. La tecnología de pirosecuenciación obtuvo además licencia para 454 Life Sciences . 454 desarrolló una tecnología de pirosecuenciación basada en matrices que surgió como una plataforma para la secuenciación de ADN a gran escala , incluida la secuenciación del genoma y la metagenómica .
Roche anunció la discontinuación de la plataforma de secuenciación 454 en 2013. [8]