La predicción de interacciones proteína-proteína es un campo que combina la bioinformática y la biología estructural en un intento de identificar y catalogar interacciones físicas entre pares o grupos de proteínas. Comprender las interacciones proteína-proteína es importante para la investigación de las vías de señalización intracelular, el modelado de estructuras de complejos proteicos y para obtener información sobre diversos procesos bioquímicos.
Experimentalmente , las interacciones físicas entre pares de proteínas se pueden inferir a partir de una variedad de técnicas, incluidos los sistemas de dos híbridos de levadura, los ensayos de complementación de fragmentos de proteína (PCA), la purificación por afinidad/ espectrometría de masas , los microarreglos de proteínas , la transferencia de energía por resonancia de fluorescencia (FRET) y la termoforesis a microescala (MST). Se están realizando esfuerzos para determinar experimentalmente el interactoma de numerosas especies. Las interacciones determinadas experimentalmente suelen proporcionar la base para los métodos computacionales para predecir interacciones, por ejemplo, utilizando secuencias de proteínas homólogas en todas las especies. Sin embargo, también hay métodos que predicen interacciones de novo , sin conocimiento previo de las interacciones existentes.
Las proteínas que interactúan tienen más probabilidades de coevolucionar, [1] [2] [3] [4] por lo tanto, es posible hacer inferencias sobre las interacciones entre pares de proteínas en función de sus distancias filogenéticas. También se ha observado en algunos casos que pares de proteínas interactuantes tienen ortólogos fusionados en otros organismos. Además, se han resuelto estructuralmente varios complejos proteicos unidos y se pueden utilizar para identificar los residuos que median la interacción de modo que se puedan localizar motivos similares en otros organismos.

El método del perfil filogenético se basa en la hipótesis de que si dos o más proteínas están presentes o ausentes simultáneamente en varios genomas, es probable que estén funcionalmente relacionadas. [5] La Figura A ilustra una situación hipotética en la que las proteínas A y B se identifican como funcionalmente vinculadas debido a sus perfiles filogenéticos idénticos en 5 genomas diferentes. El Joint Genome Institute ofrece una base de datos integrada de genomas y microbiomas microbianos (JGI IMG) que tiene una herramienta de creación de perfiles filogenéticos para genes individuales y casetes de genes.
Se observó que los árboles filogenéticos de ligandos y receptores eran a menudo más similares que debido al azar. [4] Esto es probable porque enfrentaron presiones de selección similares y coevolucionaron. Este método [6] utiliza los árboles filogenéticos de pares de proteínas para determinar si existen interacciones. Para ello, se encuentran homólogos de las proteínas de interés (utilizando una herramienta de búsqueda de secuencias como BLAST ) y se realizan alineaciones de múltiples secuencias (con herramientas de alineamiento como Clustal ) para construir matrices de distancia para cada una de las proteínas de interés. [4] Las matrices de distancia deberían usarse luego para construir árboles filogenéticos. Sin embargo, las comparaciones entre árboles filogenéticos son difíciles, y los métodos actuales evitan esto simplemente comparando matrices de distancia [4] . Las matrices de distancia de las proteínas se utilizan para calcular un coeficiente de correlación, en el que un valor mayor corresponde a la coevolución. El beneficio de comparar matrices de distancia en lugar de árboles filogenéticos es que los resultados no dependen del método de construcción de árboles que se utilizó. La desventaja es que las matrices de diferencia no son representaciones perfectas de los árboles filogenéticos, y pueden resultar imprecisiones al usar este atajo. [4] Otro factor digno de mención es que existen similitudes de fondo entre los árboles filogenéticos de cualquier proteína, incluso las que no interactúan. Si no se tienen en cuenta, esto podría conducir a una alta tasa de falsos positivos. Por esta razón, ciertos métodos construyen un árbol de fondo utilizando secuencias de ARNr 16S que utilizan como el árbol de la vida canónico. La matriz de distancia construida a partir de este árbol de la vida luego se resta de las matrices de distancia de las proteínas de interés. [7] Sin embargo, debido a que las matrices de distancia de ARN y las matrices de distancia de ADN tienen una escala diferente, presumiblemente porque el ARN y el ADN tienen diferentes tasas de mutación, la matriz de ARN necesita ser reescalada antes de que pueda ser restada de las matrices de ADN. [7] Al usar proteínas de reloj molecular, se puede calcular el coeficiente de escala para la distancia de la proteína/distancia del ARN. [7] Este coeficiente se utiliza para reescalar la matriz de ARN.

El método de fusión de dominios o de piedra de Rosetta se basa en la hipótesis de que las proteínas que interactúan a veces se fusionan en una sola proteína. [3] Por ejemplo, dos o más proteínas separadas en un genoma pueden identificarse como fusionadas en una sola proteína en otro genoma. Es probable que las proteínas separadas interactúen y, por lo tanto, es probable que estén relacionadas funcionalmente. Un ejemplo de esto es la enzima humana Succinyl coA Transferase , que se encuentra como una proteína en los humanos pero como dos proteínas separadas, Acetate coA Transferase alpha y Acetate coA Transferase beta , en Escherichia coli . [3] Para identificar estas secuencias, es necesario un algoritmo de similitud de secuencia como el utilizado por BLAST . Por ejemplo, si tuviéramos las secuencias de aminoácidos de las proteínas A y B y las secuencias de aminoácidos de todas las proteínas en un genoma determinado, podríamos verificar cada proteína en ese genoma en busca de regiones no superpuestas de similitud de secuencia con ambas proteínas A y B. La Figura B representa la alineación de secuencia BLAST de Succinyl coA Transferase con sus dos homólogos separados en E. coli. Las dos subunidades tienen regiones no superpuestas de similitud de secuencia con la proteína humana, indicadas por las regiones rosas, siendo la subunidad alfa similar a la primera mitad de la proteína y la beta similar a la segunda mitad. Una limitación de este método es que no todas las proteínas que interactúan se pueden encontrar fusionadas en otro genoma y, por lo tanto, no se pueden identificar mediante este método. Por otro lado, la fusión de dos proteínas no necesita que interactúen físicamente. Por ejemplo, se sabe que los dominios SH2 y SH3 en la proteína src interactúan. Sin embargo, muchas proteínas poseen homólogos de estos dominios y no todas interactúan. [3]
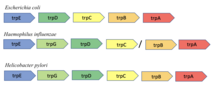
El método de vecindad conservada se basa en la hipótesis de que si los genes que codifican dos proteínas son vecinos en un cromosoma en muchos genomas, entonces es probable que estén funcionalmente relacionados. El método se basa en una observación de Bork et al. de la conservación de pares de genes en nueve genomas bacterianos y arqueológicos. El método es más eficaz en procariotas con operones, ya que la organización de los genes en un operón generalmente está relacionada con la función. [8] Por ejemplo, los genes trpA y trpB en Escherichia coli codifican las dos subunidades de la enzima triptófano sintasa que se sabe que interactúan para catalizar una sola reacción. Se demostró que la adyacencia de estos dos genes se conserva en nueve genomas bacterianos y arqueológicos diferentes. [8]
Los métodos de clasificación utilizan datos para entrenar un programa (clasificador) para distinguir ejemplos positivos de pares de proteínas/dominios interactuantes con ejemplos negativos de pares que no interactúan. Los clasificadores populares utilizados son Random Forest Decision (RFD) y Support Vector Machines. RFD produce resultados basados en la composición del dominio de pares de proteínas interactuantes y no interactuantes. Cuando se le da un par de proteínas para clasificar, RFD primero crea una representación del par de proteínas en un vector. [9] El vector contiene todos los tipos de dominio utilizados para entrenar RFD, y para cada tipo de dominio el vector también contiene un valor de 0, 1 o 2. Si el par de proteínas no contiene un cierto dominio, entonces el valor para ese dominio es 0. Si una de las proteínas del par contiene el dominio, entonces el valor es 1. Si ambas proteínas contienen el dominio, entonces el valor es 2. [9] Usando datos de entrenamiento, RFD construye un bosque de decisiones, que consiste en muchos árboles de decisiones. Cada árbol de decisión evalúa varios dominios y, en función de la presencia o ausencia de interacciones en estos dominios, toma una decisión sobre si el par de proteínas interactúa. La representación vectorial del par de proteínas es evaluada por cada árbol para determinar si son un par interactuante o un par que no interactúa. El bosque cuenta toda la entrada de los árboles para llegar a una decisión final. [9] La fortaleza de este método es que no asume que los dominios interactúan independientemente unos de otros. Esto hace que se puedan usar múltiples dominios en proteínas en la predicción. [9] Este es un gran avance con respecto a los métodos anteriores que solo podían predecir basándose en un solo par de dominios. La limitación de este método es que se basa en el conjunto de datos de entrenamiento para producir resultados. Por lo tanto, el uso de diferentes conjuntos de datos de entrenamiento podría influir en los resultados. Una advertencia de la mayoría de los métodos es la falta de datos negativos, por ejemplo, no interacciones para proteínas que se pueden superar utilizando un muestreo negativo impulsado por la topología. [10]
Este grupo de métodos [11] [9] [12] [13] [14] hace uso de estructuras de complejos proteicos conocidos para predecir y modelar estructuralmente las interacciones entre secuencias de proteínas de consulta. El proceso de predicción generalmente comienza empleando un método basado en secuencias (por ejemplo, Interolog ) para buscar estructuras de complejos proteicos que sean homólogas a las secuencias de consulta. Estas estructuras complejas conocidas se utilizan luego como plantillas para modelar estructuralmente la interacción entre secuencias de consulta. Este método tiene la ventaja de no solo inferir interacciones proteicas, sino que también sugiere modelos de cómo las proteínas interactúan estructuralmente, lo que puede proporcionar algunas ideas sobre el mecanismo de nivel atómico de esa interacción. Por otro lado, la capacidad de estos métodos para hacer una predicción está restringida por un número limitado de estructuras de complejos proteicos conocidas.
Los métodos de asociación buscan secuencias o motivos característicos que puedan ayudar a distinguir entre pares interactuantes y no interactuantes. Un clasificador se entrena buscando pares de firmas de secuencia donde una proteína contiene una firma de secuencia y su pareja interactuante contiene otra firma de secuencia. [15] Buscan específicamente firmas de secuencia que se encuentran juntas con más frecuencia que por casualidad. Esto utiliza una puntuación de probabilidades logarítmicas que se calcula como log2(Pij/PiPj), donde Pij es la frecuencia observada de los dominios i y j que aparecen en un par de proteínas; Pi y Pj son las frecuencias de fondo de los dominios i y j en los datos. Las interacciones de dominios predichas son aquellas con puntuaciones de probabilidades logarítmicas positivas y que también tienen varias ocurrencias dentro de la base de datos. [15] La desventaja de este método es que analiza cada par de dominios interactuantes por separado y asume que interactúan independientemente uno del otro.
Este método [16] [17] construye una biblioteca de interfaces proteína-proteína conocidas a partir del PDB , donde las interfaces se definen como pares de fragmentos de polipéptidos que están por debajo de un umbral ligeramente mayor que el radio de Van der Waals de los átomos involucrados. Las secuencias en la biblioteca se agrupan en función de la alineación estructural y se eliminan las secuencias redundantes. Los residuos que tienen un alto nivel de frecuencia (generalmente >50%) para una posición dada se consideran puntos calientes. [18] Esta biblioteca se utiliza luego para identificar interacciones potenciales entre pares de objetivos, siempre que tengan una estructura conocida (es decir, presentes en el PDB ).
Los métodos bayesianos [19] integran datos de una amplia variedad de fuentes, incluidos tanto resultados experimentales como predicciones computacionales previas, y utilizan estas características para evaluar la probabilidad de que una interacción proteica potencial particular sea un resultado positivo verdadero. Estos métodos son útiles porque los procedimientos experimentales, en particular los experimentos de dos híbridos en levadura, son extremadamente ruidosos y producen muchos falsos positivos, mientras que los métodos computacionales mencionados anteriormente solo pueden proporcionar evidencia circunstancial de que un par particular de proteínas podría interactuar. [20]
El análisis de exclusión de pares de dominios [21] detecta interacciones específicas de dominios que son difíciles de detectar utilizando métodos bayesianos. Los métodos bayesianos son buenos para detectar interacciones promiscuas no específicas y no muy buenos para detectar interacciones específicas raras. El método de análisis de exclusión de pares de dominios calcula un puntaje E que mide si dos dominios interactúan. Se calcula como log(probabilidad de que las dos proteínas interactúen dado que los dominios interactúan/probabilidad de que las dos proteínas interactúen dado que los dominios no interactúan). Las probabilidades requeridas en la fórmula se calculan utilizando un procedimiento de maximización de expectativas, que es un método para estimar parámetros en modelos estadísticos. Los puntajes E altos indican que es probable que los dos dominios interactúen, mientras que los puntajes bajos indican que es más probable que otros dominios que forman el par de proteínas sean responsables de la interacción. El inconveniente de este método es que no tiene en cuenta los falsos positivos y falsos negativos en los datos experimentales.
El problema de la predicción de PPI puede enmarcarse como un problema de aprendizaje supervisado. En este paradigma, las interacciones proteínicas conocidas supervisan la estimación de una función que puede predecir si existe o no una interacción entre dos proteínas a partir de datos sobre las proteínas (por ejemplo, niveles de expresión de cada gen en diferentes condiciones experimentales, información de ubicación, perfil filogenético, etc.).
El campo de la predicción de interacciones proteína-proteína está estrechamente relacionado con el campo del acoplamiento proteína-proteína , que intenta utilizar consideraciones geométricas y estéricas para encajar dos proteínas de estructura conocida en un complejo unido. Este es un modo de investigación útil en los casos en los que ambas proteínas del par tienen estructuras conocidas y se sabe (o al menos se sospecha firmemente) que interactúan, pero como muchas proteínas no tienen estructuras determinadas experimentalmente, los métodos de predicción de interacciones basados en secuencias son especialmente útiles junto con estudios experimentales del interactoma de un organismo .